畳み込みをセンサーデータ(1次元ベクトル)でやってみた
畳み込みニューラルネットワークは、画像処理で使われるのが一般的ですが、今回は、センサーデータなどにみられるような1次元ベクトルでやってみました。ポイントは、reshapeを使ったデータ構造の変換で疑似的に画像と同じ3次元(RGB,X,Y)にしています。畳み込みは、学習パラメータが少なくても特徴抽出ができ、異常検知に有効ではないかと思っています。
1.サンプルデータを作成
numpyを使って1周期分のSin波を作成しますが、
np.random.rand()で疑似ノイズをいれて
ある程度ばらつきを持ったものを100個作成します。
99個を学習用、残りの1個を検証用波形として利用しました。
data=[]
for i in range(100):
data.append([np.sin(np.pi * n /50)*(1+np.random.rand())for n in range(100)])
2.CNNのモデルを作成
ChainerのConvolution2Dを使って学習モデルを作成します。
畳み込んだデータを最後のレイヤーで元の入力データに復元させる構造となっています。
これにより、AutoEncoderとして特徴抽出を行います。
活性化関数をTanhにしたのは、ReLUでやるとConvlutionの途中データを
可視化するときにマイナス側がなく見栄えが悪いとおもったので。
(追記)可視化するときのデータは、活性化関数を通さないで取り出すようにしました。こちらの方が、正しいように思いましたので。
ReLUでやっても、Sin波のデータでは学習結果に影響はしていませんでした。
class MyChain(chainer.Chain):
def __init__(self,n_out):
super(MyChain, self).__init__()
with self.init_scope():
self.l1 = L.Convolution2D(None,2, ksize=(1,4),stride=(1,1))
self.l2 = L.Convolution2D(None,2, ksize=(1,4),stride=(1,1))
self.l3 = L.Convolution2D(None,2, ksize=(1,4),stride=(1,1))
self.l4 = L.Linear(None, n_out)
def __Call__(self,x,y):
return F.mean_squared_error(self.fwd(x),y)
def fwd(self, x):
h1 = F.tanh(F.max_pooling_2d(self.l1(x),2))
h2 = F.tanh(F.max_pooling_2d(self.l2(h1),2))
h3 = F.tanh(F.max_pooling_2d(self.l3(h2),2))
h3 = h3.reshape(h3.shape[0],-1)
return self.l4(h3)
3.学習させる
CNNを使ってやる場合、1次元のベクトルデータでは読み込めません。
そこで、学習データをReshapeを使って変換します。
また、教師データとなる方は元のベクトルデータとします。
TrainData = np.array(data,dtype=np.float32).reshape(100,1,1,100)
x=chainer.Variable(TrainData[:99])
for epoch in range(201):
model.zerograds()
loss=model(x,x.reshape(99,100))
loss.backward()
optimizer.update()
4.結果
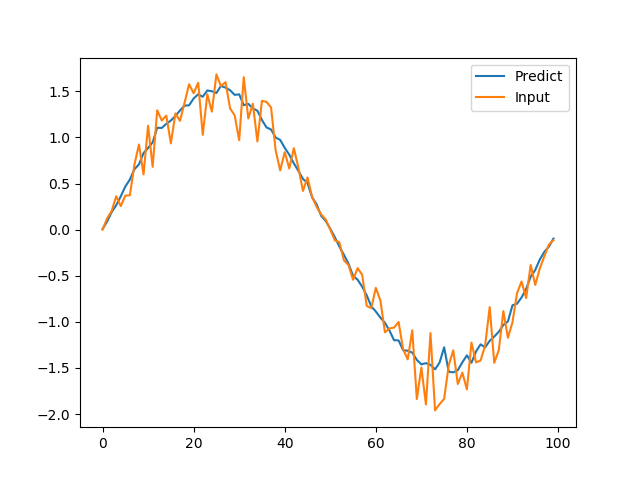
まずは、入力波形からの復元した結果。
普通に、復元できています。
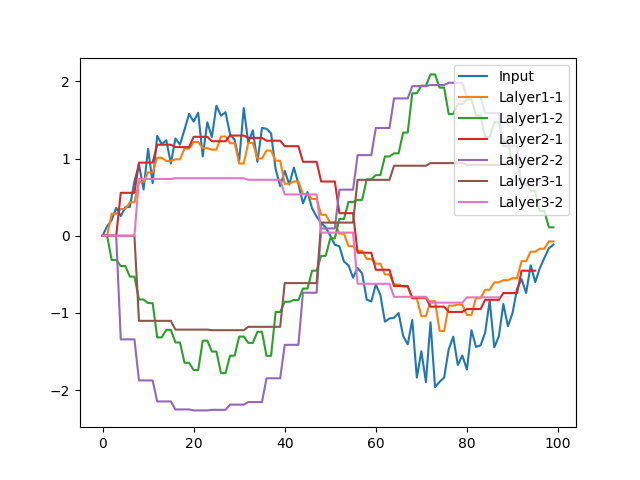
ここで参考までに、畳み込み過程の波形を見てみます。
データのサイズを疑似的に合わせてみています。
うーん、正直あまりよくわかりません。
Layer3が特徴を絞り込んだ波形といえるのですが、
Sin波自体の特徴はこんなものなのでしょうか。
山なりの形をとらえているのでしょうか。
また何回か学習をやってみましたが、毎回違う波形です。
それはそれで面白いと思います。
5.異常検知への拡張
過去にAutoEncoderをつかった異常検知を見たことがありますが、
入力と復元した出力との差を使って異常度を表す手法です。
同様に、異常データを作って検証してみました。
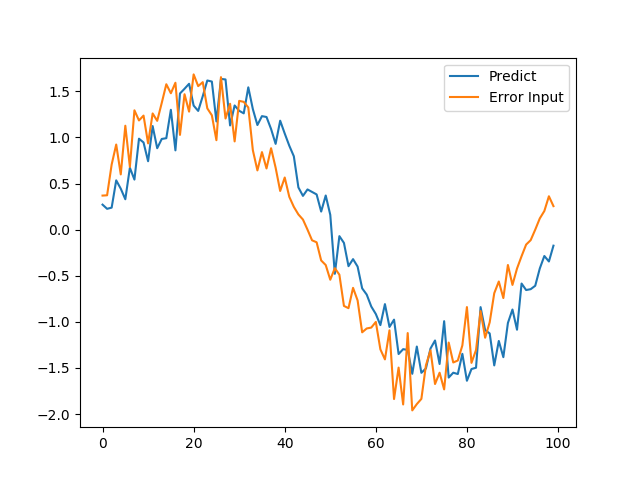
1つめは、位相ずれ
入力値を5打点(5/100周期)シフトさせてみました。
Predictの波形は、元の位相に近い結果となり、線の形もギザギザしています。
これは簡単に異常と検知できそうです。
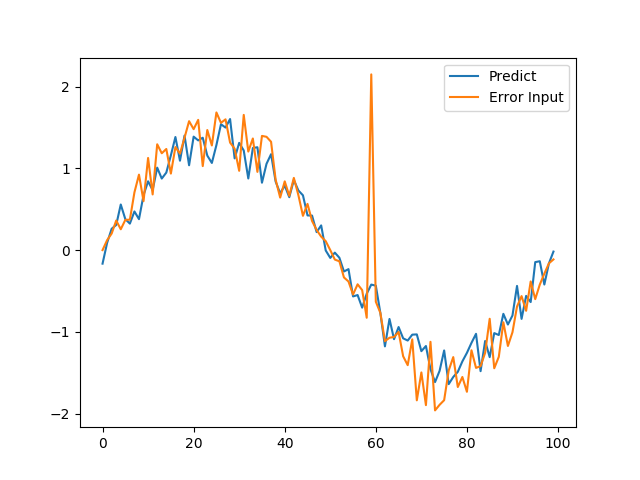
次に、スパイクのような1点が突出したような場合
こちらも、ギザギサしていますね。
このようにギザギザしてくれたら、差分などの特徴量から異常検知に使えるかもしれませんね。
6.おわりに
今回、初投稿させていただきました。
このサイトを通して、みなさまの投稿を拝見し、勉強させてもらっています。
その恩返しと思い、自分も投稿しようと思いました。
この投稿が、みなさまの何かの参考になれば幸いです。
(追記)使用した実際のサンプルコード
環境 Python 3.6.1
Anaconda 4.4.0 (64-bit)
Chainer 2.0.2
import chainer
import chainer.functions as F
import chainer.links as L
import chainer.optimizers
import numpy as np
import matplotlib.pyplot as plt
class MyChain(chainer.Chain):
def __init__(self,n_out):
super(MyChain, self).__init__()
with self.init_scope():
self.l1 = L.Convolution2D(None,2, ksize=(1,4),stride=(1,1))
self.l2 = L.Convolution2D(None,2, ksize=(1,4),stride=(1,1))
self.l3 = L.Convolution2D(None,2, ksize=(1,4),stride=(1,1))
self.l4 = L.Linear(None, n_out)
def __call__(self,x,y):
return F.mean_squared_error(self.fwd(x),y)
def fwd(self, x):
h1 = F.tanh(F.max_pooling_2d(self.l1(x),2))
h2 = F.tanh(F.max_pooling_2d(self.l2(h1),2))
h3 = F.tanh(F.max_pooling_2d(self.l3(h2),2))
h3 = h3.reshape(h3.shape[0],-1)
return self.l4(h3)
def Layaer1(self, x):
return F.max_pooling_2d(self.l1(x),2)
def Layaer2(self, x):
h1=F.tanh(F.max_pooling_2d(self.l1(x),2))
return F.max_pooling_2d(self.l2(h1),2)
def Layaer3(self, x):
h1 = F.tanh(F.max_pooling_2d(self.l1(x),2))
h2 = F.tanh(F.max_pooling_2d(self.l2(h1),2))
return F.max_pooling_2d(self.l3(h2),2)
def CreatePlotData(arr,n1):
Buf1,Buf2=[],[]
for j in range(n1):
Buf1.append(0)
Buf2.append(0)
for i in range(arr.shape[1]):
for j in range(n1):
Buf1.append(arr[0][i].real)
Buf2.append(arr[1][i].real)
return np.array(Buf1,dtype=np.float32),np.array(Buf2,dtype=np.float32)
data=[]
for i in range(100):
data.append([np.sin(np.pi * n /50)*(1+np.random.rand())for n in range(100)])
model = MyChain(100)
optimizer = chainer.optimizers.Adam()
optimizer.setup(model)
TrainData = np.array(data,dtype=np.float32).reshape(100,1,1,100)
x=chainer.Variable(TrainData[:99])
ValidationData=TrainData[99].reshape(1,1,1,100)
PlotInput = ValidationData.reshape(100)
for epoch in range(201):
model.zerograds()
loss=model(x,x.reshape(99,100))
loss.backward()
optimizer.update()
if epoch%20==0:
Layer1Arr = np.array(model.Layaer1(ValidationData).data).reshape(2,-1)
Layer1Arr1,Layer1Arr2 = CreatePlotData(Layer1Arr,2)
Layer2Arr = np.array(model.Layaer2(ValidationData).data).reshape(2,-1)
Layer2Arr1,Layer2Arr2 = CreatePlotData(Layer2Arr,4)
Layer3Arr = np.array(model.Layaer3(ValidationData).data).reshape(2,-1)
Layer3Arr1,Layer3Arr2 = CreatePlotData(Layer3Arr,8)
plt.plot(PlotInput,label='Input')
plt.plot(Layer1Arr1,label='Lalyer1-1')
plt.plot(Layer1Arr2,label='Lalyer1-2')
plt.plot(Layer2Arr1,label='Lalyer2-1')
plt.plot(Layer2Arr2,label='Lalyer2-2')
plt.plot(Layer3Arr1,label='Lalyer3-1')
plt.plot(Layer3Arr2,label='Lalyer3-2')
plt.legend()
plt.savefig(str(epoch)+'-Epoch Convolution Graph.png')
plt.close()
predict = model.fwd(ValidationData)
predict=np.array(predict.data).reshape(100)
plt.plot(predict,label='Predict')
plt.plot(PlotInput,label='Input')
plt.legend()
plt.savefig(str(epoch)+'-Epoch Validation Graph.png')
plt.close()
ErrorPlot = [PlotInput[i+5]for i in range(len(PlotInput)-5)]
for i in range(5):
ErrorPlot.append(PlotInput[i])
predict = model.fwd(chainer.Variable(np.array(ErrorPlot,dtype=np.float32)).reshape(1,1,1,100))
predict=np.array(predict.data).reshape(100)
plt.plot(predict,label='Predict')
plt.plot(ErrorPlot,label='Error Input')
plt.legend()
plt.savefig('Shift Error Input Graph.png')
plt.close()
Rnd = np.random.randint(0,99)
ErrorPlot2=np.array(PlotInput)
ErrorPlot2[Rnd]=ErrorPlot2[Rnd]+3
predict = model.fwd(chainer.Variable(np.array(ErrorPlot2,dtype=np.float32)).reshape(1,1,1,100))
predict=np.array(predict.data).reshape(100)
plt.plot(predict,label='Predict')
plt.plot(ErrorPlot2,label='Error Input')
plt.legend()
plt.savefig('Spike Error Input Graph.png')
plt.close()