はじめに
*この記事はコロナウィルスに対する政府や自治体の対応や、特定のプロパガンダを流すための記事ではありません。
私は東京都に住んでいるのですが、連日コロナの感染者数が増えていると報道がなされています。最近では連日200人超えとの報道で若い人が大半を占めているとの報道です。
ただ、観測範囲で若い人ばかりコロナにかかったとは聞かず、一応データサイエンティストの端くれとして、これは開示されたデータを元にちゃんと調べるべきだと思った次第です。
まぁ、pandasの練習問題としても適切かなぁと。。今回は年代ごとにどのようにコロナの新規感染者数が推移しているか調べることにします。
データソース
データはこちらから落とせます。
https://catalog.data.metro.tokyo.lg.jp/dataset/t000010d0000000068/resource/c2d997db-1450-43fa-8037-ebb11ec28d4c
csv形式になっていて、何も入っていないカラムが多いものの、データも綺麗で扱いやすいかなぁと思います。これを書いている時点で7/9までのデータがあるようです。
環境
今回はDockerで立てたJupyter上で分析環境を作りました。
だいぶ適当ですが、他で使ったものをただ使い回しているだけなのでご了承を(こんなにrequireしないw)。
FROM python:3.8.2
USER root
EXPOSE 9999
ENV PYTHONDONTWRITEBYTECODE 1
ENV PYTHONUNBUFFERED 1
RUN mkdir /code
WORKDIR /code
RUN apt-get update && apt-get -y install locales default-mysql-client && \
localedef -f UTF-8 -i ja_JP ja_JP.UTF-8
ENV LANG ja_JP.UTF-8
ENV LANGUAGE ja_JP:ja
ENV LC_ALL ja_JP.UTF-8
ENV TZ JST-9
ENV TERM xterm
ADD ./requirements_python.txt /code
RUN pip install --upgrade pip
RUN pip install -r /code/requirements_python.txt
WORKDIR /root
RUN jupyter notebook --generate-config
RUN echo c.NotebookApp.port = 9999 >> ~/.jupyter/jupyter_notebook_config.py
RUN echo c.NotebookApp.token = \'jupyter\' >> ~/.jupyter/jupyter_notebook_config.py
CMD jupyter lab --no-browser --ip=0.0.0.0 --allow-root
glob2
json5
jupyterlab
numpy
pandas
pyOpenSSL
scikit-learn
scipy
setuptools
tqdm
urllib3
matplotlib
xlrd
実装
ここからは実装について記載していきます。
1. importと読み込み
まずは必要なパッケージとファイルを読み込みます。今回はmatplotlibとpandasしか使わないです。
公表年月日についてはこのタイミングでdatetime型に変更しておきます。
import pandas as pd
import matplotlib.pyplot as plt
import matplotlib as mpl
%matplotlib inline
df_patient = pd.read_csv('./data/130001_tokyo_covid19_patients.csv')
df_patient['公表_年月日'] = pd.to_datetime(df_patient['公表_年月日'])
df_patient.head()
2. データの取り出しと加工
今回のデータ加工ですが以下の要領で行います。
- 年代ごとに新規感染者数をカウント
- 曜日ごとの検査数などの要因を排除するために7日間の移動平均でカウントする。
2-1 年代ごとに新規感染者数をカウントする
これはgroupbyを用いればそんなに難しくないです。またこのタイミングで必要なカラムだけにしておきます。
df_patient_day = df_patient.groupby(['公表_年月日','患者_年代']).count().reset_index()[['公表_年月日','患者_年代','No']]
df_patient_day
また、年代の記載に日本語があるとmatplotlibの箇所で文字化けする(上記の通り環境使い回しで対応するのが面倒)ので、以下の通り置き換えてしまいます。
genes_dict = {'10歳未満':'under 10',\
'10代': '10', \
'20代': '20', \
'30代': '30', \
'40代': '40', \
'50代': '50', \
'60代': '60', \
'70代': '70', \
'80代': '80', \
'90代': '90', \
'100歳以上': 'over 100', \
"'-": '-',
'不明': 'unknown'
}
df_patient_day['患者_年代'] = [genes_dict[x] for x in df_patient_day['患者_年代'].values.tolist()]
df_patient_day
上記の場合問題があって、新規感染者数がその日、その年代に存在しない場合データがなく、後で移動平均を取るときに問題になってしまうので、ここでこのデータがある範囲で上記年代×日付の範囲全体の直積を作ってそれと上記DataFrameを結合します(ここはもうちょっといいやり方知っている方いたら教えてください!)。
genes = ['under 10',\
'10', \
'20', \
'30', \
'40', \
'50', \
'60', \
'70', \
'80', \
'90', \
'over 100', \
'-',
'unknown'
]
days = pd.date_range(start=df_patient['公表_年月日'].min(), end=df_patient['公表_年月日'].max(), freq='D')
data = [[x, y] for x in days for y in genes]
df_data = pd.DataFrame(data, columns=['公表_年月日', '患者_年代'])
df_data = pd.merge(df_data, df_patient_day, on=['公表_年月日', '患者_年代'], how='left').fillna(0)
df_data = df_data.rename(columns={'No':'人数'})
df_data
2-2 年代ごとに移動平均をとる
年代ごとに移動平均をとります。pandasの機能で簡単に移動平均は取れます。7日間移動平均の場合はrolling(7)とやるだけです。平均を取りたい場合はrolling(7).mean()とやります。そして、最初の6日間がnanとなるので、dropna()で消します。今回は後の実装のために、年代ごとにDataFrameにして、辞書に格納することにします。これで完成です!
result_diff = {}
for x in genes:
df = df_data[df_data['患者_年代'] == x]
df = pd.Series(df['人数'].values.tolist(), index=df['公表_年月日'].values)
result_diff[x] = df.rolling(7).mean().dropna()
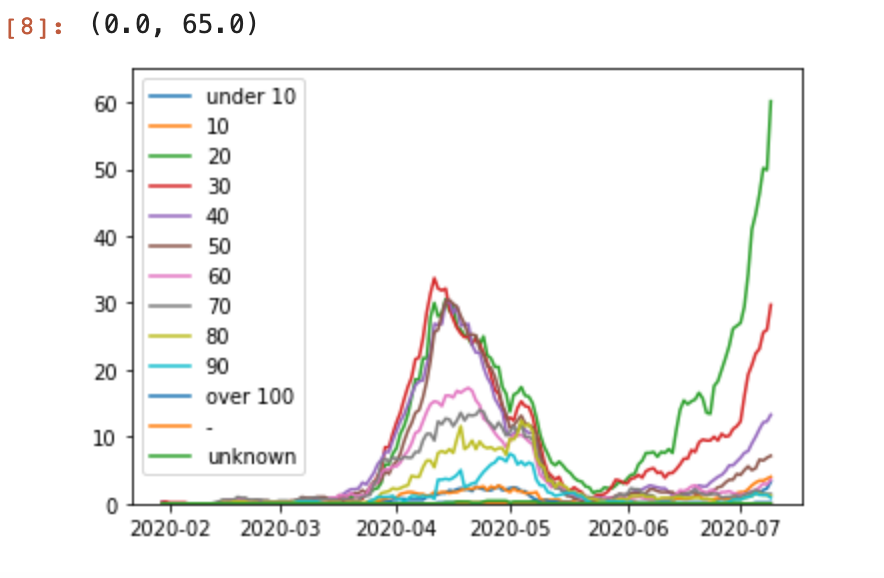
3. 可視化する
最後に可視化します。
fig, axe = plt.subplots()
for x in genes:
df_diff = result_diff[x]
axe.plot(df_diff.index, df_diff.values, label=x)
axe.legend()
axe.set_ylim([0,65])
結果
20代から50代までは大体年代が若い順にここ最近は推移していることが確認できました。それにしても20代の増え方が驚異的ですね。東京都の発表は嘘でなかったですね。
まとめ
ここでは、要因がどうとかいうつもりはないのですが、このように公開データでさくっと報道の内容を調べることもできますので、練習も兼ねてやってみてはどうでしょうか。これも実際の人口分布と照らし合わせてどうとかまだまだ、調べられることもたくさんありますし、実際にデータ加工の練習としてもいい教材だと思います。