(1)まず、CT画像で3D表示をやってみる
3Dslicer (https://www.slicer.org/)は、画像を3D 表示することができる無料ソフトである(今では多くの病院でCTからの3D表示を実際おこなっている。もちろんそれは有料ソフトをつかったもの)。
素材は、ここ20年ずっと、大きな病院から病診連携のさい提供されている、CD-ROM(AOC診断画像ディスク)からのCT画像である。
今回、ぼくにとって、使う経験ははじめてだった。
まず、WEBで紹介されているやり方にならってみたが、うまく表示できず、結局ChatGPTに聞きながら、ずいぶん違った手続きが必要だったので、ここに簡単に紹介する。
ネットより、3D slicerダウンロード後
① CD内のDICOMフォルダ内の画像ファイル(例 785個)をPCデスクトップに保存。しかし、その画像ファイルには、拡張子、.dcmがついていない!
⇒785個の画像ファイルすべての名前の末尾に、拡張子、.dcmをつける
(方法)C:\dicom_workを作成し、その中に785個のファイルをいれ、さらに.batファイルを作成
*作成した .bat ファイルは、同じ C:\dicom_work フォルダの中に置いてOK
.bat の中身(このままコピペOK)
@echo off
cd /d "C:\dicom_work"
for %%f in (*) do (
ren "%%f" "%%f.dcm"
)
pause
(参考) 内容の意味:
cd /d:対象フォルダへ移動
for %%f in (*):全ファイルを対象
ren "%%f" "%%f.dcm":ファイル名の末尾に .dcm を追加
pause:結果を確認できるように一時停止
その後、
rename_dicom.bat などの名前で保存(拡張子 .bat)
右クリック → 管理者として実行
実行後の確認
フォルダ内のファイルがすべて 00000001.dcm のようになる
*最初、デスクトップでつくっていたら、やっているうちにデスクトップ上のすべての書類名に.dcmがついてしまい、手動で名前をもとにもどさねばならなかったので、注意が必要
② このフォルダを 3D Slicer で Import DICOM files → 読み込み(load) ⇒ これだけではだめだった。読み込んだファイルから、3D表示を邪魔しているファイルを一部手動で削除する必要があった(左側に表示)
不要(どころか表示を邪魔する)Warning例
Scout 撮影前の位置合わせ画像(1枚) ❌ 不要
Dose Report 放射線量のテキスト画像 ❌ 不要
Lung 5mm / 4th 5mmなど 実際のCTスライス ✅ 読み込むべき
そして、ようやく本来の手順へ
③左上のモジュ-ル、クリック、プルダウンから「Volime Rendering」選択。
左側のDisplayのSelect a presetから適当なものを選んで、それから、左上方の「Volume」表示左の眼をクリック(そうすると、目が開き、3D表示が右上にあらわれる)
*表示の調整方法
マウス操作:
左ドラッグ:回転
右ドラッグ:ズーム
ホイール:スライス調整
「Shift + 左ドラッグ」でより柔らかく動かせる
④「胆のうの抽出と表示」
Segment Editor モジュールに入る
上部「Modules」から Segment Editor を選択
Add ボタンで新しいセグメントを作成(名前例:gallbladder)
Threshold(CT値で胆のう領域を抽出)
胆のうは中に胆汁があり、CT値が低く(-20〜+30 HUくらい)、周囲とコントラストがあるため、それを利用。
手順
Threshold ツールを選択
スライダーで範囲を指定:
最小値:-50〜0 最大値:+50〜100
※この範囲内だけがセグメントに塗られる
抽出範囲が緑に表示される → 良ければ Apply をクリック→ Show 3D をクリックして立体表示確認
Scissors(3Dの空間範囲でくりぬく)
3D表示されたVolumeから、空間的に「胆のうの位置だけ」残すように切り抜く
手順
Scissors ツールを選択
Operation を「Erase outside」に設定
下記の「効果範囲」を 3D に変更
3Dビューで、ドラッグして胆のうのある範囲を選択(立体切り抜き)
Apply をクリック
*左の「Segmentations」で Show 3D を押すと、切り出された立体だけが表示される
*Segment Editorで Smoothing(滑らか化)を適用すると綺麗な形になる
実際のところ、CTでは、Scissors処理が胆のう近くに多く、大変だった
(2)次にエコー画像で
200枚の超音波スライス画像(.jpg)を3D Slicerに読み込んで、Volume(体積データ)として扱うには、以下のような手順が必要。
-
画像を連番ファイルに揃える(すでに済)
slice_001.jpg
slice_002.jpg
...
slice_200.jpg -
Slicerに読み込む(Image Stackとして)
Slicer起動
メニュー:Add Data (Add Dicom dataでない!)または Ctrl + A
ダイアログで .jpg をすべて選択
✔️「Show Options」ボタンをクリック(表示されない場合は下にスクロール)
「Single file」ではなく「Image Stack」にする(重要)
OKを押すと 3D Volume として読み込まれる
*「JPEG画像は階調が荒く、モノクロ濃淡が不正確な場合があり、PNG や TIFF に変換してから読み込むとより適切です」との注意もあったが、今回JPEGでもかなりうまくいくことがわかった
CTの時と同様、Segment Editor モジュールにはいり、Threshold(CT値で胆のう領域を抽出)を、胆のう部分だけが緑になるように(実際はピンポイントはむずかしいが、そのような傾向になるように)値を設定し、3D表示した。
矢印の先がどうやら胆のうである。
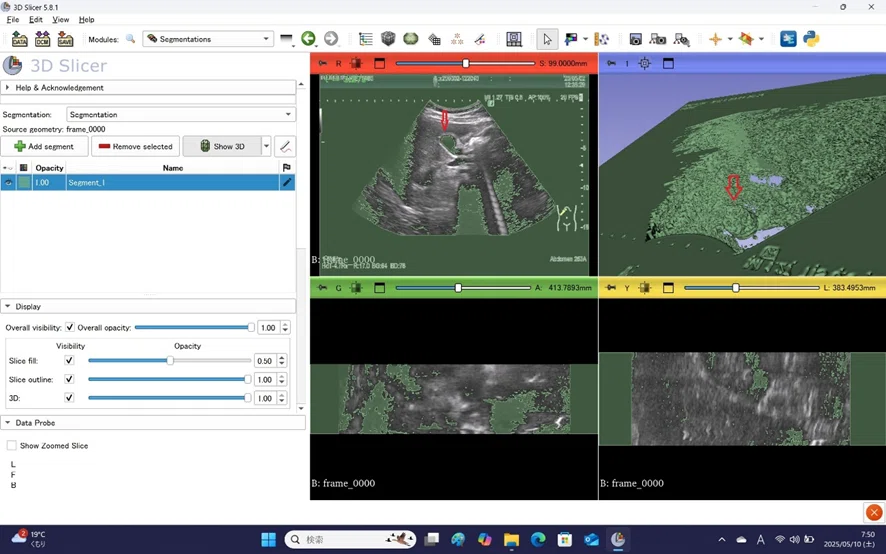
拡大すると、うまくいっている
CTの時、Scissors処理が胆のう近くに多く大変だった。だが、エコーの時は、胆のうはその周囲から最初から分離していた

Scissors(矢印)とは:胆のう以外の表示を囲って順にけすだけのこと
このSlicerでは、自分のオリオジナルでは「jpgをThreshold後、白黒2極のpngに変換」という作業を、一気におこなえた。

(参考)エコー画像でのpngをとりこんで、直接3D表示をいてみた
もう、Thresholdしてあるので、(slicerでthresholdせず)直接3D表示でも、下記のように、それなりの画像ができた。

(3)まとめ
自己作成のオリジナルアプリ群での3D表示を、Slicerと比較すると。
slicer: けっこうきれい。辺縁もスムース。
今回は、ThresholdとScissorsしかつかってないが、paint,drawなどほか のツールが充実
また、わざわざpng化せずともjpgのままThreshold→白黒2極化、を自動でしてくれる。
とはいえ、Slicerをもってしても、なかなかスムーズにはいかない。また、胆管までの3D表示はむずかしそう。
結局、slicerをつかったとしても、現状では、3Dで「見やすく患者に提示できる」ようにはなるが、新たな診断知見を得ることはむずかしいそう。
だが胆のうの周囲との分離は、CT画像よりエコー画像のほうがしやすいことがわかった(CTでは、Scissors処理が胆のう近くに多く大変だった)。
*まあ、これは、普段CTやエコーをみていて納得できる観察であるが。
(4)今後の予定
その3 以降では
①応用範囲を 盲腸~虫垂 /S状結腸~直腸 に広げる
②3D表示にこだわらず、スクロール+ピクセル表示も含め、診断にいかせるところがないか探っていく
③3D表示については:虫垂の有無、S状結腸の蛇行がアルファか逆アルファか?の判別に役立つことを期待

