特に今回の目標は、エコーによる消化管粘膜病変の検出を、少しでも向上させることにある。
消化管は、エコーでもずいぶんみえるようになった。だが、主な問題として(1)一瞬にしかみえない(2)臓器の全体像がわからないので、また、プローブの入射角や向きは自由な操作のため様々なので、どんな形の物体のどのような断面図をみているのかわからない
今回の3D画像作成への挑戦は、上記の2つの問題を解決する一助となりうる。なぜなら、この作業によって、(1)病変は一瞬から固定化した画像になり(2)3D化作業により、全体の臓器の中のROIの位置がより明確にイメージできる。
別の言い方をしてみよう。CTは撮影中には病変がわからず、画像ができてからその読影で病変を知る。一方、エコーは走査中に病変がわかる。エコー画像を3D画像にするのは、その情報を再現し、臓器の中の位置を予想しやすくするためであるのだ。 そのためのステップをここで一度まとめてみたい。
Step1 各臓器をみていき、一瞬病変らしきものがみえたとき、それを平面画像だけでなく、動画にしてとる。
Step2 動画からスライス画像を作成、それを再構築することで、3D画像を作成する
Step3 Step2で作成した画像を目視でチエック。スライスの観察は、細かい粘膜病変はわからないにせよ、少なくとも粗大病変(としても一瞬しかうつらない)を再確認できるだろう。さらに、3D画像をつくり、その病変の臓器内の位置を示すための準備(ピクセル値の閾値を利用し白黒画像に変換する「マスク法」)もここで行う。
Step4 Step3でチエックした場所をふくめ、3D画像上に表示。すなわち、まず、どのスライスか?次に、そのスライスの、その場所か?3D画像上に表示する。
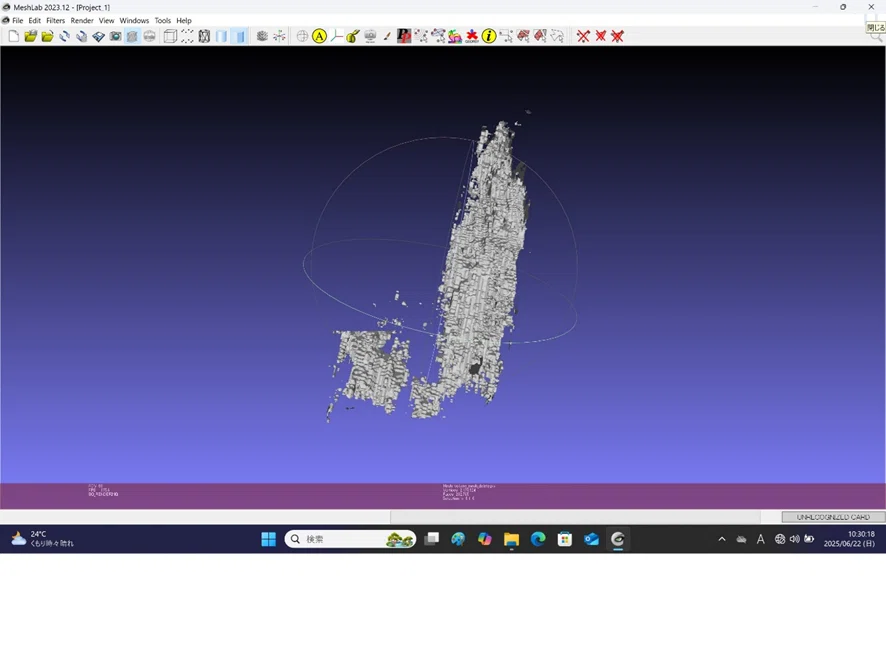
*作成された3Dは、MeshLabでノイズを削除し見やすくする、ということも適宜行う。
それぞれのステップでさらなる改良が必要である。
Step1 Ste2 プローブの入射角や向きは自由な操作のため様々である。だが、この3D画像構築のため動画撮影をとる場合、プローブの操作方法に制限をもうける。それにより正確な3D画像再構成を実現するためである。
① 水平移動はとりあえず、プローブに対し90度の方向に動かすこと。(例えば、プローブに対し45度の角度に固定して動かすということも、角度を動かさねば、悪いわけでないが)
このように動画を撮影した後、(今まで、その1~3で紹介してきたように)まず動画(mp4)を適当なスライスに分割
mp4_to_jpg_with_time.py(その1)
場合によっては、移った箇所のピクセル値を計測して域値設定の情報にする
pixel_point.py (その1)
設定した域値で、jpg画像を白黒のpng画像に変換 (その1)
generate_threshold_masks.py あるいは、
generate_threshold_masks_maxmini.py
3D表示(その1)
main_pyvista_3d.py
場合によっては、エコーで取得した画像全体でなく、その中の一部の範囲を3Dにする方が、よりよく見えることがあるかもしれない。そのために、arc_masks_trimmed.pyで画像上にポリゴンで狭い範囲を選択する(その5)。
② あるいは、プローブを固定して、前後に倒すこと。このとき、「移動角度」は30度と仮定(その5)。
*今回簡易法センサーとして、M5StickC plusをエコープローブにつけて角速度モニタリングをおこなってみた(位置モニタリングはできない)が、(あまりにリアルに近すぎて?)今のところ、大きな優位性が認められないため積極的な導入は見送った。
*将来、プローブそのものにセンサーを内蔵し、ロボット手術などに用いられる、なんらかのトラッキング技術を導入することで、自由なプローブの動きを正確にモニターすることも考えられなくもない。例えば、部屋の4隅、あるいはエコー装置そのものや検査用ベッドにモニターをつけて、センサーの情報をキャッチするのだ。
Step3 ここで、「基本となる目視」に加え、Cannny法は、チエックをより正確にするのは役に立つかもしれない。また症例をつみかさねれば、チエックを学習したAIにまかせられるようになるかもしれない。
Canny法をまじえ、特徴のあるスライスをメモしておく
CannyViewer_FINAL_FIXED.py (その3)
ターゲットとなる病変部位が3Dの中でどの辺にあるか?3D空間の中でどのスライスか?については、
show_slice_in_volume_1.py (その3)
(または、3DSliceViewer_2.py)
さらにそのスライスから場所病変候補を選択して3D表示
click_view_center.py (その4)
view_polyp_2.py (その4)
も有用である。
*ここで観察された、胆のう外からの視点、だけでない「胆のう内からの視点」は、将来、胃や腸を疑似内視鏡観察をしたような「大腸CT」の手法への第1歩である。解像度等の限界もあり、まだ大腸CTのような、内視鏡をおこなっているようなな変化していく内部画像を得るまでには、まだ時間が要しそうであるが、この大腸CTのような映像は目標のひとつである
そして、3D画像をつくり、その病変の臓器内の位置を示すための準備(ピクセル値の閾値を利用し白黒画像に変換する「マスク法」)の中心は、その閾値の決定にある。基本は、以前に書いたような、移った箇所のピクセル値を計測して域値設定の情報にする
pixel_point.py (その1)
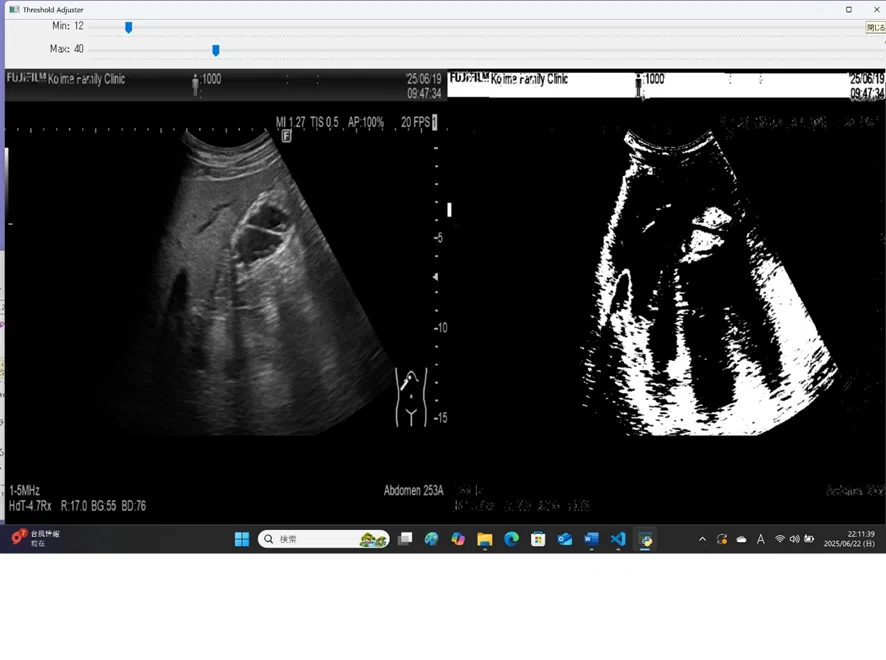
さらに、これをもっと直感的にするように、たとえば、OpenCVのスライダーUIでしきい値を調整しながら、1枚の画像を2値化して確認できるPythonコード:try.py もいいかもしれない。
try.py(図では、左の元画像から、胃の部分が白く「マスク化」されるように、上のmax,minの閾値をスライダーで調整している)
このtry.pyをさらに発展させたプログラムをつくろうとしたとき、これは、tkinkerベースの手法では複雑すぎて間に合わず、Flaskの導入によって達成した。
達成したシステム:threshold_viewer_flask どの閾値(マスク)を使用するといいのか、対象jpgファイル(例200枚)すべてをながめながら決定(閾値は全ファイル共通、ひとつだけ) app.py実行後、localhost (http://127.0.0.1:5000) にて描画:
(注 この派生版、threshold_viewer_flask_2もある。そこでは、1枚ずつ最適な「マスク」を個別の画像につくり保存していく(閾値の種類はファイルの数だけある))
水を飲み、座位で、扇形操作で撮った動画をもとに作成したもの。 十二指腸球部と胃のようなものがみてとれる。 だが、特に、胃上部の胃とその周辺組織との境界があいまいである。 いずれにせよ、目視可能な粗大粘膜病変はなかった(自分自身の胃)。
終わりに
各臓器別のアプローチ。
① 胃を、扇形アプローチでとる(M5StickC plusなし。角度は30度と仮定)]
② 回盲部を、平行移動アプローチでとる
③S状結腸を、平行移動アプローチで?直腸を、扇形アプローチで?
*消化管は5か所、体内で固定されている