Atomeyeとは
分子動力学(Molecular Dynamics, MD)シミュレーションを行った際の出力として、しばしば、原子の配置をそのまま可視化したいということがあります。
Atomeyeはそのような可視化を行うためのツールです。
Website: AtomEye: atomistic configuration viewer
Paper: "AtomEye: an efficient atomistic configuration viewer", J. Li, Modelling Simul. Mater. Sci. Eng. 11 173–177 (2003).
この記事では、Atomeyeのビルド時の引っかかりポイントを解説し、記事を読んだ人が自分の環境でもビルドできるようにすることを目的としています。
Atomeyeはcfgファイルという形式の構造データ(原子の位置と各原子に紐づけたオプションパラメータから構成される)を読みます。LAMMPSからは直接dumpコマンドでcfg形式のファイルを出力することが可能です。この際はdump_modifyコマンドで元素も指定しておきましょう。
この記事ではAtomeyeの操作については詳しく紹介していません。詳しく知りたい方は公式ページのマニュアルが参考になります。よく使うキーの簡単な手引きとしては、Delete/Insertキーで連番ファイルの移動、bキーでボンドの表示切り替え、k/lキーで配位数・原子種の表示切り替え、Alt+数字キーで出力時に原子にふったパラメータの表示、原子やボンドのダブルクリックで詳細表示、ドラッグで回転、Shift+ドラッグで原子の平行移動、Ctrl+ドラッグで格子の平行移動、Home/End/PageUp/PageDownキーで原子やボンドの大きさ切り替えなどです。たいていの場合ではボンドを表示して原子をかなり小さくしたほうが見通しが良いと思います。
公式ページでは、オリジナルバージョンの他に、並列計算を目的として改造されたAtomeye version 3というものが公開されています。並列計算以外にも設定の保存と読み込みやキーの変更などいくつかの機能が追加されているので、この記事ではこちらのv3を中心に書いていきます。以下の説明のA3ディレクトリをAディレクトリに読み替えればオリジナルバージョンもビルド可能です。
MDの可視化とAtomeyeの使い道
MDは3次元空間で粒子を動かすタイプの物理シミュレーションです。とはいえ、実際に見たいものが個別の原子の位置そのものではないことも多くあります。例えば空孔、不純物、転位や結晶粒界などの格子欠陥を見たい場合、熱や応力分布など位置以外の情報を見たい場合、相を知りたい場合、ネットワーク構造の解析など様々な場合が考えられ、問題ごとに特有であることも多いです。ポスト処理を自分で書くこともあるでしょう。このためのツールとしては例えばOVITO、Paraviewなど高機能なソフトがいくつか考えられます。MDを計算サーバーで行う状況を考えると、これらは基本的にポスト処理を行ったファイルを手元の環境に持ってきて可視化ツールを動かすようなイメージでしょうか。
しかし一方で、生データを見るかのごとくサクッと可視化したいという要求もあります。例えばMDの初期にきちんと所望の配置になっているか、何かの手違いで系が崩壊や発散したりしていないか、反応がどこまで進んだか、などの確認を行うのに毎回ポスト処理をかけてファイルを転送し、可視化ソフトで読み込んで…という処理をかけるのは結構な手間です。計算サーバー側で、理想をいえばログファイルに対するlessコマンドくらい手軽に可視化できると助かります。
このため、個人的には手元でしっかり可視化するツールとサーバーで動く確認用の軽いツールが別にあると便利だと思っています。Atomeyeはこの後者の要件に向いているソフトだと思います。
個人的にはデータのグラフ化でいうところのGnuplotとよく対応していると思います。
事前準備
はじめに依存するライブラリをインストールします。以下はapt形式で書いています。
$ sudo apt install gfortran xterm libopenmpi-dev liblapack-dev libblas-dev libreadline-dev libgsl-dev libx11-dev libxext-dev libxpm-dev libpng-dev libjpeg-dev
個別に自分でビルドすることもできなくはないですが、X11関連を0からインストールするのは少々骨が折れると思います。
一般にGUIを動かすときに共通の話ですが、もしSSH接続などリモート環境の場合には手元側でX Window Systemのサーバーとフォワーディングの設定をしておきましょう。WindowsならXmingなどのソフトが使えます。
gnuplotとかxeyesとか何らかのGUIツールを起動して画面に表示できていれば大丈夫です。
次はAtomeye (v3)自体をダウンロードしてきましょう。オリジナルのAtomeye本体とv3パッチの2種類のファイルからなっています。
内部のAディレクトリをA3ディレクトリにコピーしてパッチを当てるというスタイルをとっています。
$ wget http://li.mit.edu/A/Graphics/A3/Src/Src-20101019.tar.bz2
$ wget http://li.mit.edu/A/Graphics/A3/Patch/A_20101019-A3_20121115.patch
$ tar xf Src-20101019.tar.bz2
$ cd Src-20101019
$ cp -r A A3
$ cd A3
$ patch < ../../A_20101019-A3_20121115.patch
上記のように普通にWebページに置いてありますが、素朴にダウンロードするのが怖い人向けに、
(安心にはならないと思いますが、)参考までに手元で計算したmd5sumとsha256sumを載せておきます。
$ md5sum Src-20101019.tar.bz
7a55a63a1d1b97fb2193a04cab39a7f4 Src-20101019.tar.bz
$ sha256sum Src-20101019.tar.bz
79d72c4464ea282b0a1fd75fee746ede396d1060daa267e8fd9322cc88f5a78f Src-20101019.tar.bz
$ md5sum A_20101019-A3_20121115.patch
96d9c68660b5b54cd7d9ad459ef4e78f A_20101019-A3_20121115.patch
$ sha256sum A_20101019-A3_20121115.patch
da6babbabb00d43916965b68360fb5db9b14310605290aa55d831f9b222d7abc A_20101019-A3_20121115.patch
makeの前に、環境変数を通してインストールする場所の設定を行っておきます。
場所は任意ですが、指定したディレクトリをこの段階で作っておくことが必要です。デフォルトは${HOME}/Coです。
最終的には自分でビルドしたものはすべて静的リンクされるので、実行ファイル本体だけあれば大丈夫です。
その意味ではインストールする場所は/tmp以下でも構いません。環境変数の設定を.bashrcに書く必要もありません。
$ export SYS=Linux
$ export LIB_PATH=${HOME}/my_path/lib
$ export BIN_PATH=${HOME}/my_path/bin
$ export INC_PATH=${HOME}/my_path/include
$ export MAN_PATH=${HOME}/my_path/man
$ mkdir ~/my_path
$ mkdir ~/my_path/lib
$ mkdir ~/my_path/bin
$ mkdir ~/my_path/include
$ mkdir ~/my_path/man
シェルに慣れていない人は、exportで設定した環境変数は再起動などで一度シェルを抜けるとなかったことになるので注意してください。
ビルド
基本的には、readme.txtに書かれた順にディレクトリに入ってmakeを繰り返すことになります。(AのみA3に置き換え)
- 書き下すと以下の順になります。*マーク付きはこの記事内に追加のコメントがあるディレクトリです。
- Timer/
- IO/ *
- Scalar/
- VecMat/
- VecMat2/
- VecMat3/
- Min/ *
- Atoms/
- AX/ *
- A3/ *
ただ、そのままmakeしてもいろいろと問題が起きると思うので、先に以下の変更点を適用しておきましょう。
まず、A3を除くすべてのディレクトリについて、Makefile内の ifeq ($(SYS), Linux) 以降の行のコンパイラの指定がデフォルトでiccになっているのでgccに変更しておきましょう。具体的には
CC=gcc
LD=gcc -static
CCFLAGS=-O2 -mtune=native
としておけば大丈夫です。CCFLAGSのオプションはお好みで。手元の環境にIntel Compilerが入っているならiccのままで大丈夫です。
また、コンパイルを通すのに&バグを回避するのに必要な個別の変更点が結構あります。
見つけたものを以下に列挙しておきます。
-
Min ディレクトリ
-
Makefile- FC=ifcをFC=gfortranに
-
-
AX ディレクトリ
-
Makefile- LD=の行がなぜか2行あるので注意 (どちらか1行を消してしまってよい)
-
AX.c- 693行目
if (setjmp(png_ptr->jmpbuf))をif (setjmp(png_jmpbuf(png_ptr)))に (libpngの仕様に関連した変更)
- 693行目
-
AX.h- 52行目
#include <zlib.h>を新しく追加 - 64行目
#define AX_MAXSTRSIZEを256とかに (文字列処理の安全のため)
- 52行目
-
-
A3 ディレクトリ
-
Makefile- CC、LD、CCFLAGSはmpiccのまま変更しなくてもよい
- 132行目
/usr/lib/libm.aを-lmに変更、-lX11を追加
-
cui.c(v3のみ)- 1506行目
change_bond_radius( 1-c);をchange_bond_radius(1.0/(1-c));に変更 (ボンドを細くするコマンドを使用すると太くなるバグの修正)
- 1506行目
-
cui.h(v3のみ)- 94行目
strncpy(cui_title+1, AX_title[iw], sizeof(cui_title))-1)をstrncpy(cui_title+1, AX_title[iw], sizeof(cui_title)-1))に変更 (カッコの位置ミス、修正しないと未定義動作になり大体ファイル移動時に落ちる)
- 94行目
-
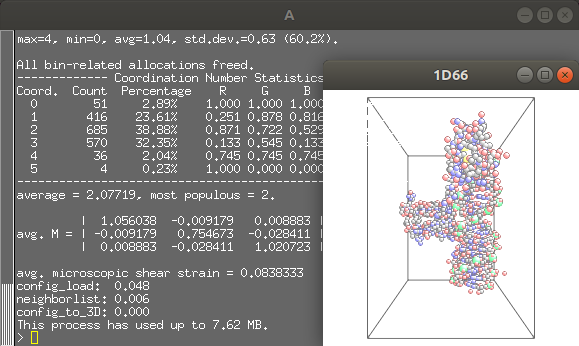
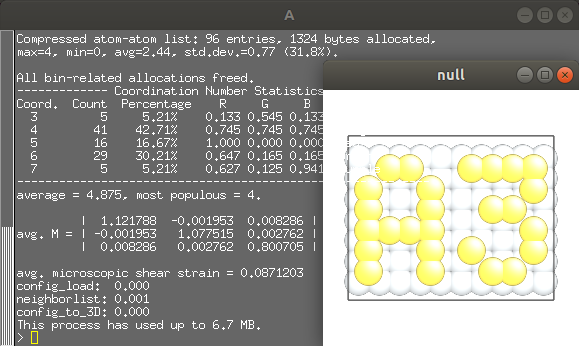
最終的にA3ディレクトリをmakeしたときに画像のように自動でAtomeyeが起動すれば成功です。終了はqキーです。
このとき表示される構造はDNAのようですね。
パラメータなしで起動すると銀の表面の上に金で"A3"と文字を書いた構造が表示されます。
実行の際の設定
呼び出すのが楽になるように、バイナリを指定した場所(上の例では~/my_path/bin)にパスを通しておきましょう。具体的には~/.bashrcの末尾などにexport PATH=${HOME}/my_path:${PATH}という行を書き足しておきます。
実行ファイル名は堂々のA。依存関係はないので適当に変えて大丈夫です。ただし実行ファイルの名前がpで終わると起動時に並列計算用の設定で起動するという独特の振る舞いがあるので、興味がなければ避けたほうが無難です。
また、Linuxデスクトップ環境で起動するとAtomeyeが透過してしまうことがよくあります。(上の画像でもそうなっています。)これを防ぐためには、環境変数の設定でexport XLIB_SKIP_ARGB_VISUALS=1を追加しておきましょう。ログインシェル自体にこれを入れていいかどうかはちょっと確認してください。(環境によってはデスクトップの描画がバグることがある。)安全な方法は例えばログインシェルの設定でalias A=XLIB_SKIP_ARGB_VISUALS=1 Aなどと設定する方法です。
おまけ: 巨大なディレクトリ対策
LAMMPSが長時間実行されて出力ディレクトリが巨大になってくると、Atomeye上でのファイルの前後移動が極めて緩慢になってきます。
これは描画そのものに時間がかかっているのではなく、ファイルの前後移動をするたびに毎回ディレクトリのファイル一覧を取得しているために発生する現象です。力任せなら出力ファイルをSSDとかRamdiskとかに置いておけば高速化します。逆に言うと、ファイルが遠距離にあるなどの遅延の大きなファイルシステムだとひどい目にあいます。
Atomeye実行中に追加されたファイルを読めるのは重要な機能なので捨てたくはありません。しかし、基本的にファイルの一覧は連番が大きくなるほうに一方的に増えていくのみです。このため事前に取得したファイル一覧を覚えていれば、ほとんどの場合はファイル一覧を更新しなくても次のファイル名はわかることになります。
この場合にファイル一覧を更新するべきタイミングは、「現在持っているファイル一覧の末尾にいるときに次ファイルを要求されたとき」と「ファイル一覧の先頭にいるときに前ファイル(末尾に戻る)を要求されたとき」だけでよいことになります。
このあたりを変更したければ、IO/filelist.cを書き換えましょう。
以下に解決案の一例を載せておきます。Numerically_sorted_glob_first関数を以下のように書き換えます。
char *Numerically_sorted_glob_advance (char *fname, int glob_advance)
{
static glob_t globbuf = {0, NULL, 0};
static int i = 0;
if (i == globbuf.gl_pathc)
{
numerically_sorted_glob(fname, &globbuf);
for (i=0; i<globbuf.gl_pathc; i++)
if (!strcmp(fname,globbuf.gl_pathv[i])) break;
if (i == globbuf.gl_pathc)
{
/* pe ("Numerically_sorted_glob_advance: \n" */
/* "glob list is not well defined.\n"); */
return(fname);
}
}
if((i + glob_advance) >= globbuf.gl_pathc || (i + glob_advance) < 0){
globfree (&globbuf);
numerically_sorted_glob(fname, &globbuf);
}
i = (i + glob_advance) % INT(globbuf.gl_pathc);
if (i < 0) i += globbuf.gl_pathc;
strcpy(fname, globbuf.gl_pathv[i]);
return(fname);
} /* end Numerically_sorted_glob_advance() */
ここでは全体の書き換えがなるべく少なくなるように書いているため、もっときれいに書くこともできると思います。
反映させるには一旦make cleanして再度makeしましょう。A3ディレクトリも同じです。