本記事サマリ
- K-Shape法を用いて時系列データをクラスタリングしてみる
データセット
今回はカリフォルニア大学リバーサイド校の時系列データコレクション(UCR Time Series Classification Archive)を使います
心電図(ECG)は心疾患検出を促進するものとして知られています
使用するECGFiveDays_TRAIN.tsvの中身は以下の通りです
df = pd.read_table("ECGFiveDays_TRAIN.tsv",header=None)
df.head(10)
データサマリはこちら
import numpy as np
import matplotlib.pyplot as plt
from tslearn.utils import to_time_series_dataset
data_train = np.loadtxt("ECGFiveDays_TRAIN.tsv")
X_train = to_time_series_dataset(data_train[:,1:])
print("時系列データの総数 : ",len(data_train))
print("クラスの数 : ", len(np.unique(data_train[:,0])))
print("時系列の長さ : ",len(data_train[0,1:]))
----------------------
#時系列データの総数 : 23
#クラスの数 : 2
#時系列の長さ : 136
136スナップある時系列データが計23本含まれております。欠損値はありません。
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
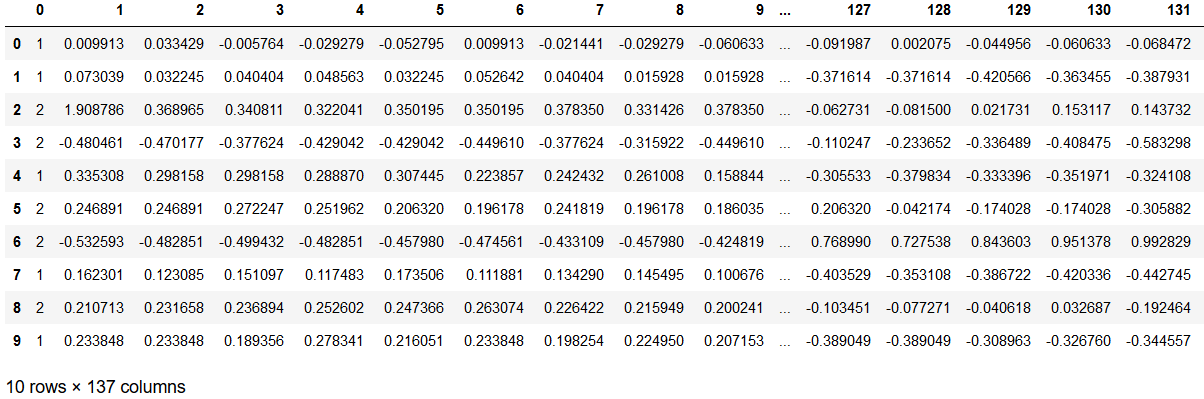
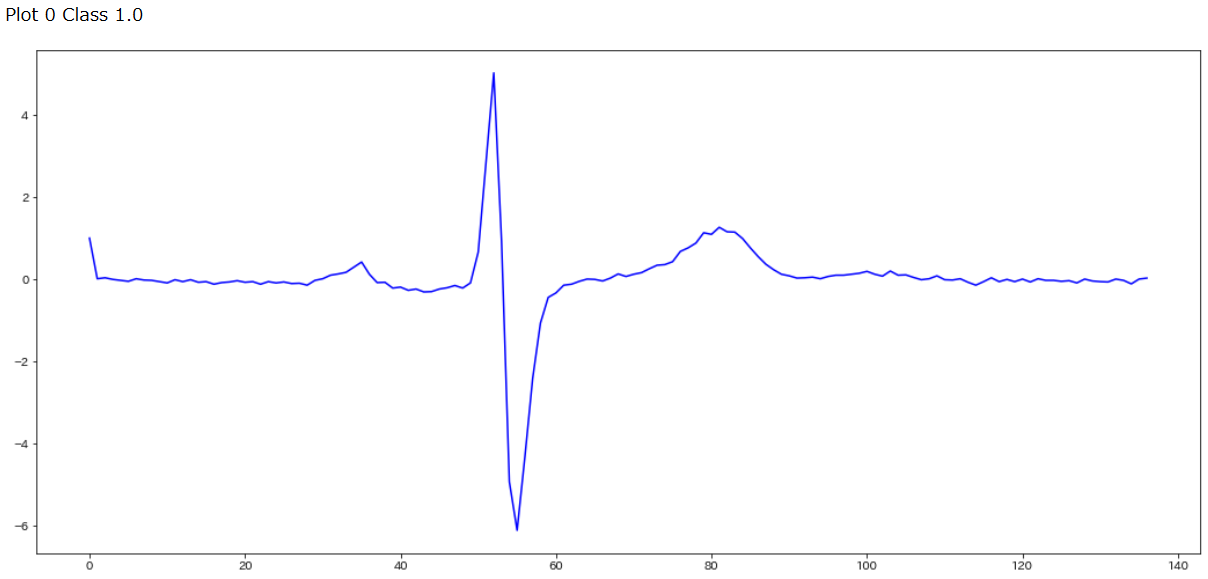
for i in range(0,3):
if data_train[i,0]==2.0:
plt.figure(figsize=(18, 8))
print("Plot",i,"Class",data_train[i,0])
plt.plot(data_train[i],c='r')
plt.show()
これらのラベルは心電図の専門家がつけたラベルなので、クラス1.0もクラス2.0も素人の目だとほとんど区別がつきません。また補足で、ECGFiveDays_TRAIN.tsvと一緒にダウンロードされるReadme.mdによるとこの心電図は1990年に測定された67歳の男性のECGデータであるそうです。
前処理
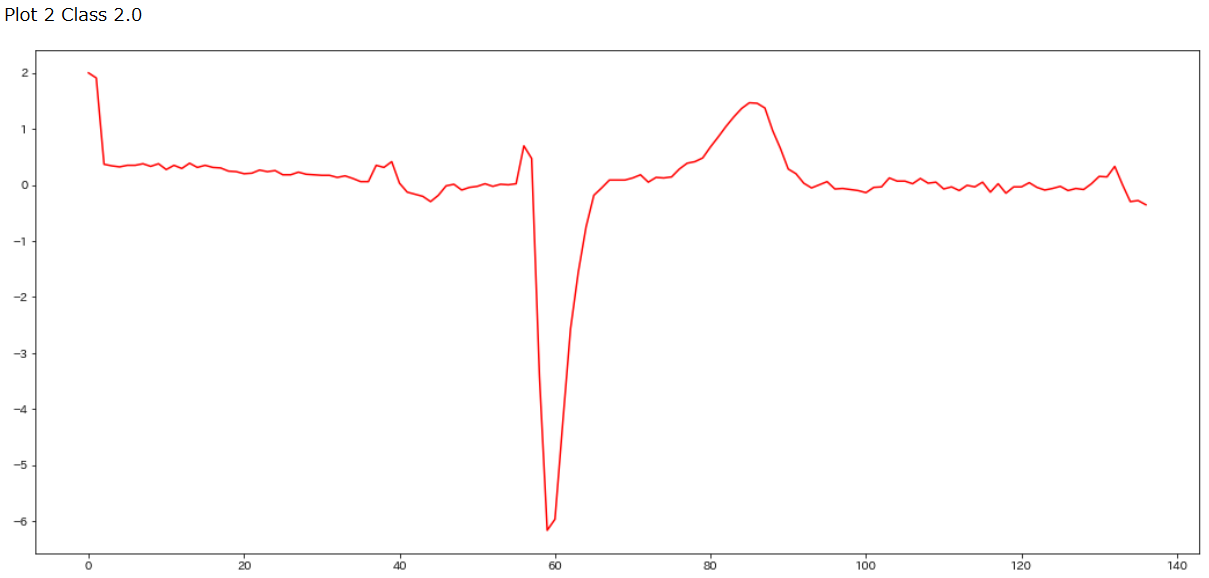
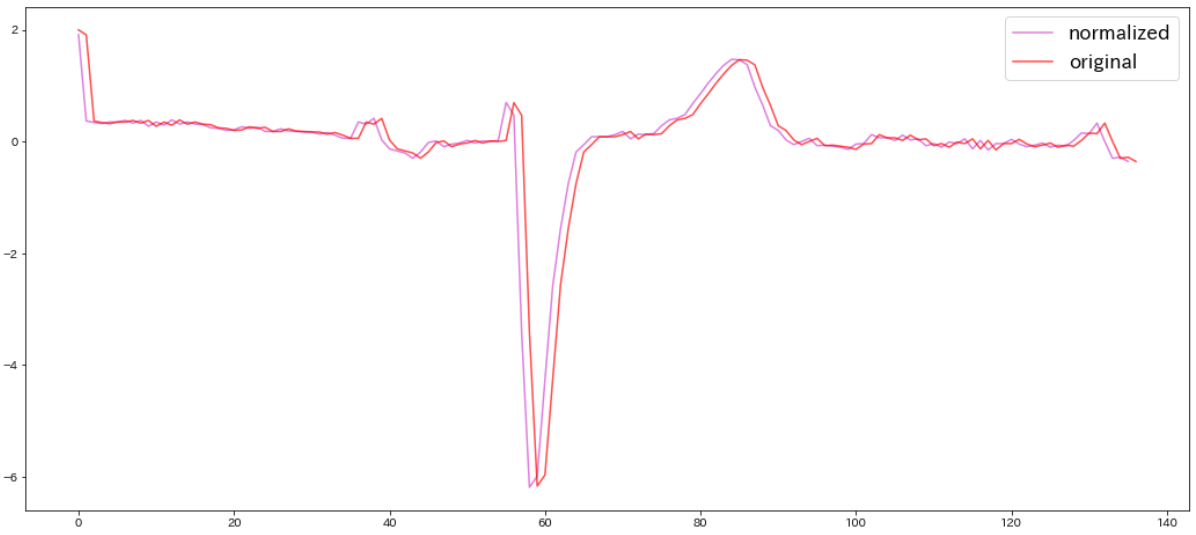
K-Shapeに持ち込むために、各時系列データを平均0、標準偏差1になるように正規化します。ここでは先ほどのPlot0,class1.0の心電図を正規化し、グラフ化して比較してみます。
from tslearn.preprocessing import TimeSeriesScalerMeanVariance
from tslearn.utils import to_time_series_dataset
X_train = to_time_series_dataset(data_train[:,1:])
X_train = TimeSeriesScalerMeanVariance(mu=0.,std=1.).fit_transform(X_train)
plt.figure(figsize=(18, 8))
plt.plot(X_train[1],c='magenta',alpha=0.8,label="normalized")
plt.plot(data_train[1],c='blue',alpha=0.8,label="original")
plt.legend(fontsize=17)
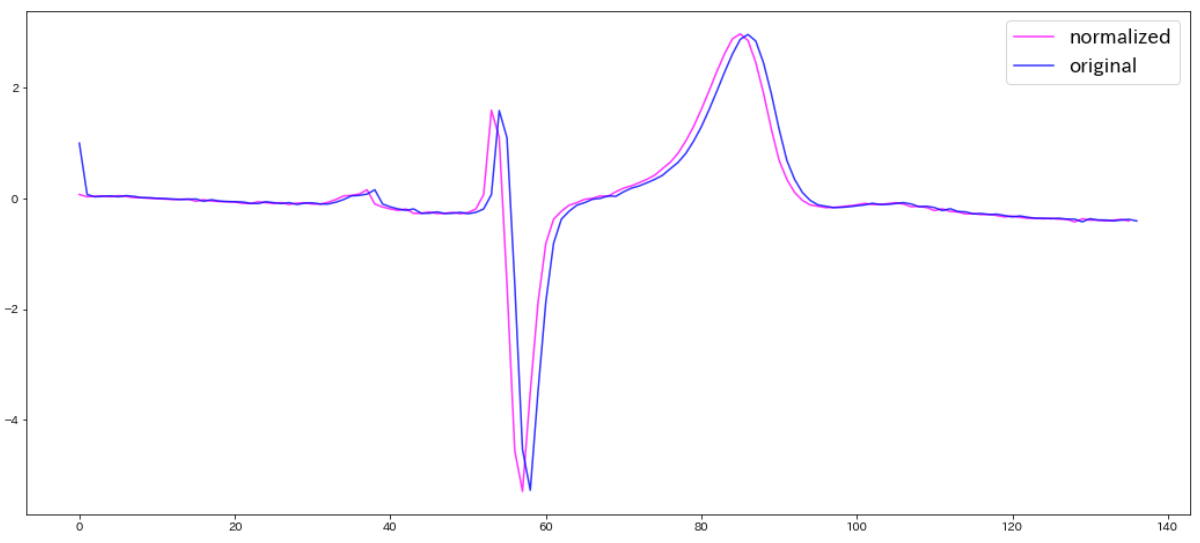
※先ほどのクラス2.0の赤いグラフも正規化したものと比較すると下図のようになります。
K-Shape法について
端的に3点でまとめてみます。
- 時系列データを対象とした形状ベースのクラスタリング手法です
- 基本的にはk-meansと同じですが、距離の測り方が違います
- 下記がK-Shapeの論文です。2015年に書かれたものなのでアルゴリズム自体は比較的新しいです( https://dl.acm.org/doi/10.1145/2723372.2737793)
いざ、訓練
クラスタ数を2、最大繰り返し回数100、訓練回数を100として実行します。
from tslearn.clustering import KShape
ks = KShape(n_clusters=2,max_iter=100,n_init=100,verbose=0)
ks.fit(X_train)
y_pred = ks.fit_predict(X_train)
print(y_pred)
---
#[0 1 0 0 1 0 0 1 0 1 1 1 1 0 0 0 1 0 1 1 1 0 1]
計23本の時系列に対して、2種類のクラスどちらに分類されるか予測ラベルを付けることが出来ました。K-Shapeに関するハイパーパラメータは下記URLを参照しております。
https://tslearn.readthedocs.io/en/stable/gen_modules/clustering/tslearn.clustering.KShape.html
評価
時系列クラスタリングの評価尺度として、調整ランド指標を用います。一言で言うと、この指標は「予測されたクラスタリングと真のクラスタリングの間でどれだけクラスタ割り当てが一致しているか」を測るものです。
ここではScikit-learnのadjusted_rand_scoreを用います。
https://scikit-learn.org/stable/modules/generated/sklearn.metrics.adjusted_rand_score.html
from sklearn.metrics import adjusted_rand_score
ars = adjusted_rand_score(data_train[:,0],y_pred)
print("調整ランド指標 : ",ars)
---
#調整ランド指標 : 0.668041237113402
次はテストデータに対するarsを求めます。
import numpy as np
import matplotlib.pyplot as plt
from tslearn.utils import to_time_series_dataset
data_test = np.loadtxt("ECGFiveDays_TEST.tsv")
X_test = to_time_series_dataset(data_test[:,1:])
pred_test = ks.predict(X_test)
ars = adjusted_rand_score(data_test[:,0],pred_test)
print("調整ランド指標 : ",ars)
---
#調整ランド指標 : 0.06420517898316379
とても低いスコアが出てしまいました。
0に近いスコア=ランダム割り当てした時のスコア
というロジックなので、良いモデルであるとは決して言えません。テストデータに対するスコアが低い理由としては「圧倒的にデータ数が少ない」からと考えられます。そこでデータ数を増やして同じトライしてみます。
ECG5000データを使って訓練/評価
以下では、UCR Archiveに梱包されているECG5000_TRAIN.tsvとECG500_TEST.tsvを使用します。
import numpy as np
import matplotlib.pyplot as plt
from tslearn.utils import to_time_series_dataset
from sklearn.model_selection import train_test_split
data_test_ = np.loadtxt("ECG5000_TEST.tsv")
data_train_ = np.loadtxt("ECG5000_TRAIN.tsv")
data_joined = np.concatenate((data_train_,data_test_),axis=0)
data_train_,data_test_ =train_test_split(data_joined,test_size=0.2,random_state=2000)
X_train_ = to_time_series_dataset(data_train_[:,1:])
X_test_ = to_time_series_dataset(data_test_[:,1:])
print("時系列データの総数 : ",len(data_train_))
print("クラスの数 : ", len(np.unique(data_train_[:,0])))
print("時系列の長さ : ",len(data_train_[0,1:]))
---
#時系列データの総数 : 4000
#クラスの数 : 5
#時系列の長さ : 140
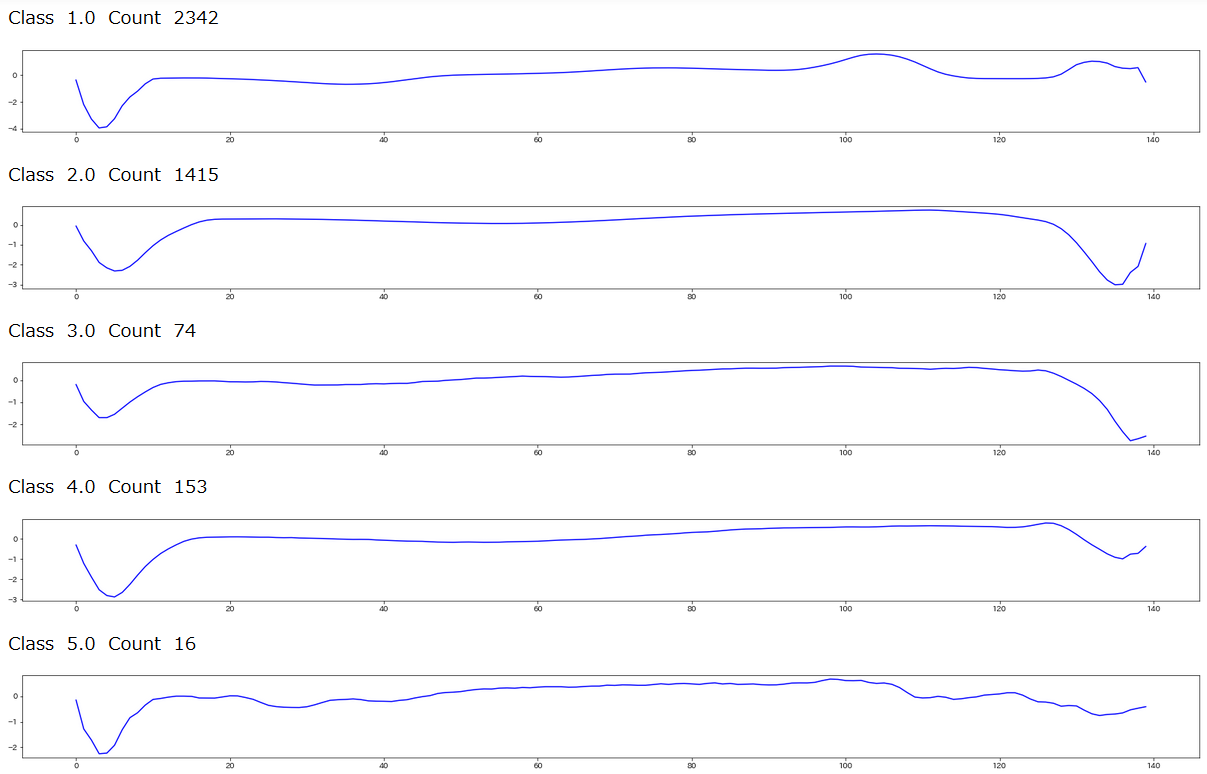
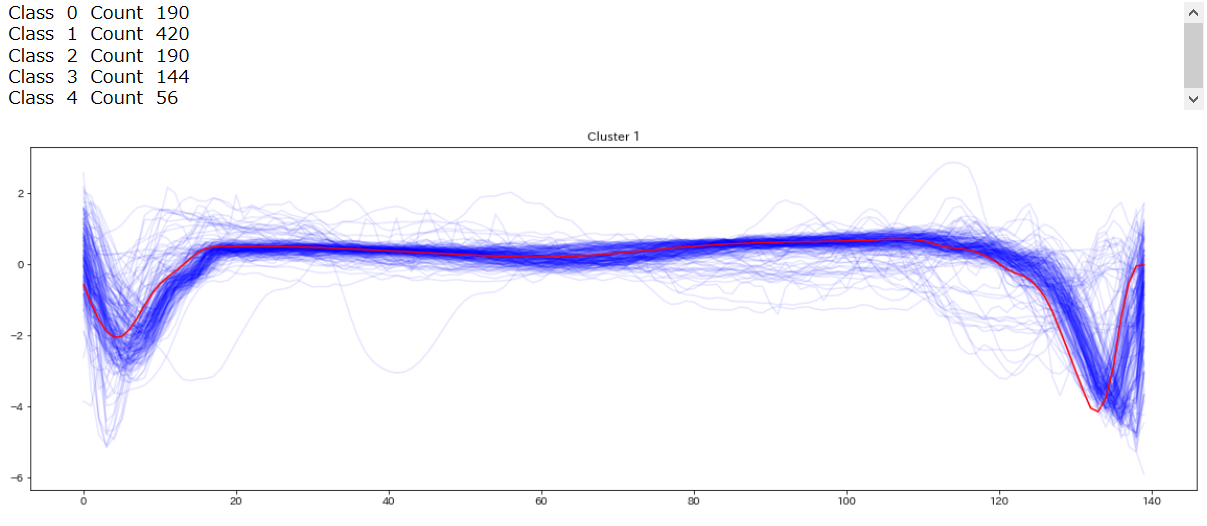
クラスタに属しているデータ数はこちら
print("クラス1.0に属するデータ数",len(data_train_[data_train_[:,0]==1.0]))
print("クラス1.0に属するデータ数",len(data_train_[data_train_[:,0]==2.0]))
print("クラス1.0に属するデータ数",len(data_train_[data_train_[:,0]==3.0]))
print("クラス1.0に属するデータ数",len(data_train_[data_train_[:,0]==4.0]))
print("クラス1.0に属するデータ数",len(data_train_[data_train_[:,0]==5.0]))
---
#クラス1.0に属するデータ数 2342
#クラス1.0に属するデータ数 1415
#クラス1.0に属するデータ数 74
#クラス1.0に属するデータ数 153
#クラス1.0に属するデータ数 16
グラフにするとこんな感じです
%matplotlib inline
import matplotlib.pyplot as plt
for j in np.unique(data_train_[:,0]):
plt.figure(figsize=(28,2))
dataPlot = data_train_[data_train_[:,0]==j]
cnt = len(dataPlot)
dataPlot = dataPlot[:,1:].mean(axis=0)
print(" Class ",j," Count ",cnt)
plt.plot(dataPlot,c='b')
plt.show()
先ほどと同様に訓練&評価していきます
from tslearn.preprocessing import TimeSeriesScalerMeanVariance
from tslearn.utils import to_time_series_dataset
from tslearn.clustering import KShape
from sklearn.metrics import adjusted_rand_score
X_train_ = to_time_series_dataset(data_train_[:,1:])
X_train_ = TimeSeriesScalerMeanVariance(mu=0.,std=1.).fit_transform(X_train_)
X_test_ = to_time_series_dataset(data_test_[:,1:])
X_test_ = TimeSeriesScalerMeanVariance(mu=0.,std=1.).fit_transform(X_test_)
ks = KShape(n_clusters=5,max_iter=100,n_init=100,verbose=1,random_state=2000)
ks.fit(X_train_)
preds_test = ks.predict(X_test_)
ars_ = adjusted_rand_score(data_test_[:,0],preds_test)
print("調整ランド指標 : ",ars_)
---
#調整ランド指標 : 0.4984050982000773
テストデータに適用したときの評価ランド指標は、約0.498となりました。先ほどのデータ適用と比べると雲泥の差です。訓練セットのサイズを23本から4000本にしたことではるかに精度の良いモデルが作成されました。
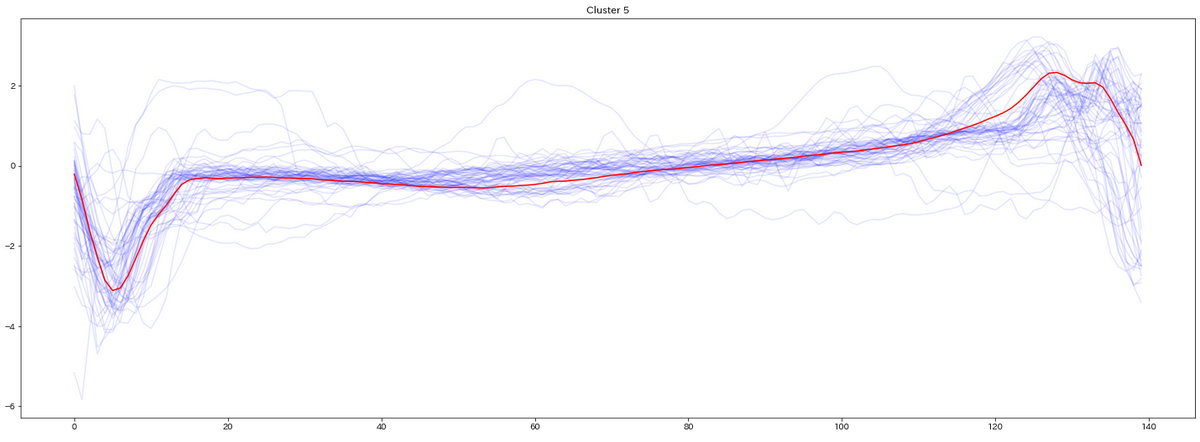
最後にテストデータを見てみます
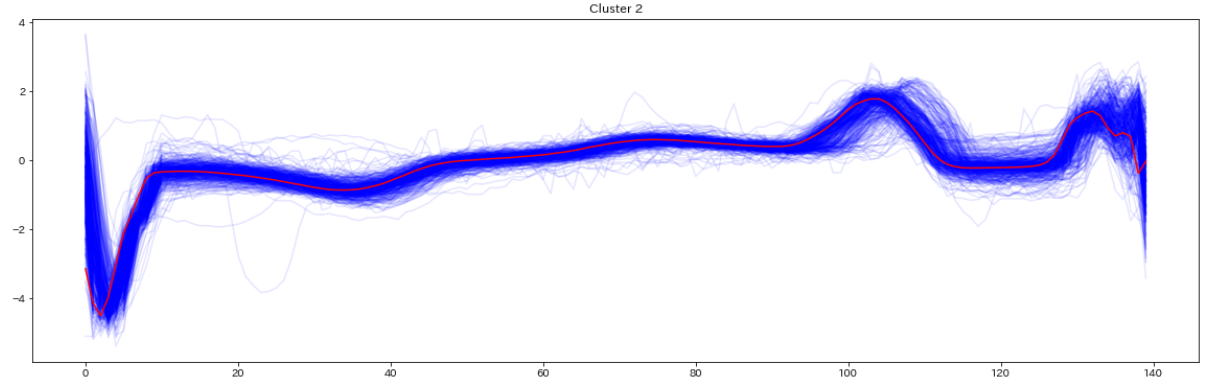
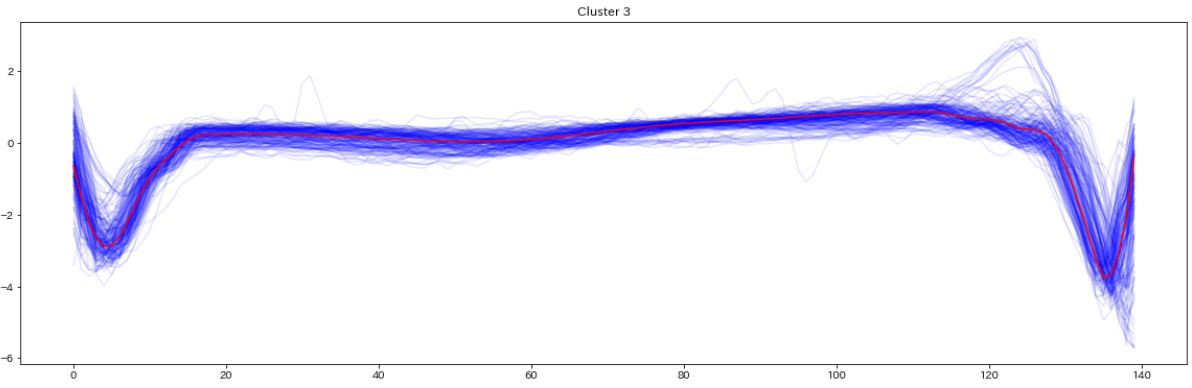
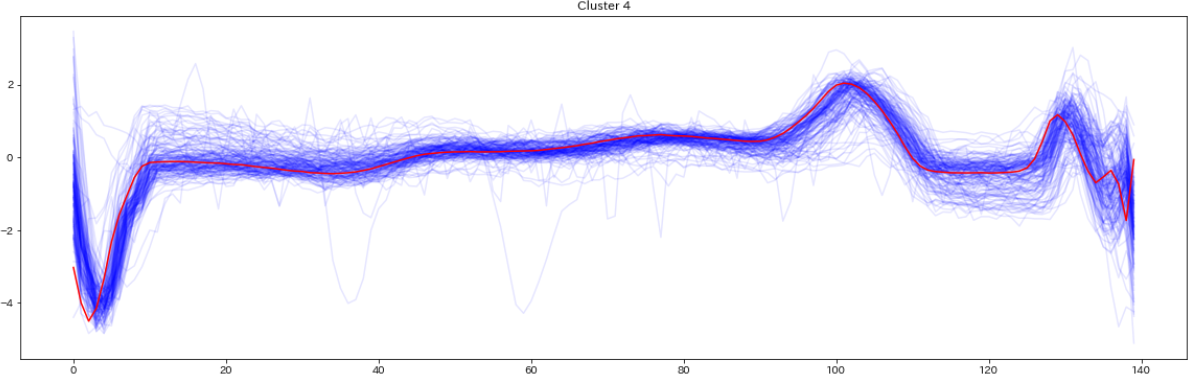
for yi in range(5):
plt.figure(figsize=(20,35))
plt.subplot(5, 1, 1 + yi)
for xx in X_test_[preds_test == yi]:
plt.plot(xx.ravel(), alpha=0.1,c='blue')
cnt = len(X_test_[preds_test == yi])
plt.plot(ks.cluster_centers_[yi].ravel(), "red")
print(" Class ",yi," Count ",cnt)
plt.title("Cluster %d" % (yi + 1) )
plt.tight_layout()
plt.show()
テストデータの分類結果を可視化したものがこちら(赤い線はセントロイド)
まとめ
- 今回は形状ベースのK-Shapeと心電図データを使って時系列クラスタリングを試みました
- データ数を増やすことで、調整ランド指標のスコアが爆上がりしました
- K-Shape以外にもk-meansやDBSCANでもクラスタリングできるので今後比較も行いたい
補足
K-Shapeの距離測定
SBD(\vec{x},\vec{y})=1-\underset{w}{max}\frac{CC_w(\vec{x},\vec{y})}{\sqrt{R_0(\vec{x},\vec{x})}\sqrt{R_0(\vec{y},\vec{y})}}
- 時系列データに上手く適合する類似度、相互相関$CC_w$を導入しています
- $SBD$は $\vec{x},\vec{y}$ の距離を意味しています
tslearnとは
tslearnは機械学習を使った時系列分析用のPythonパッケージです。 K-Shape以外にも様々な時系列分析アルゴリズムが含まれています