はじめに
クラスター分析を行った際に表示させるデンドログラム、クラスタリングの構造が一目で見れて非常に便利である反面、RやPython等で出力した結果は基本画像ファイルになるため下記の点で使いにくい点がある。
- 変数が大量にある場合、サイズ調整などを行ってもラベルが潰れてしまう。
- 画像ファイルであることから当然ラベルの検索ができない。
デンドログラムを画像ではないフォーマットに変換する必要があり、表現の自由度などを考えるとd3.jsが適切と考える。既存の記事を探したものの、以下の点で望みのものが見つからなかった。
- d3.jsのバージョンが古い(v3の記事がいくつか見つかる)。
- デンドログラムの高さの情報が反映されない(ここがデンドログラムの本質でもある)。
そこで、同様に悩んでいる人もいるだろうということで、変換するスクリプトの作成にトライしてみた。
できたこと
- Rで描いたデンドログラムをd3.jsに変換した。
やったこと
基本的な戦略としては、
- デンドログラムを作成する。
- デンドログラムをJSONデータに変換する。
- JSONデータをHTMLテンプレートに流し込む。
となる。ここで足りない機能は「デンドログラムをJSONに変換する」機能と「d3.jsでデンドログラムを表示させるHTMLテンプレート」となるので、この2つを開発した。
Rでデンドログラムを作成
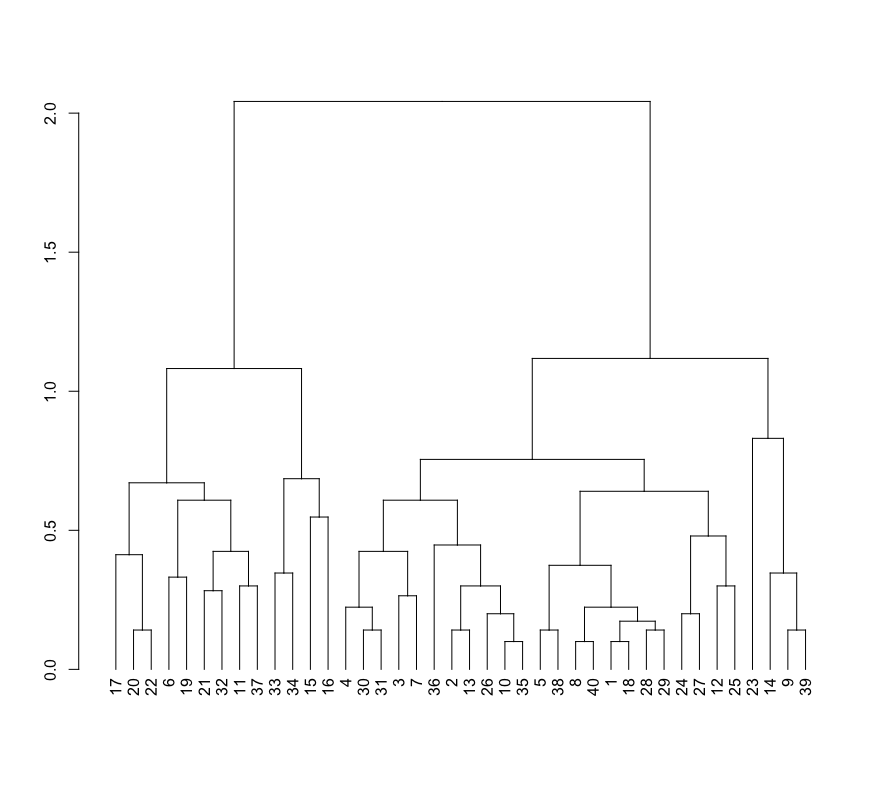
まずはお馴染みのアヤメデータを使って階層クラスタリングを行う。適度なサイズ感にするため、150あるデータのうち上から40行のみを抽出してクラスタリングする (setosaだけになってしまうが、今回はクラスタリングの内容は興味ないので放置)。
library(tidyverse)
library(dendextend)
data(iris)
ddg <- iris[1:40,1:4] %>%
dist() %>%
hclust() %>%
as.dendrogram()
plot(ddg)
ここまでで作成されたデンドログラムが上記の「Rで作成したデンドログラム」になる。
デンドログラムをJSONに変換
以下の記事を参照してデンドログラムをJSON形式に変換する関数を定義する。
d3 dendrograms with R
as.json.dendrogram <- function(d){
# internal helper function
add_json <- function(x){
v <- attributes(x)
lab <- ifelse(is.null(v$label), "", v$label)
json <<- paste(json,sprintf('{ "name" : "%s", "h" : %s',lab,v$height))
if ( is.leaf(x) ){
json <<- paste(json, "}\n")
} else {
json <<- paste(json, ',\n "children" : [' )
for ( i in seq_along(x) ){
add_json(x[[i]])
s <- ifelse(i<length(x), ",", "")
json <<- paste(json, s)
}
json <<- paste(json, " ]}")
}
}
json <- ""
add_json(d)
json
}
HTMLテンプレート
sprintfを用いてテンプレート文字列を生成する。基本的にはHTML文字列の「data = 」部分に先ほどの関数を用いて生成したJSONを埋め込む構成になっている。ここで書いているd3.jsのコードは解説が必要なものであると思われるが、とりあえず今回は結果を生成できるということを記事の主旨とするため省略する。
convertD3js <- function(json){
sprintf('
<!DOCTYPE html>
<html>
<head>
<meta charset="utf-8">
<title>Dendrogram with d3.js</title>
<script src="https://d3js.org/d3.v7.min.js"></script>
<style>
.link {
fill:none;
stroke:#555;
stroke-opacity: 0.4;
stroke-width: 1.5px;
}
</style>
</head>
<body>
<div id="dendrogram"></div>
<script>
var data = %s;
let width = 1000;
let height =1200;
let margin = 100;
let heightMax = data.h;
let heightMin = 0;
let scale = d3.scaleLinear()
.domain([heightMin, heightMax])
.range([width - margin, 0]);
let root = d3.hierarchy(data);
let cluster = d3.cluster()
.size([height - margin, width - margin]);
cluster(root)
let svg = d3.select("#dendrogram")
.append("svg")
.attr("width", width)
.attr("height", height);
svg.append("g")
.attr("transform", "translate(" + margin / 2 + "," + margin / 2 + ")")
.call(d3.axisTop(scale));
let g = svg.append("g")
.attr("transform", "translate(" + margin / 2 + "," + margin / 2 + ")");
let link = g.selectAll(".link")
.data(root.descendants().slice(1))
.enter()
.append("path")
.attr("class", "link")
.attr("d", function(d){
return "M" + scale(d.data.h) + "," + d.x +
"L" + scale(d.parent.data.h) + "," + d.x +
"L" + scale(d.parent.data.h) + "," + d.parent.x;
});
let node = g.selectAll(".node")
.data(root.descendants())
.enter()
.append("g")
.attr("transform", function(d){
return "translate(" + scale(d.data.h) + "," + d.x + ")";
});
node.append("text")
.attr("dy", 5)
.attr("x", 0)
.style("text-anchor", function(d){
return d.children ? "end" : "start";
})
.attr("font-size", "15px")
.text(function(d){
return d.data.name;
});
</script>
</body>
</html>', json)
}
HTMLを生成
ここまでの結果を統合する。出力はhtmlファイルとなる。svgのサイズ、フォントサイズなどはハードコーディングになっているので、出力を見て適宜調整してほしい。
html <- ddg %>%
as.json.dendrogram()
convertD3js()
writeLines(html, "hoge.html")
まとめ
Rで出力したデンドログラムをd3.jsで表示するスクリプトを開発した。向きが横向きになるなど課題はあるものの、基礎技術はこれでカバーできていると考える。