神経活動をコンピュータ上でシミュレーションする研究は以前から行われています。
シミュレータとしてはイェール大学のNEURONが有名ですが、他にも様々なソフトがあります。
今回はその中でも、去年(2019年)にeLifeで論文発表されたBrian2(バージョン1とは異なる)を使ってみます。
*間違えやすいですが、名前は**Brain(脳)ではなくてBrian(ブライアン)**ですね。
ホームページ:https://briansimulator.org/
GitHub:https://github.com/brian-team/brian2
論文:https://elifesciences.org/articles/47314
開発元はフランスのソルボンヌ大学などです。
インストール
環境は
Ubuntu 16.04
Anaconda
Python 3.6
です。
インストールはcondaでもpipでも出来るようです。
pip install brian2
これでインストールできました。
バージョンはbrian2-2.3.0.2でした。
次項からドキュメントのチュートリアルを参考にしながら基本的なところをやってみます。
動かしてみる
まずは単純な膜電位シミュレーションをやってみます。
インポートします。
(この項ではどれがモジュールかを分かりやすくするために、あえてマニュアル通りのfrom brian2 import *ではなく普通にインポートします。)
import brian2
Brian2には物理単位がモジュールになっていて、Hzやmsなどをそのまま定義することができます。
これを使うことで、パラメータや定数を簡潔に記述できます。
また、計算時に物理単位が揃わないとエラーになるようになっているので設定ミスを防ぐことができます。
v0 = -10 * brian2.mV
tau = 10 * brian2.ms
シミュレーションを始めるときにはまずstart_scope()を入れます。
brian2.start_scope()
微分方程式(膜電位の変化を定義する)はString型で定義するようです。
eqs = 'dv/dt = (v0-v)/tau : volt (unless refractory)'
上記のvoltは変数の物理単位です。SI単位で表します。
ここでは膜電位が変数になります。
(unless refractory)は不応期を使う場合に必要です。
神経細胞はNeuronGroupモジュールで定義します。
G = brian2.NeuronGroup(1, eqs, threshold='v > -40*mV', reset='v = -60*mV', refractory=5*brian2.ms, method='exact')
第一引数は神経細胞の数です。今回は1個で設定しました。
第二引数は膜電位の微分方程式です。
他の引数は
thresholdはスパイクになる閾値電位
resetはスパイク後のリセット電位
refractoryは不応期
methodは数値計算の方法
を設定しています。
膜電位の初期値を設定します。
G.v = -70 * brian2.mV
StateMonitorによってモニタリングができます。
M = brian2.StateMonitor(G, 'v', record=True)
シミュレーションはrun(<時間>)によって開始されます。
brian2.run(50*brian2.ms)
これでシミュレーションできました。
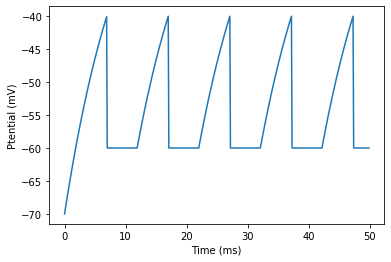
結果をプロットします。
brian2.plot(M.t/brian2.ms, M.v[0]/brian2.mV)
brian2.xlabel('Time (ms)')
brian2.ylabel('Ptential (mV)');
この結果はLIFモデルのニューロンのスパイクをシミュレーションした結果になります。(ピーク電位がありませんが。)
これでシミュレーションの流れが分かりました。
発展として、xi(標準正規分布の確率変数)を使うことで、ノイズを含んだ確率微分方程式を扱うこともできるようです。
複数の神経細胞のスパイクシミュレーション
次に複数の神経細胞のスパイクをシミュレーションします。
今回は各細胞に異なる入力電位(I)を与えてみます。
インポートからやっていきます。
from brian2 import *
start_scope()
N = 100 #細胞の数
tau = 10*ms #膜時定数
v0 = -50*mV #静止膜電位
eqs = '''
dv/dt = (v0-v+I)/tau : volt (unless refractory)
I : volt
'''
G = NeuronGroup(N, eqs, threshold='v>-40*mV', reset='v=-60*mV', refractory=1*ms, method='exact')
G.v = -70*mV
膜電位変化ではなくスパイクのみを見たいときはSpikeMonitorを使います。
M = SpikeMonitor(G)
各細胞に異なる入力を定義します。
I_max = 40*mV
G.I = 'i*I_max/(N-1)'
iは細胞のインデックスになります。これで異なる入力を定義できました。
runします。
duration = 1000*ms
run(duration)
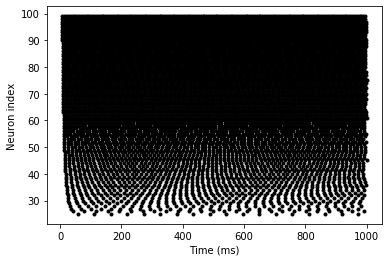
ラスタープロットを作成します。
plot(M.t/ms, M.i, '.k')
xlabel('Time (ms)')
ylabel('Neuron index');
細胞のインデックスが上がるごとに入力電位が上がるので、スパイクが多くなりました。
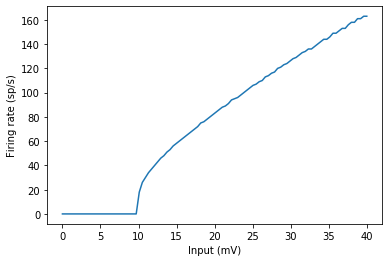
よく見るF-Iカーブも作ってみます。
plot(G.I/mV, M.count/duration)
xlabel('Input (mV)')
ylabel('Firing rate (sp/s)');
神経回路をシミュレーションするには
詳細は割愛しますが、神経回路のシミュレーションも簡単に出来るようです。
シナプスはSynapsesモジュールで定義できます。
connectメソッドで細胞の結合が行われます。(どの細胞からどの細胞へつなげるか)
シナプスの強度に微分方程式を定義することもできて、STDPモデルも作れるようです。
他にもNetworkモジュールでネットワークの定義ができたりするようです。
感想
シミュレーションのための機能が充実していて、使いやすいと思いました。
ドキュメントには他にも様々な例が載っています。
https://brian2.readthedocs.io/en/stable/examples/index.html
コンパートメントモデルの例もありました。
これらも試してみたいですね。
*追記①
今年の1月にBrian2GeNNというものが発表されていました。
https://www.nature.com/articles/s41598-019-54957-7
これでGPUも活用できるようになったみたいです。
*追記②
またbrian2toolsというライブラリもあり、これを使うと可視化が簡単に出来るようになって便利です。
https://brian2tools.readthedocs.io/en/stable/