こんにちは、Exploratoryの白戸です。
日本ではまだ一部のメディアがコロナウイルスに関しての報道を続けていますが、コロナウイルス以外の感染症についてはあまり報道されていません。コロナウイルスは話題性もあるためか、特殊なウイルスとしてメディアは取り上げていますが、実は毎年流行している季節性のインフルエンザなどでも死亡してしまうことがあります。
果たして、コロナウイルスの死亡者数は、他の感染症と比較して高いと言えるのでしょうか?その疑問に答えるために、e-Statという政府がデータを公開・運営しているサービスから感染症における死亡者数のデータを取得していきます。
e-Statで公開されているデータは、Hiroaki Yutani氏が開発されたRパッケージのestatapiを使うことで、簡単に取得することができます。
このパッケージを使うと、e-Statから下記のようなデータを取得することができます。
- 統計表情報
- メタ情報
- 統計データ
- データカタログ情報
このパッケージを使うと簡単にデータを取ってくることができるのですが、e-Statのデータは「汚い」ことで有名です。
というものエクセルでただ数字を眺めるぶんにはいいのですが、これをデータとして分析したり可視化するには、「データの前処理職人」を喜ばせるほどの様々なデータラングリングの技を必要とします。
今回は、RのUIとしても有名なExploratoryを使って、厚生労働省が公開している「感染症による死因」のデータをe-Statから取得し、そのデータを加工、可視化する方法について紹介したいと思います。
事前準備
Exploratoryのインストール
Exploratoryはこちらのページからサインアップした後、ダウンロードしてインストールできます。
パブリック版は無料でお使いいただけます。さらに学生や先生であればコミュニティ版が使えますがこちらも無料です。
下記のノートにて、詳しいセットアップ方法が書かれていますので、ぜひお試しください。
Exploratoryのセットアップ方法 - リンク
e-StatのアプリケーションIDを取得する
e-StatのAPIを利用するには、アプリケーションIDを取得する必要があります。
こちらの手順に従って、アプリケーションIDの取得をしてください。
APIにアクセスする際は、appIdというパラメータに取得したアプリケーションIDを指定します。
estatapiパッケージをインストールする
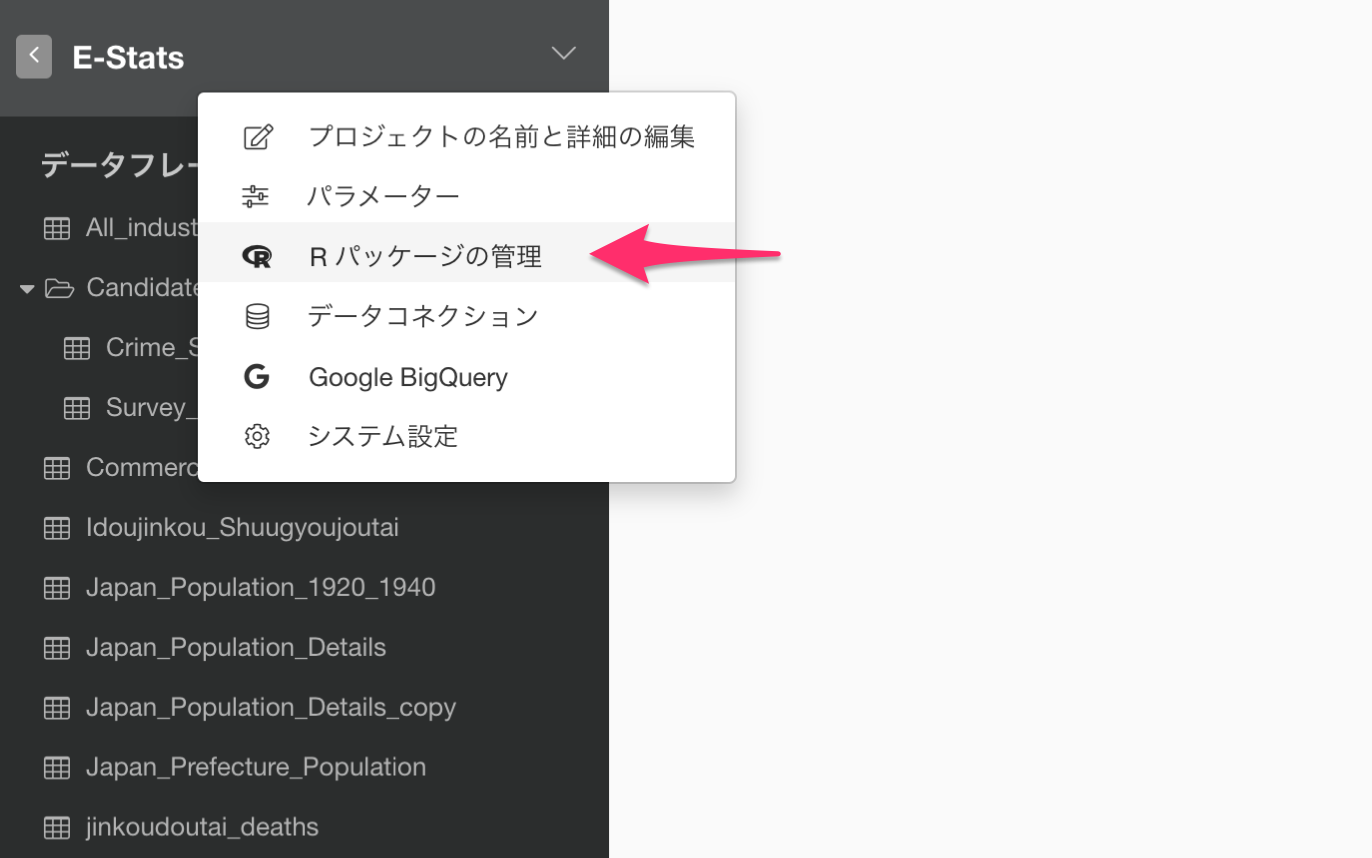
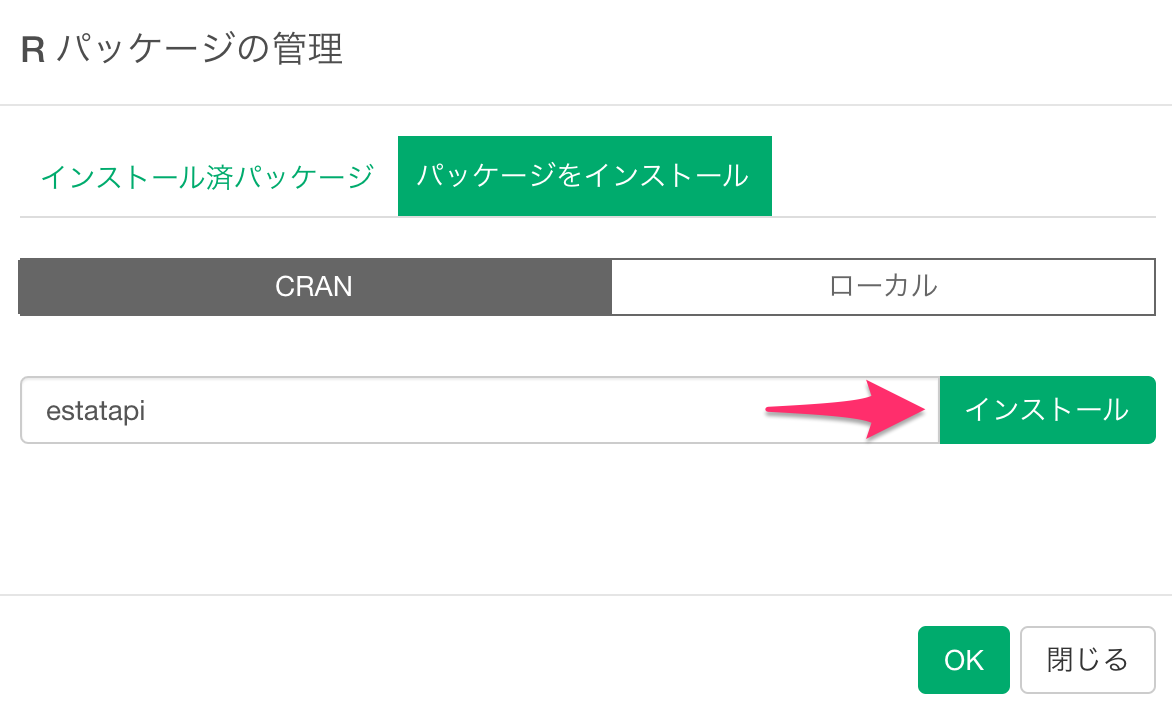
プロジェクトのメニューから、「R パッケージの管理」を選択して、パッケージをインストールします。
「パッケージをインストール」のタブを選んで、「estatapi」とタイプして、「インストール」ボタンを押します。
インストールが終われば、後は使うだけです!
データのIDを取得する
今回は、感染症に関連する統計データのリストを取得していきます。
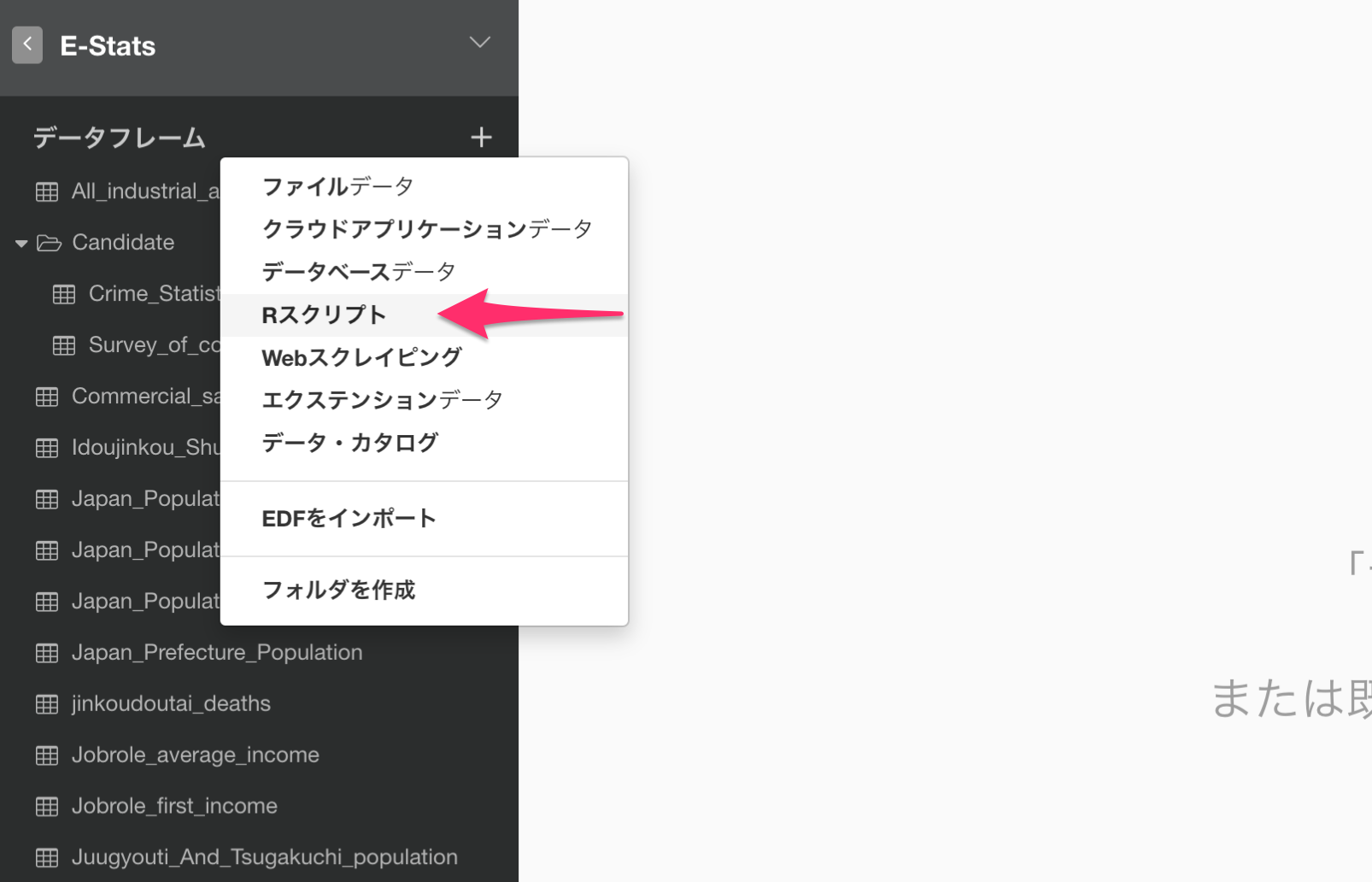
estatapiの関数を使うためにRスクリプトを書き、それをデータソースとしたいので、データフレームの横から「Rスクリプト」というメニューを選択します。
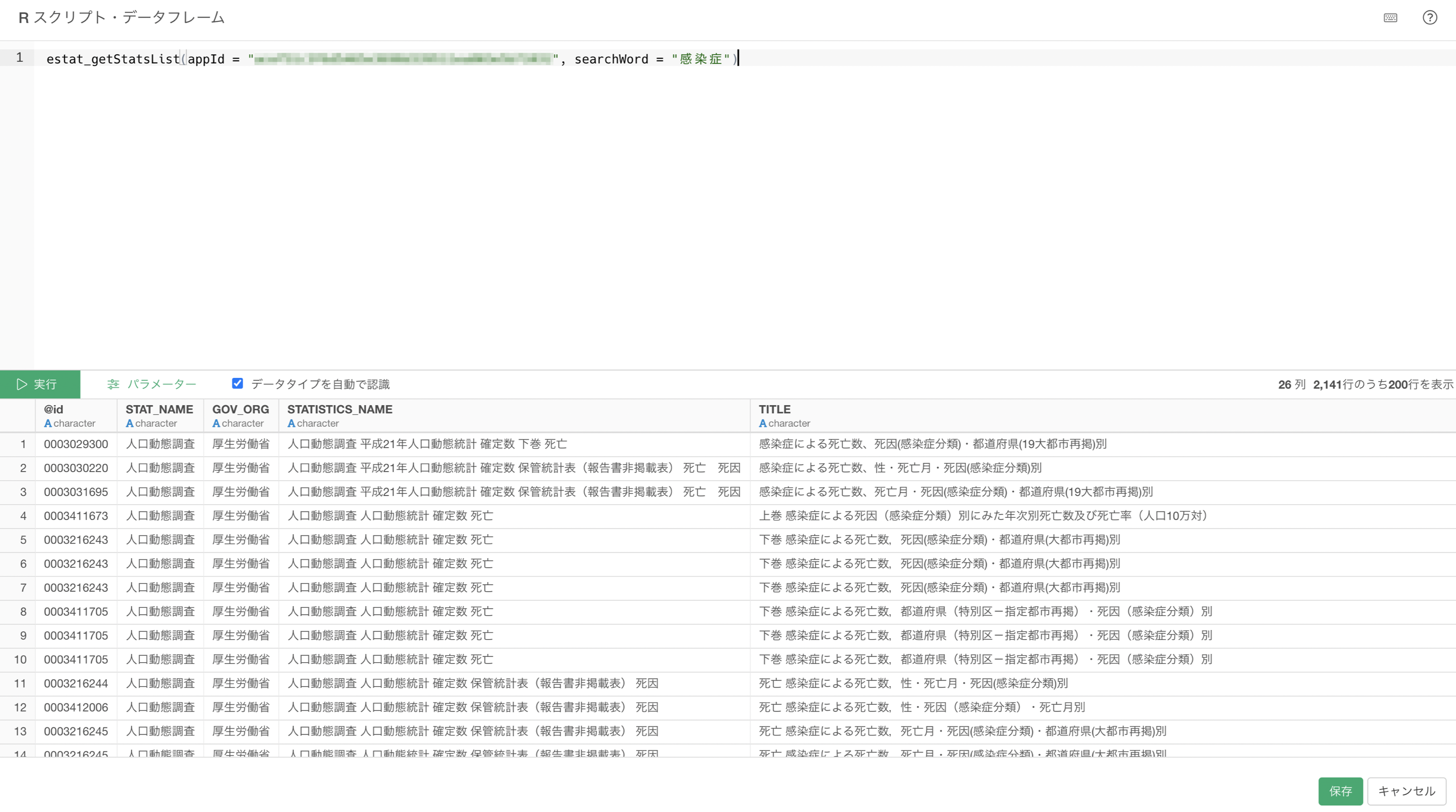
Rスクリプト・データフレームのダイアログが表示されるので、下記のスクリプトを入力して実行します。今回は検索キーワードに、感染症と入力して実行します。
estat_getStatsList(appId = <アプリケーションID>, searchWord = <検索キーワード>)
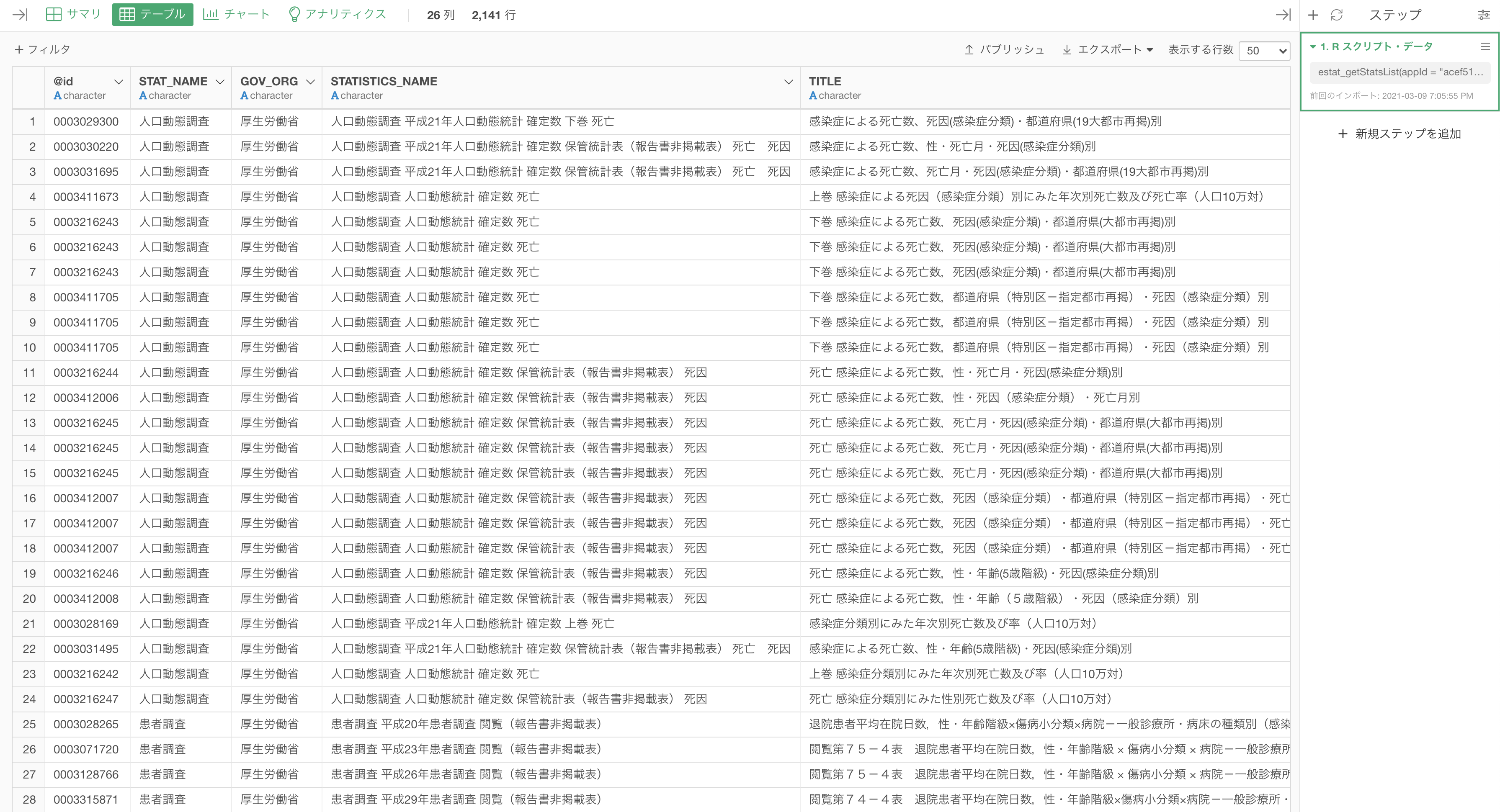
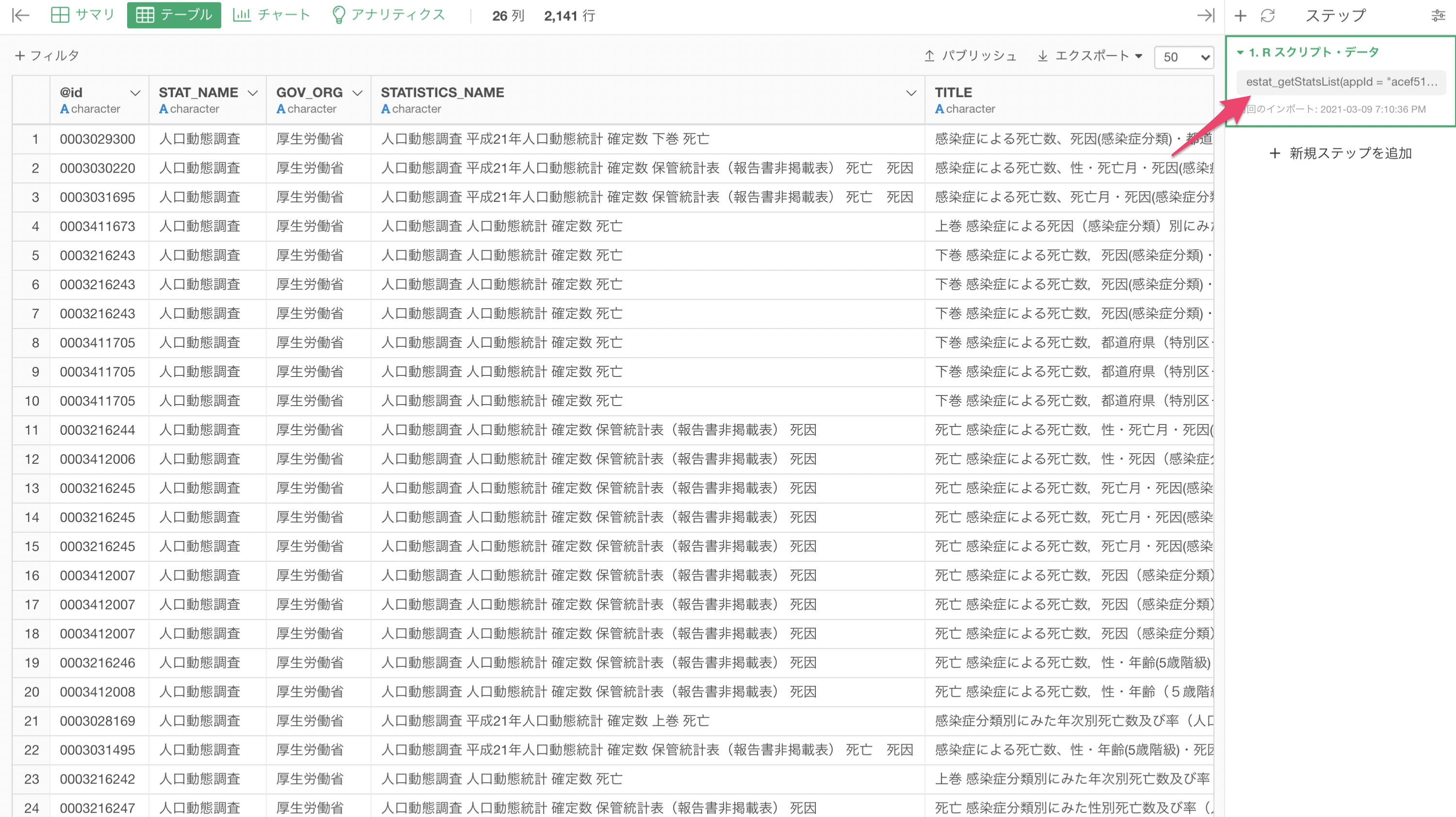
実行すると、関連した統計表が表示されます。
e-Statから「感染症」のキーワードで統計データのリストを取得してデータフレームとして保存することができました。
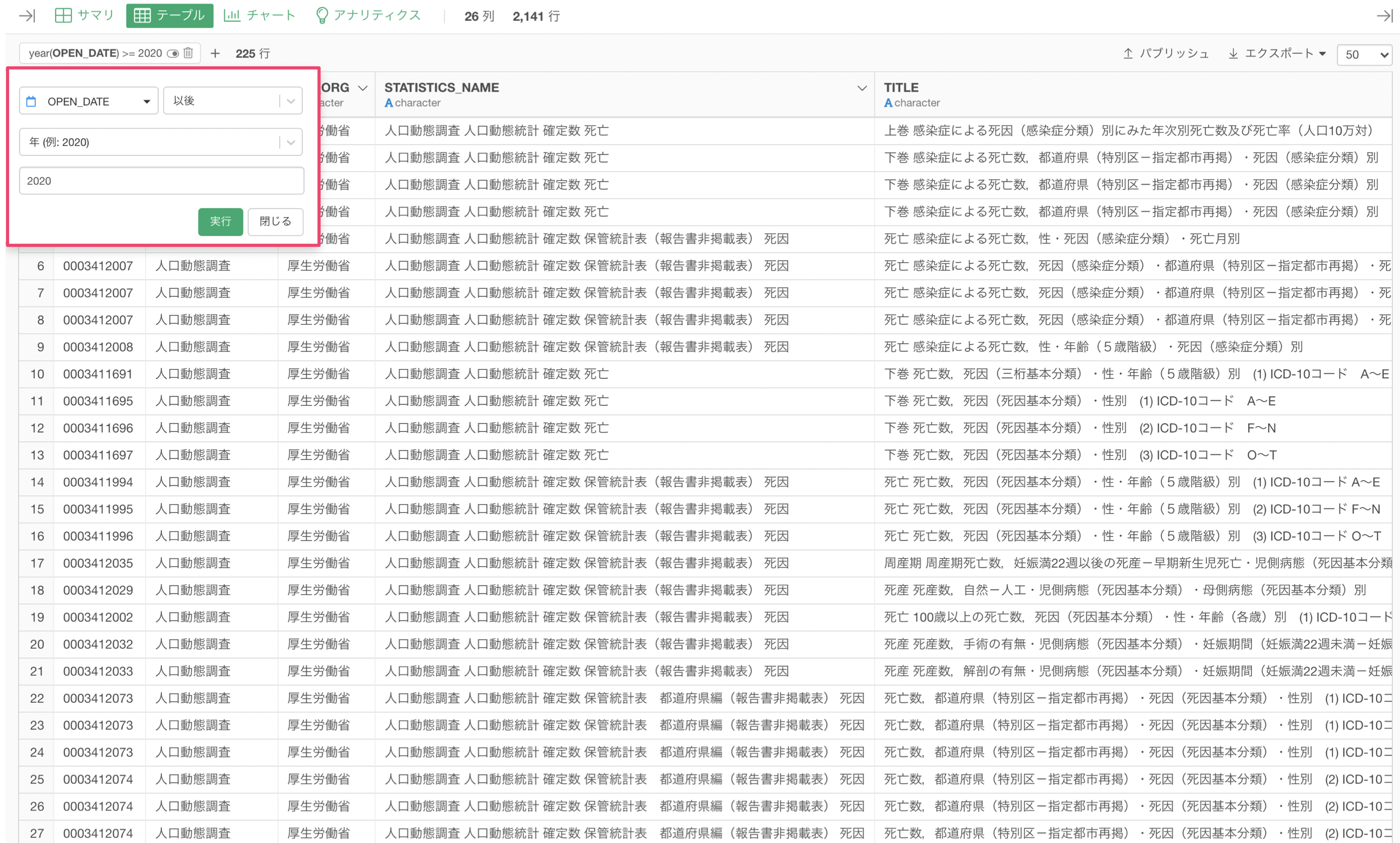
キーワードによっては取得される統計データのリスト数が多くなってしまうので、テーブルビューで使えるフィルタ機能を使って絞り込むことができます。
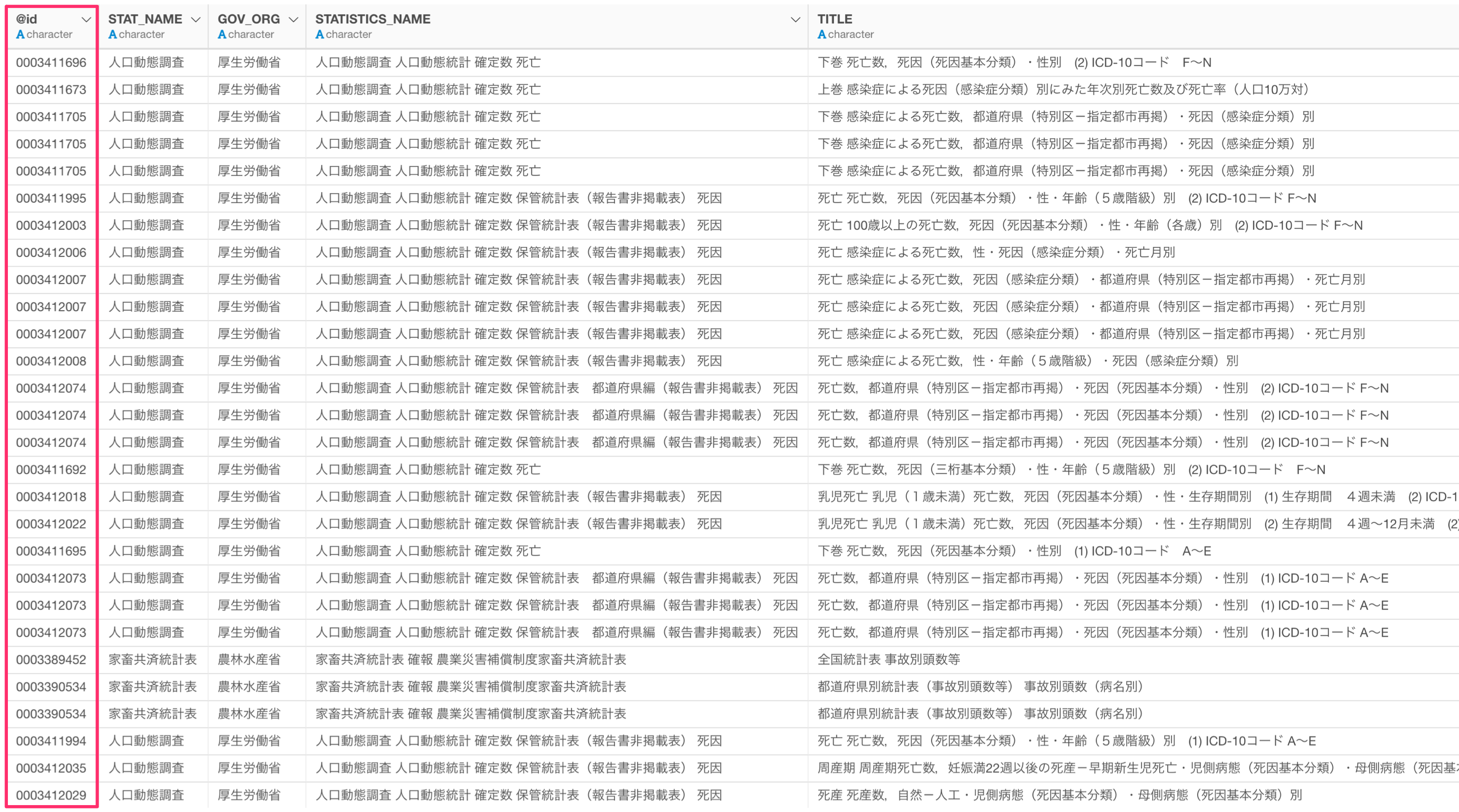
統計表の@id列が統計データの一意なIDになり、統計データを取得する際に必要になります。
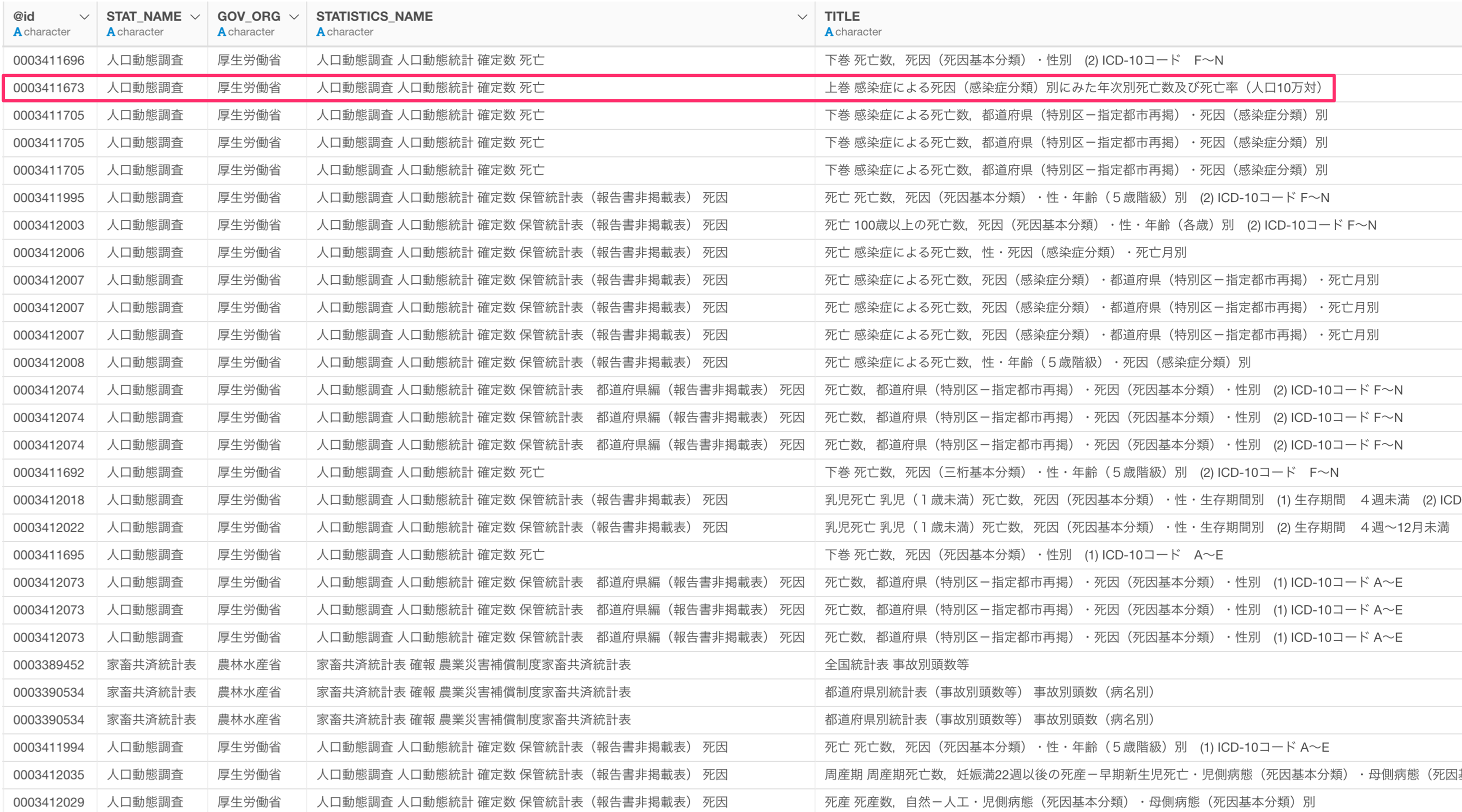
今回は、厚生労働省が公開している「人口動態調査の感染症による死因」のデータを使用したいため、@id列にある「0003411673」をコピーしておきます。
統計データを取得する
それでは、「人口動態調査の感染症による死因」データのIDがわかったので、これからいよいよそのデータ自体をとってみましょう。
データソースのステップの中のトークンをクリックします。
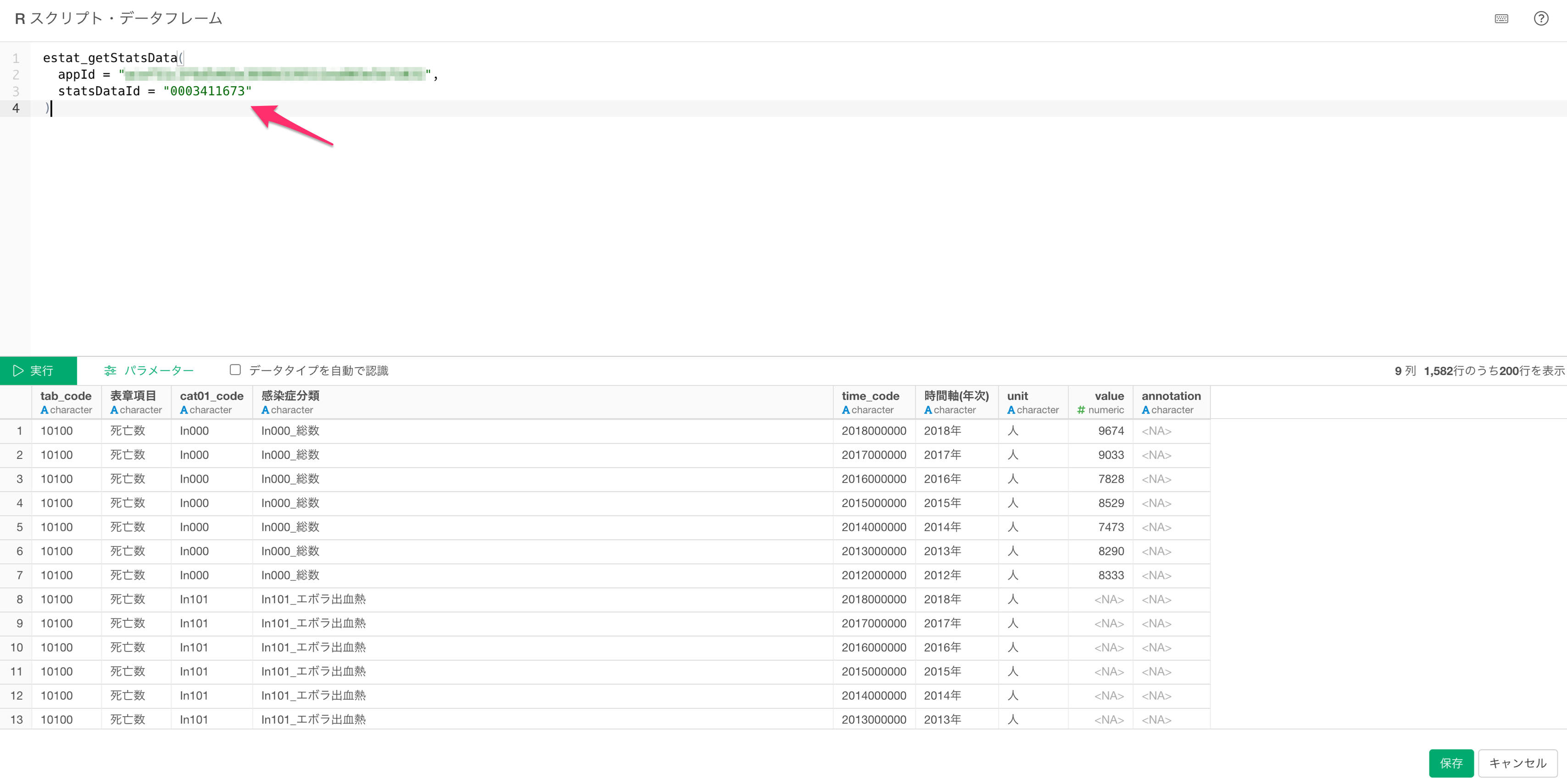
Rスクリプト・データフレームのダイアログが表示されるので、下記のスクリプトを実行します。
estat_getStatsData(
appId = <アプリケーションID>,
statsDataId = <統計データのID>
)
statsDataIdには統計表の@id列にある値を入力します。今回は、厚生労働省が公開している「人口動態調査の感染症による死因」のデータを取得するためstatsDataIDに"0003411673"を指定します。
「保存」ボタンをクリックするとデータがインポートされます。
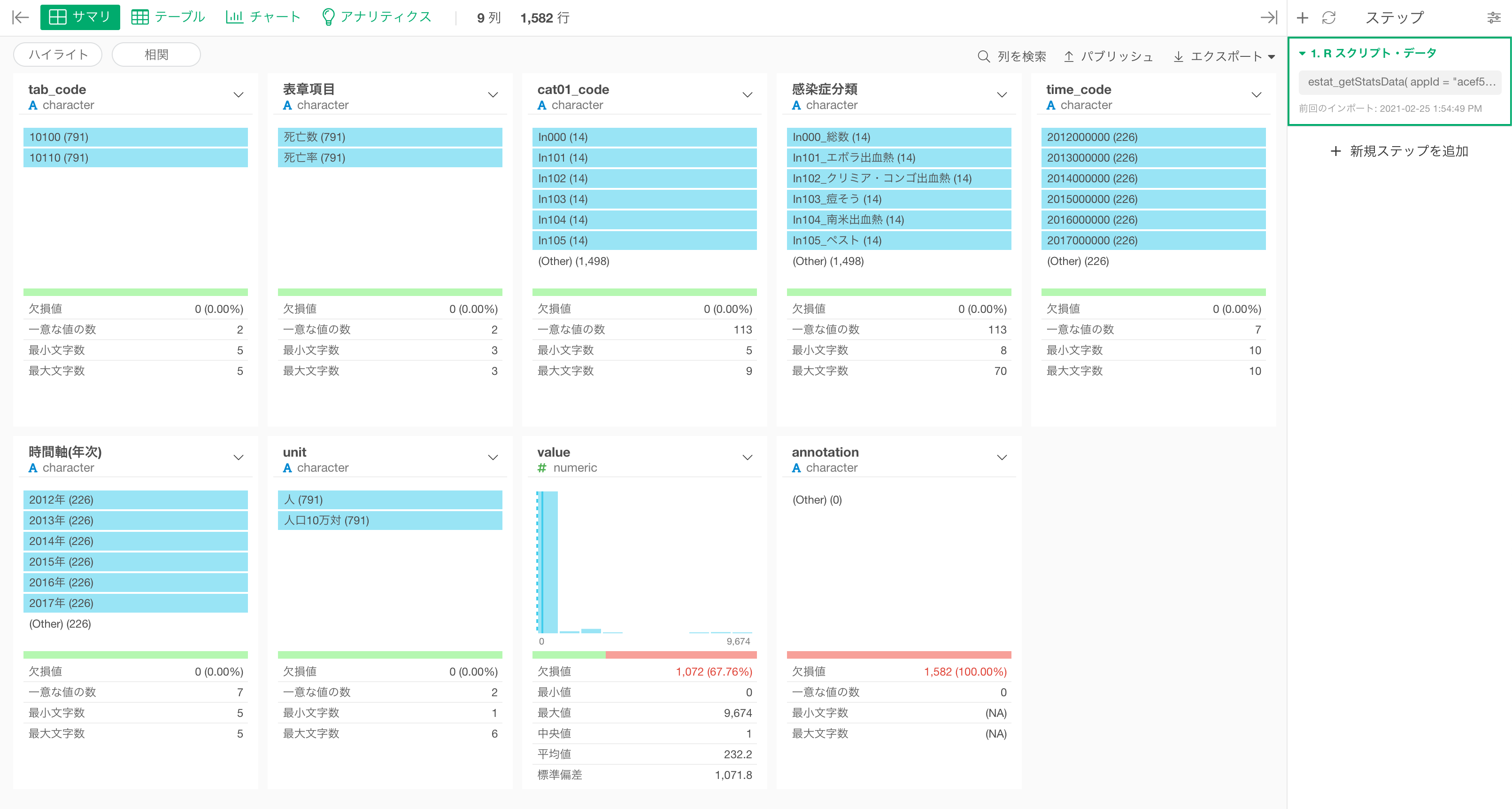
最初にデータのサマリ情報が自動生成されます。ここからは各列の基本統計値やデータの分布、欠損値の数などを速攻で把握することができます。
データを使える形に加工・整形する
データはインポートできましたが、多くの場合は可視化やアナリティクスで使える形になっていません。
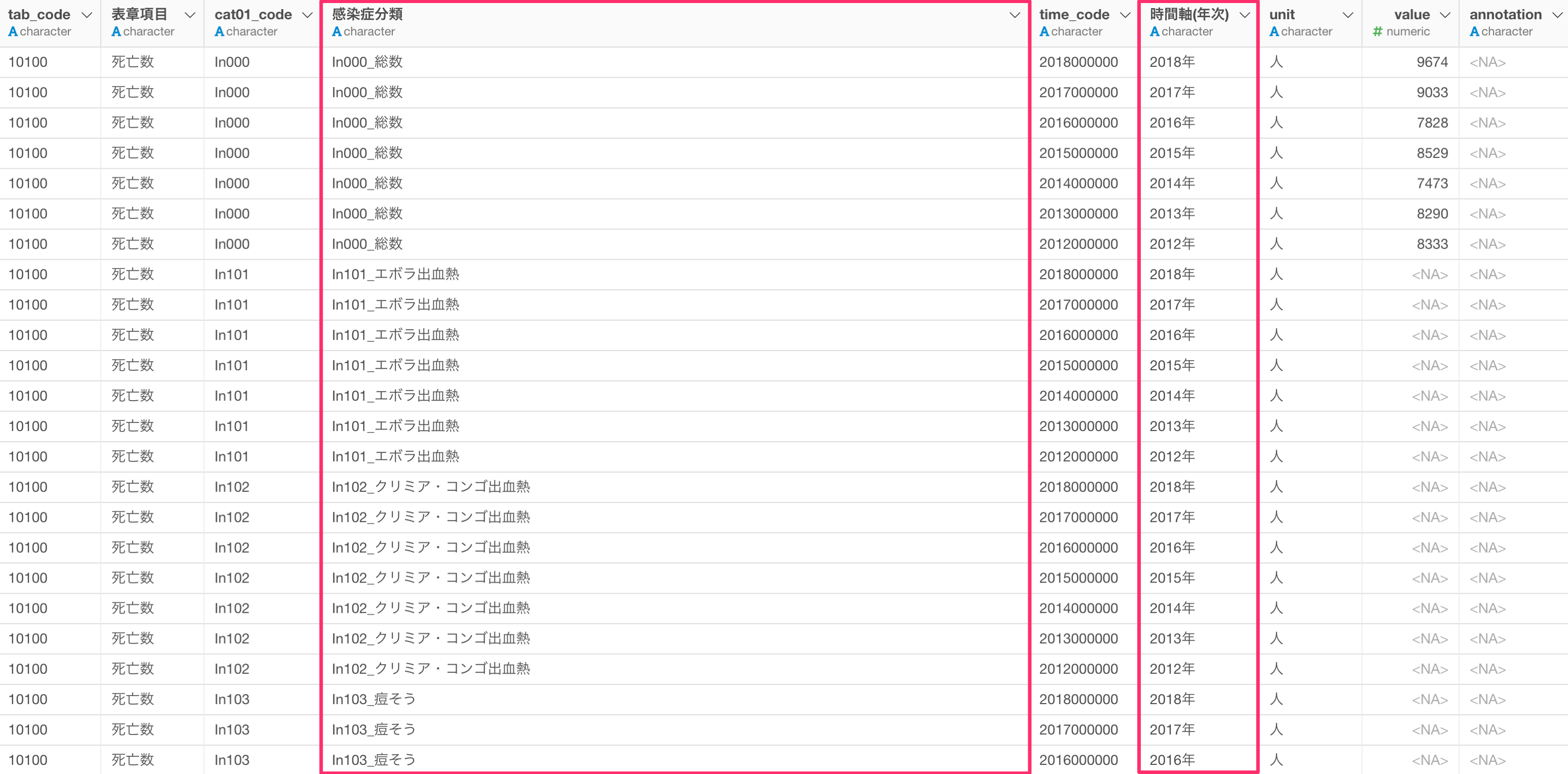
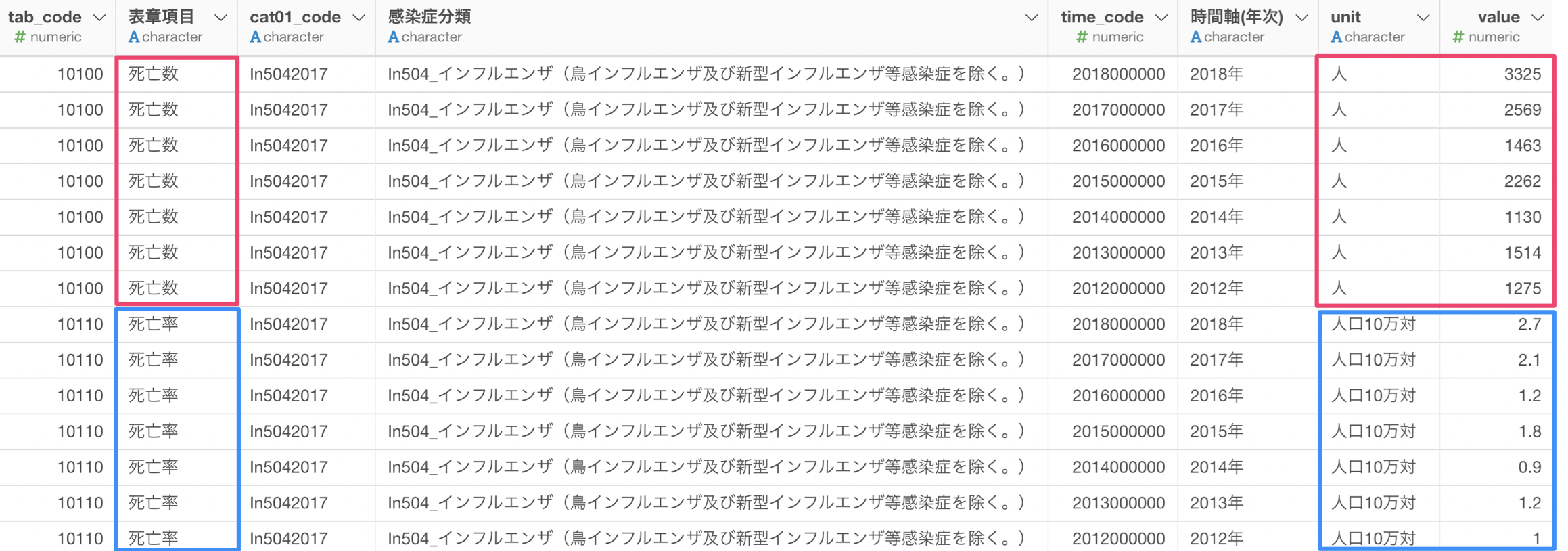
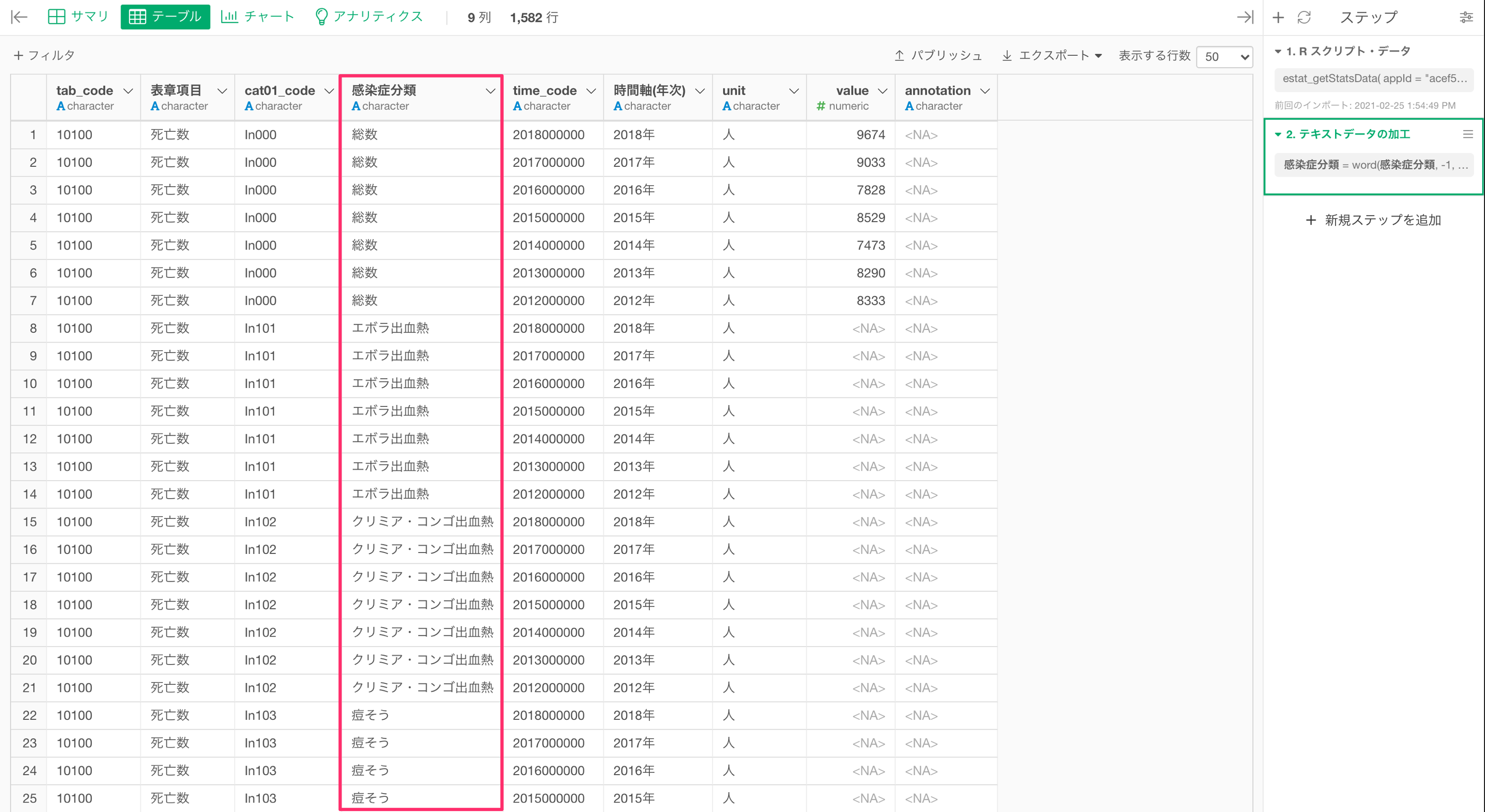
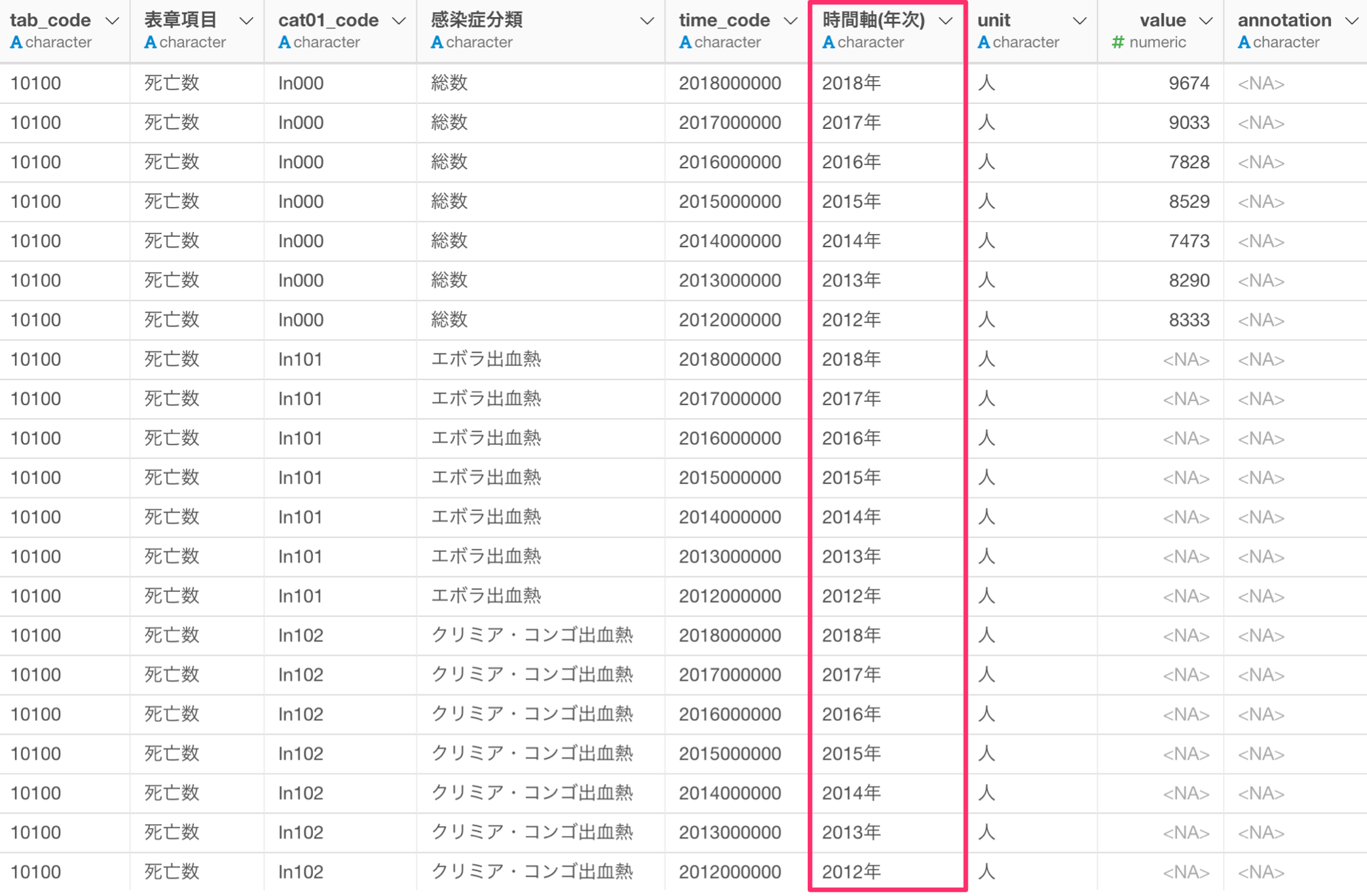
このデータではいくつかの問題があります。例えば、感染症分類の列に、カテゴリーの分類コードと感染症名がまとめられていたり、時間軸(年次)の列は数値型になっていません。
そして、死亡者数と人口10万人あたりの死亡率が同じ列にまとめられており、死亡者数と死亡率をいっしょに可視化してしまう可能性があります。
そのため、データラングリングをして可視化やアナリティクスで使えるように、加工・整形する必要があります。今回は下記のように、1行が感染症の分類と年ごとにして、死亡数と死亡率を別々の列にしたデータにしていきます。
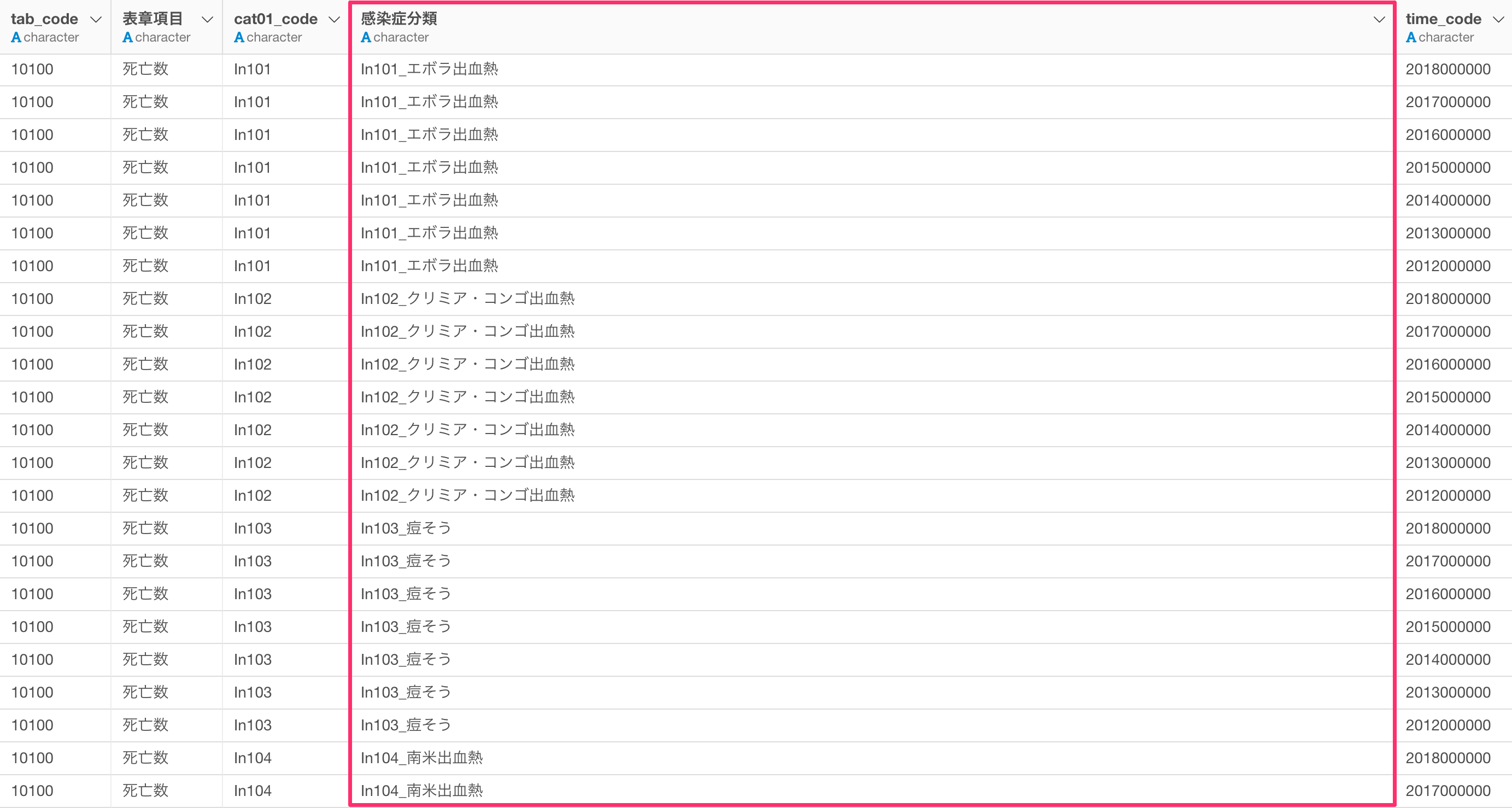
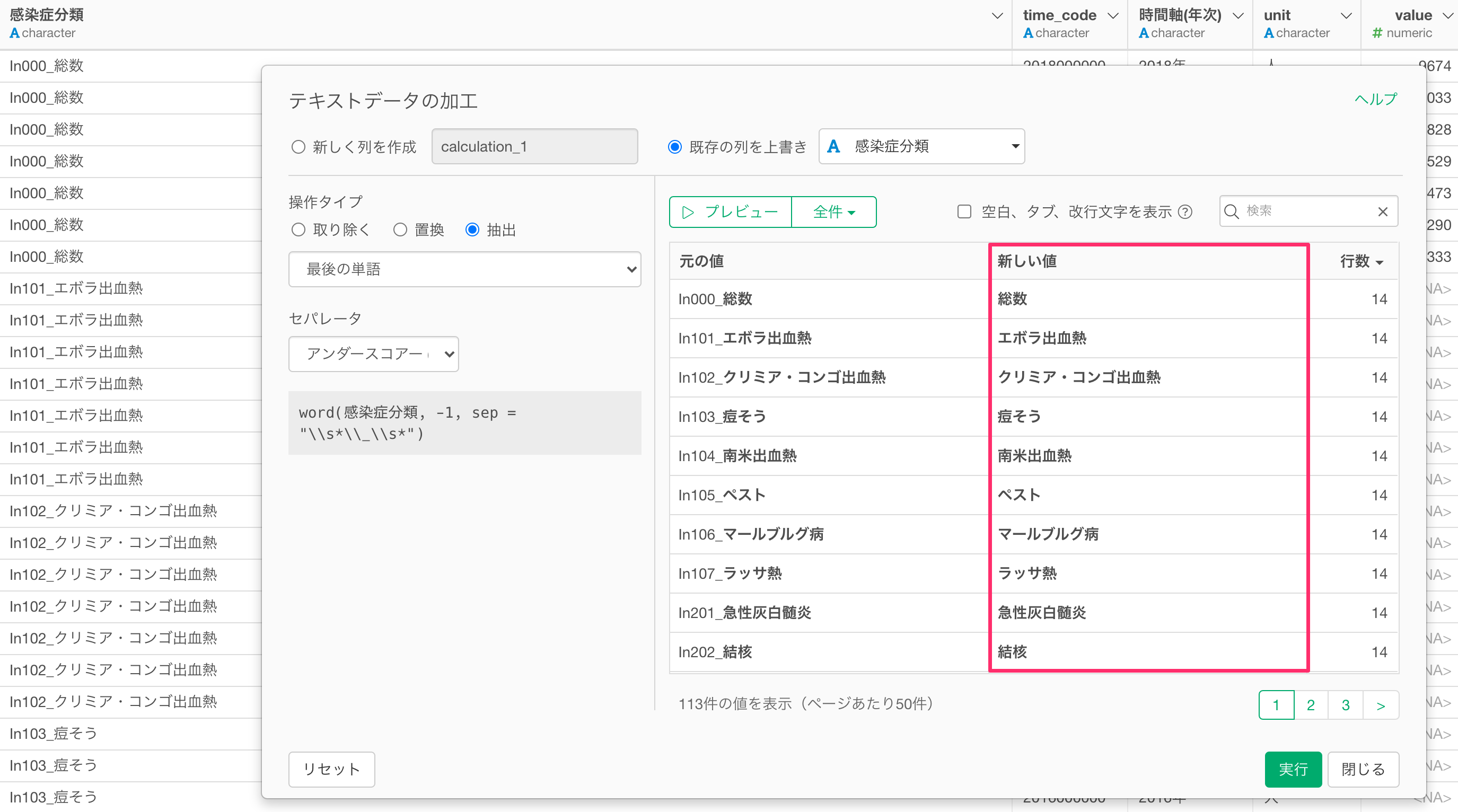
感染症分類の列には感染症のコードと感染症名が一つの列にまとめられているため、感染症名だけを抽出したいです。
値には「感染症コード_感染症名」となっているため、テキストデータの加工の最後の単語を抽出することで、感染症名を取り出すことができます。
感染症の分類の列から感染症名だけを抽出することができました。
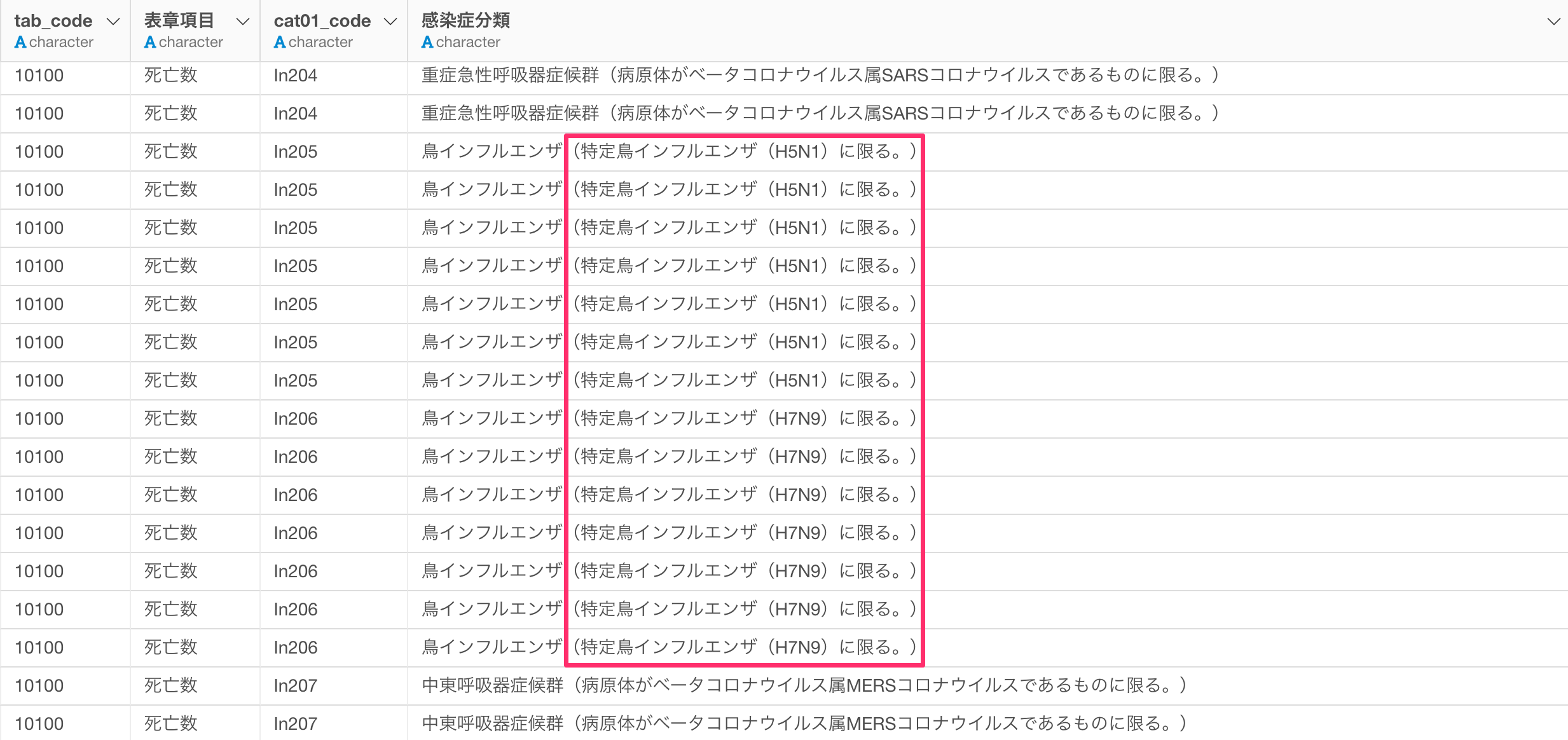
感染症分類の列にはまだ問題があり、鳥インフルエンザの中にも「H5N1」と「H7N9」で分かれているため、どちらも鳥インフルエンザとしたいです。
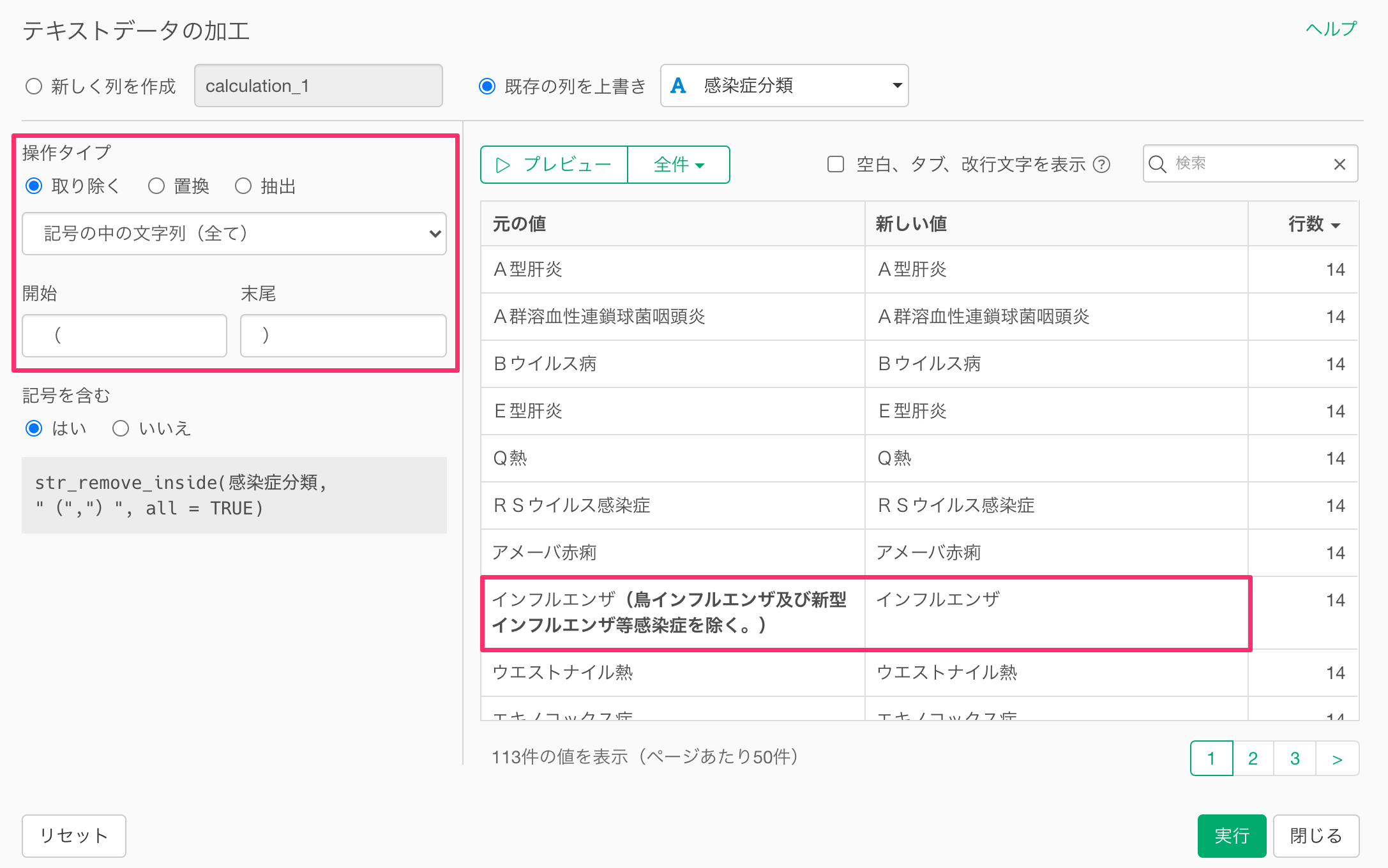
テキストデータの加工から記号の中の文字列を使うことで、括弧の中の文字列を取り除くことができます。
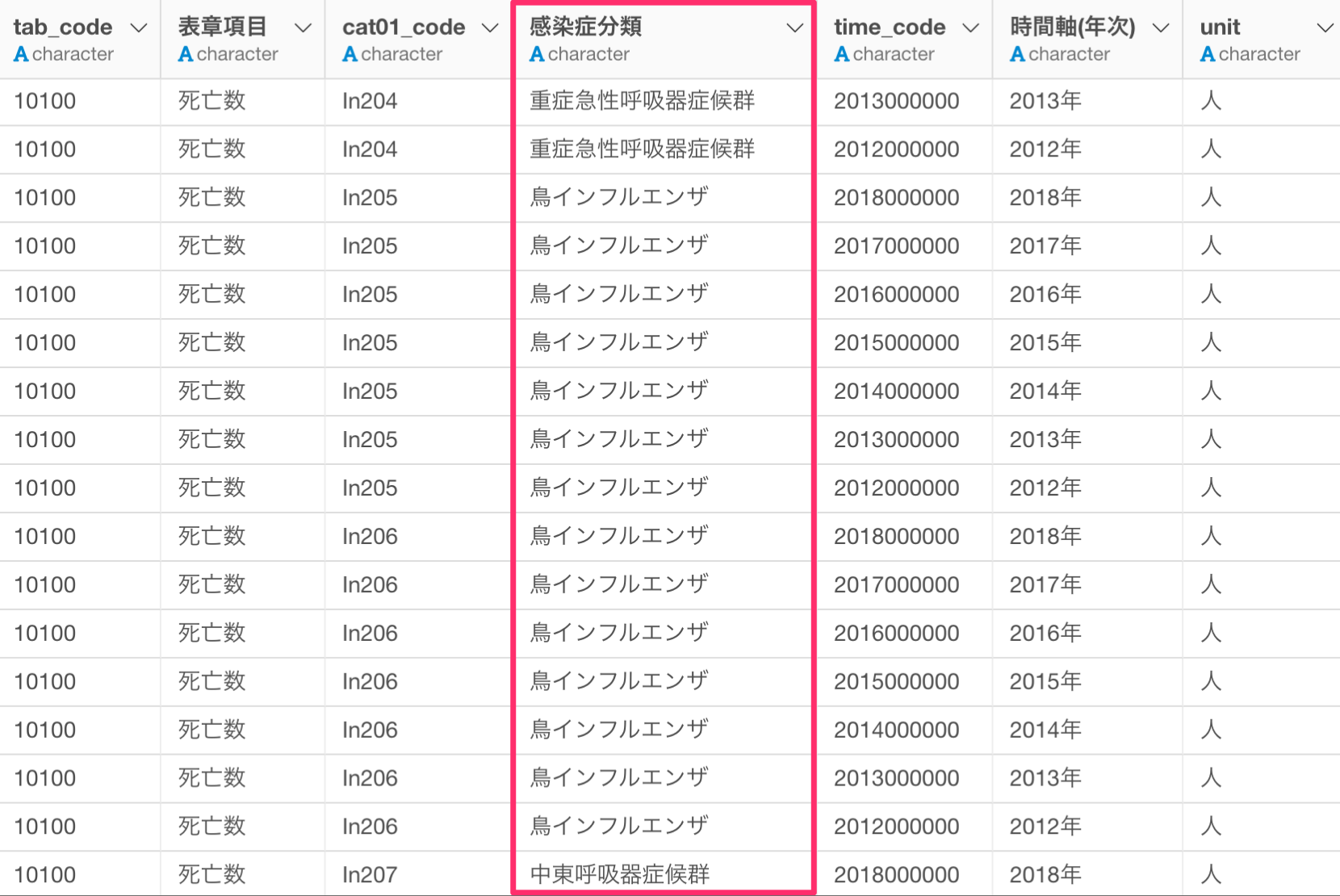
これにより感染症分類にあった括弧の中の文字を取り除き整えることができました。
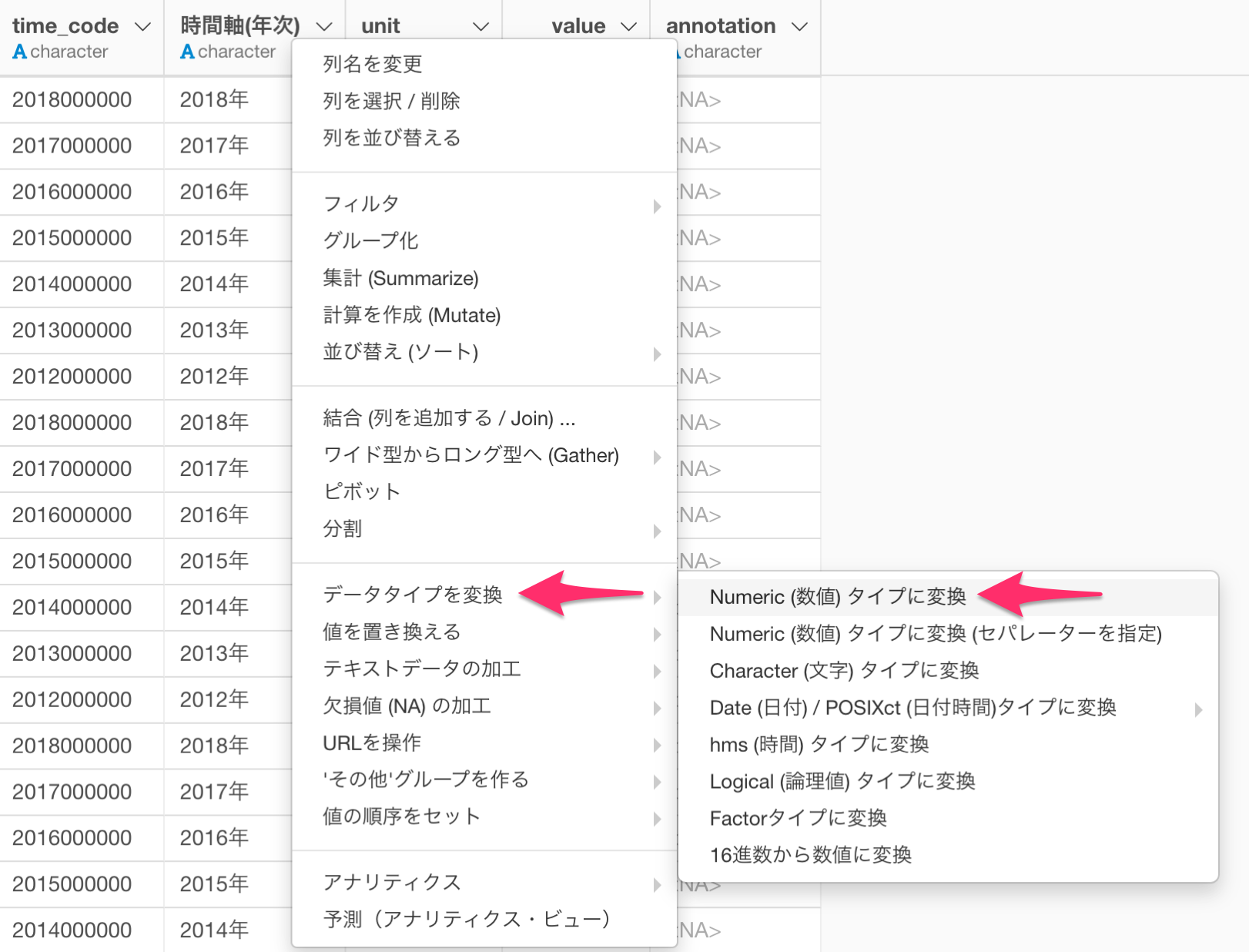
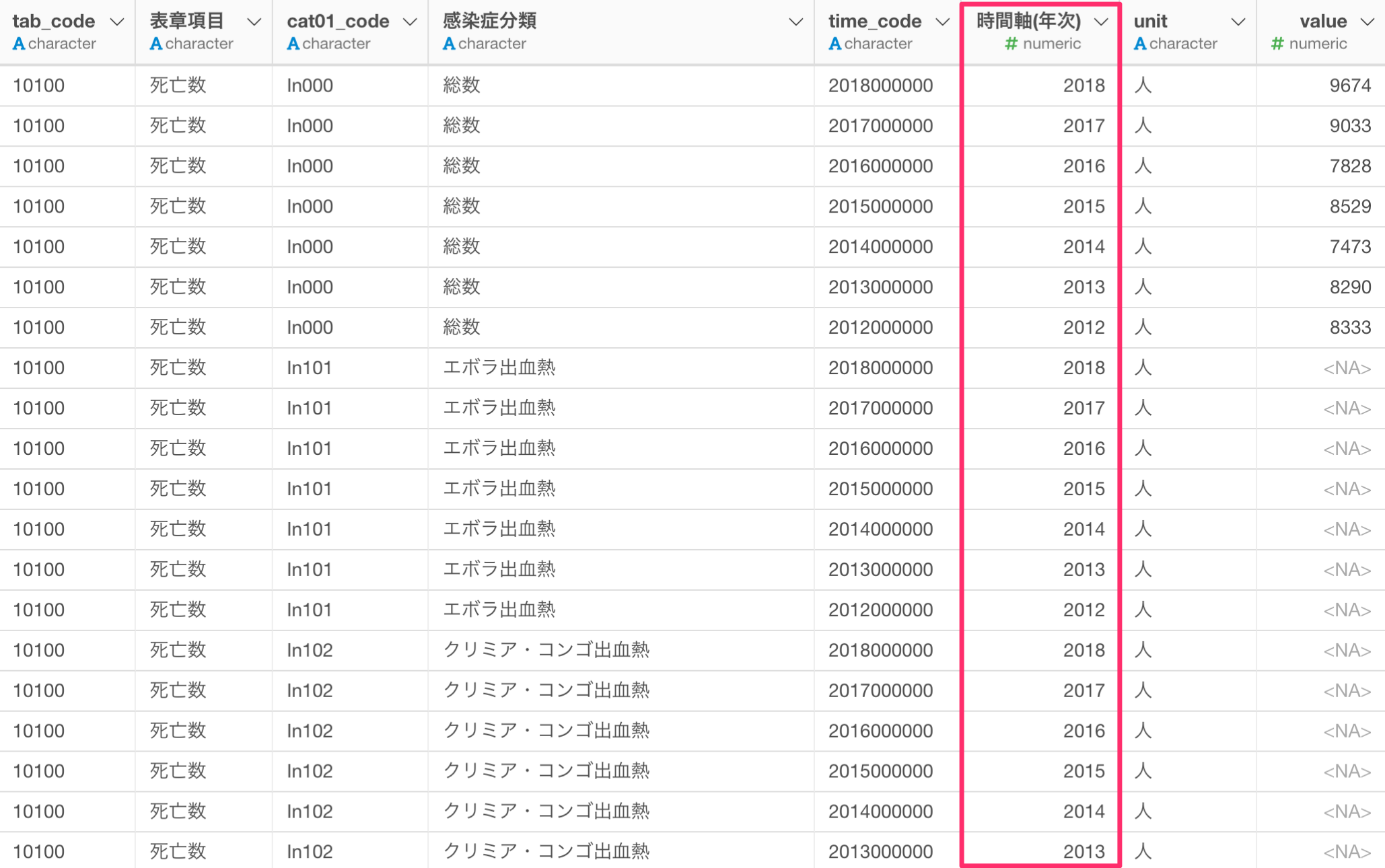
次に、時間軸の値には年がついているため、データタイプがカテゴリー型 (character)になっています。時間軸のように値に順序があるようなデータでは、データタイプがカテゴリー型になっていると可視化する際に年の並び順がおかしくなることがあります。
そのためデータタイプを数値型 (numeric)に変換する必要があります。Exploratoryでは、列ヘッダメニューから簡単にデータタイプを数値型に変換していくことができます。
時間軸の列のデータタイプを数値型に変換することができました。
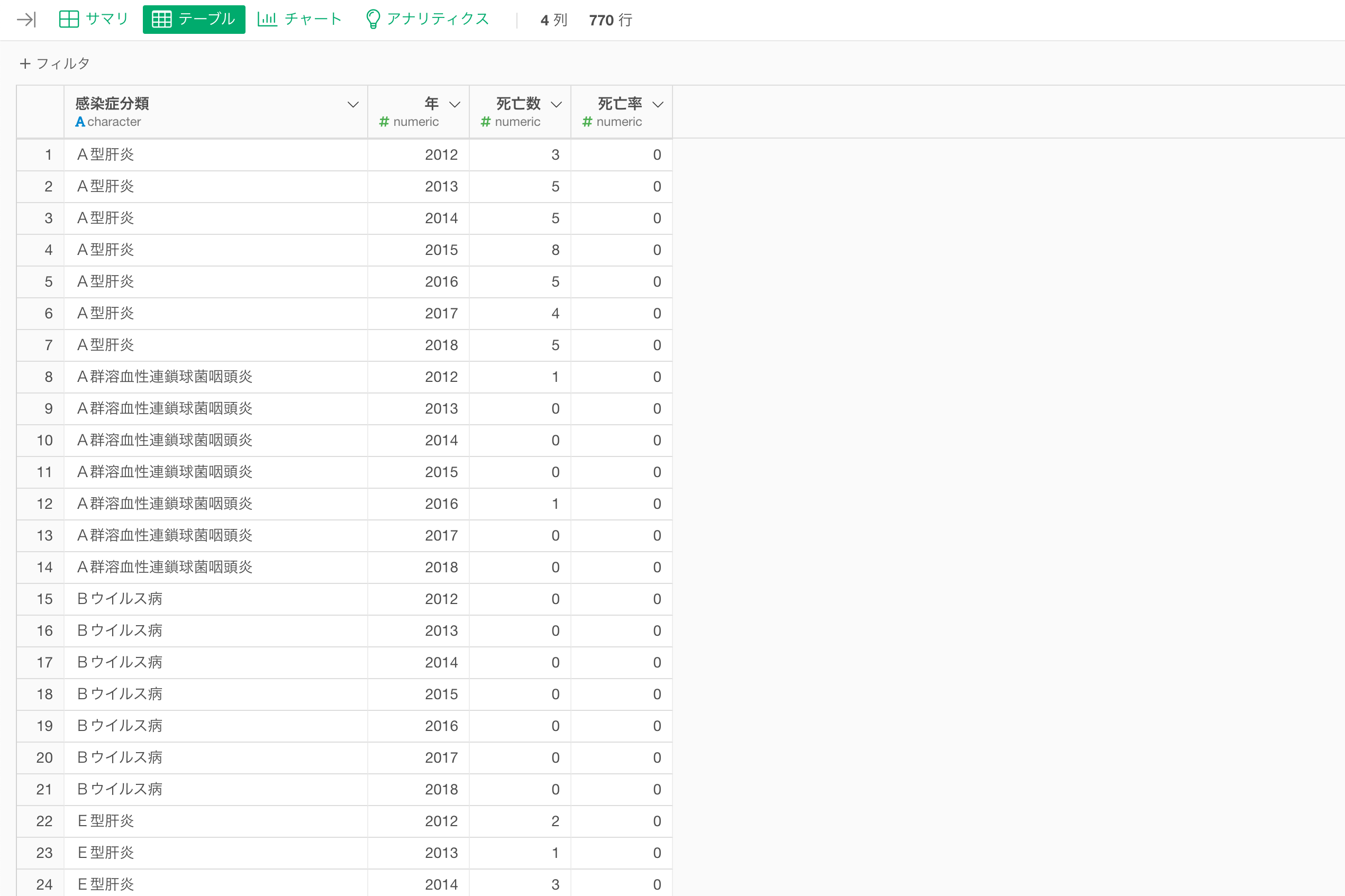
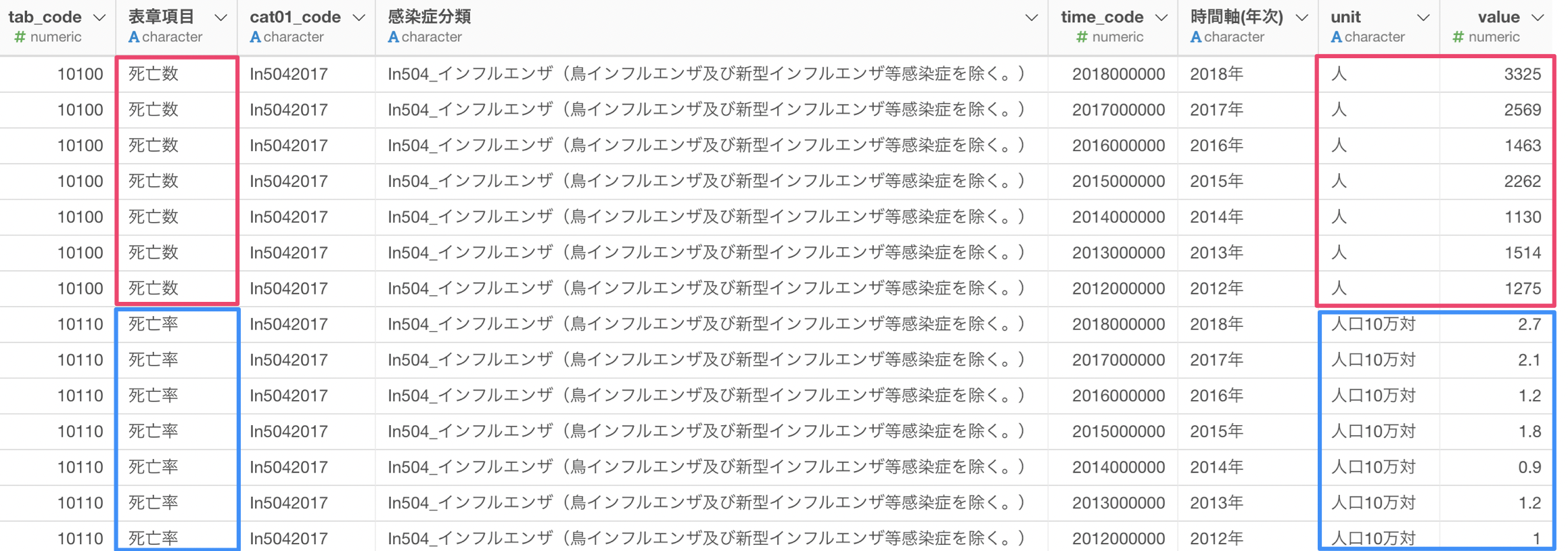
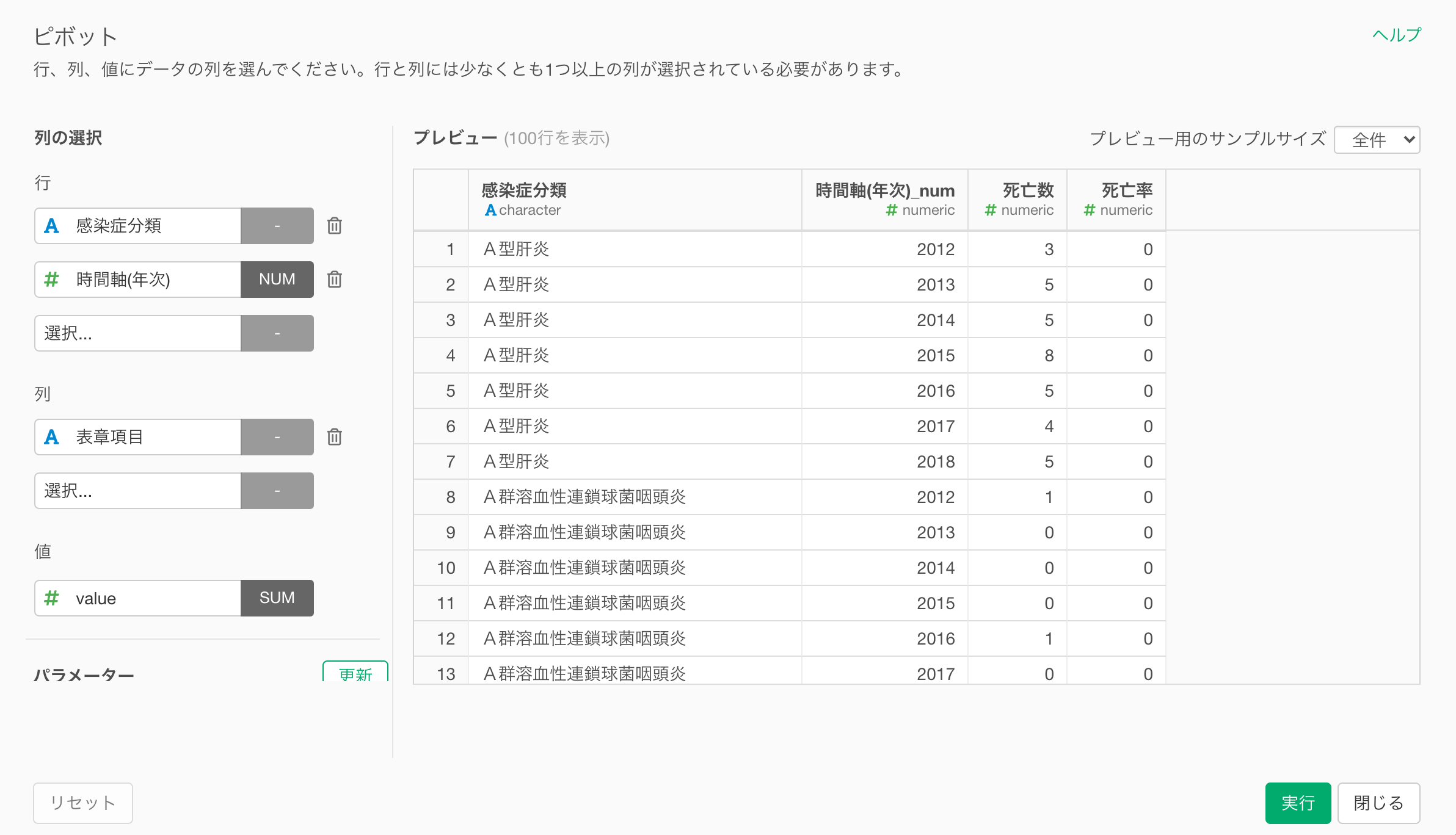
最後に、valueの列には死亡数と死亡率が一つの列にまとめられています。このように異なる値を持つ指標を一つの列にまとめてしまうと混乱を招き、死亡者数と死亡率をいっしょに可視化してしまう可能性もあります。そのため、死亡者数と死亡率の列を別々の列として分割していきます。
今回はピボットを使うことで、感染症の分類と時間軸ごとに行として割り当て、死亡数と死亡率の列を作成します。
これによりデータを可視化しやすい形に整えることができました。
死亡者数の推移を感染症ごとに可視化する
データラングリングをしてデータを可視化しやすい形に加工・整形することができたら、あとは可視化していきます!
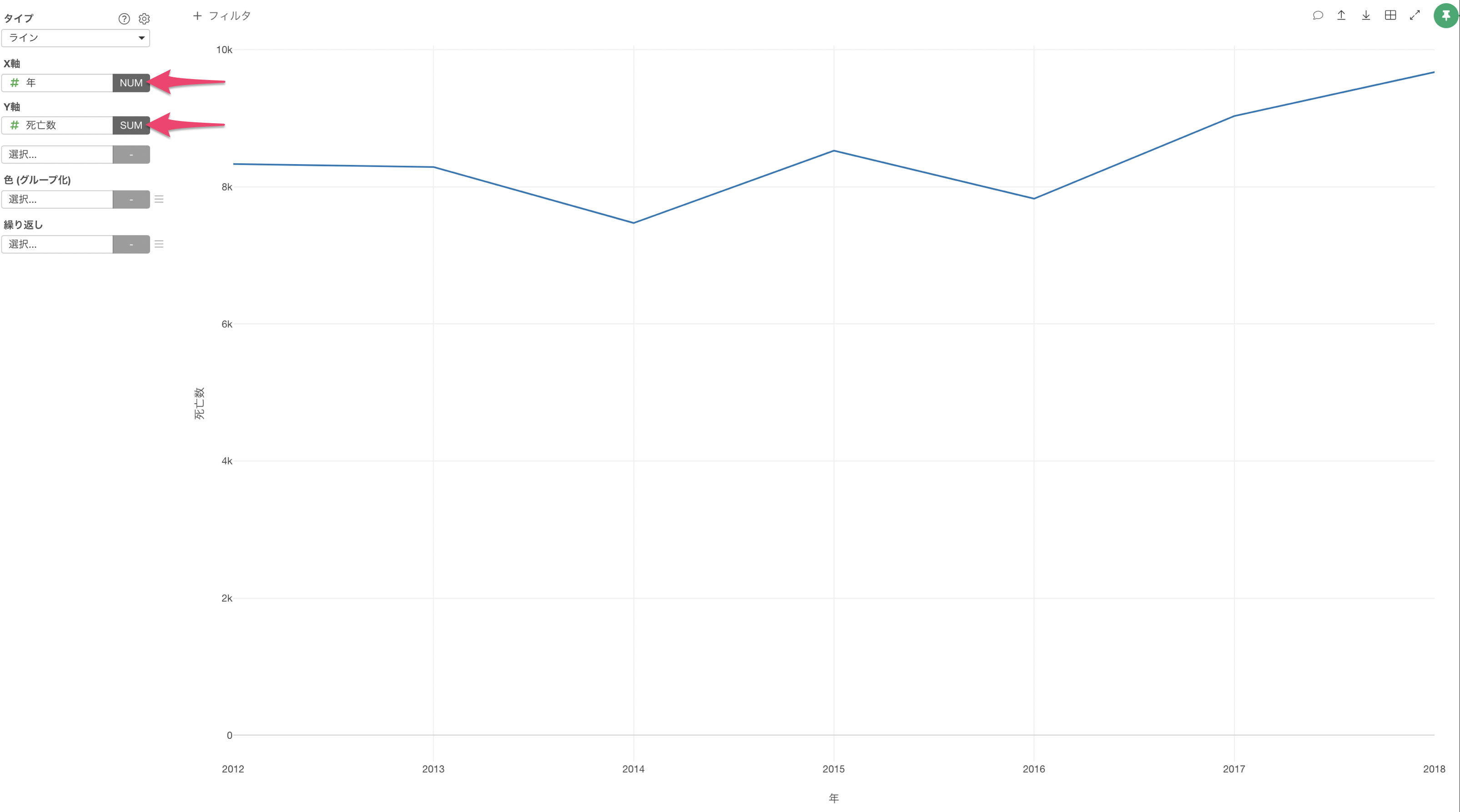
ラインチャートでX軸に年を、Y軸に死亡数を割り当てます。
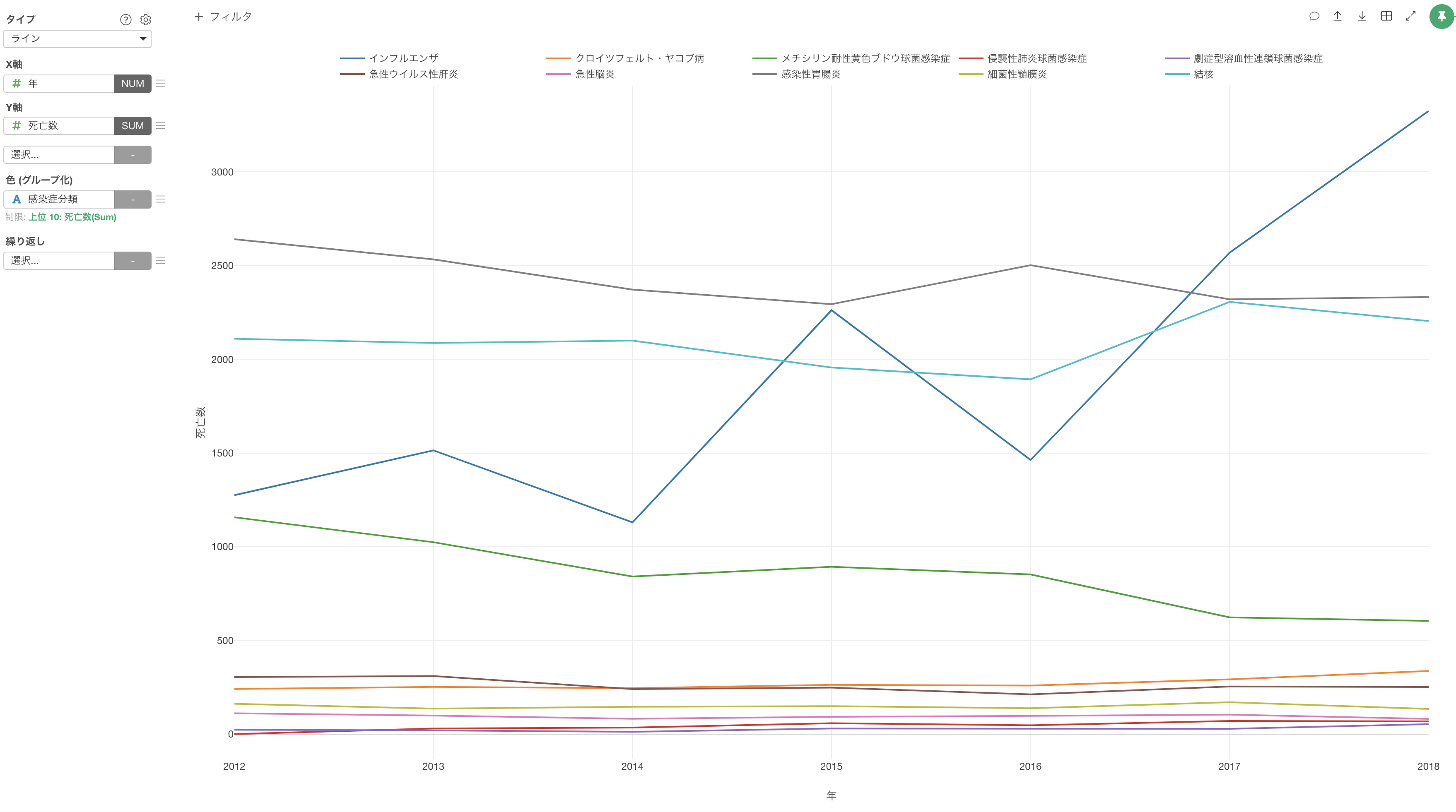
色に感染症分類を割り当てることで、感染症の分類ごとに年ごとの死亡者数の推移を可視化できますが、感染症の分類は110種類ほどあるため、すべてをラインで可視化してしまうとチャートが見づらくなってしまいます。
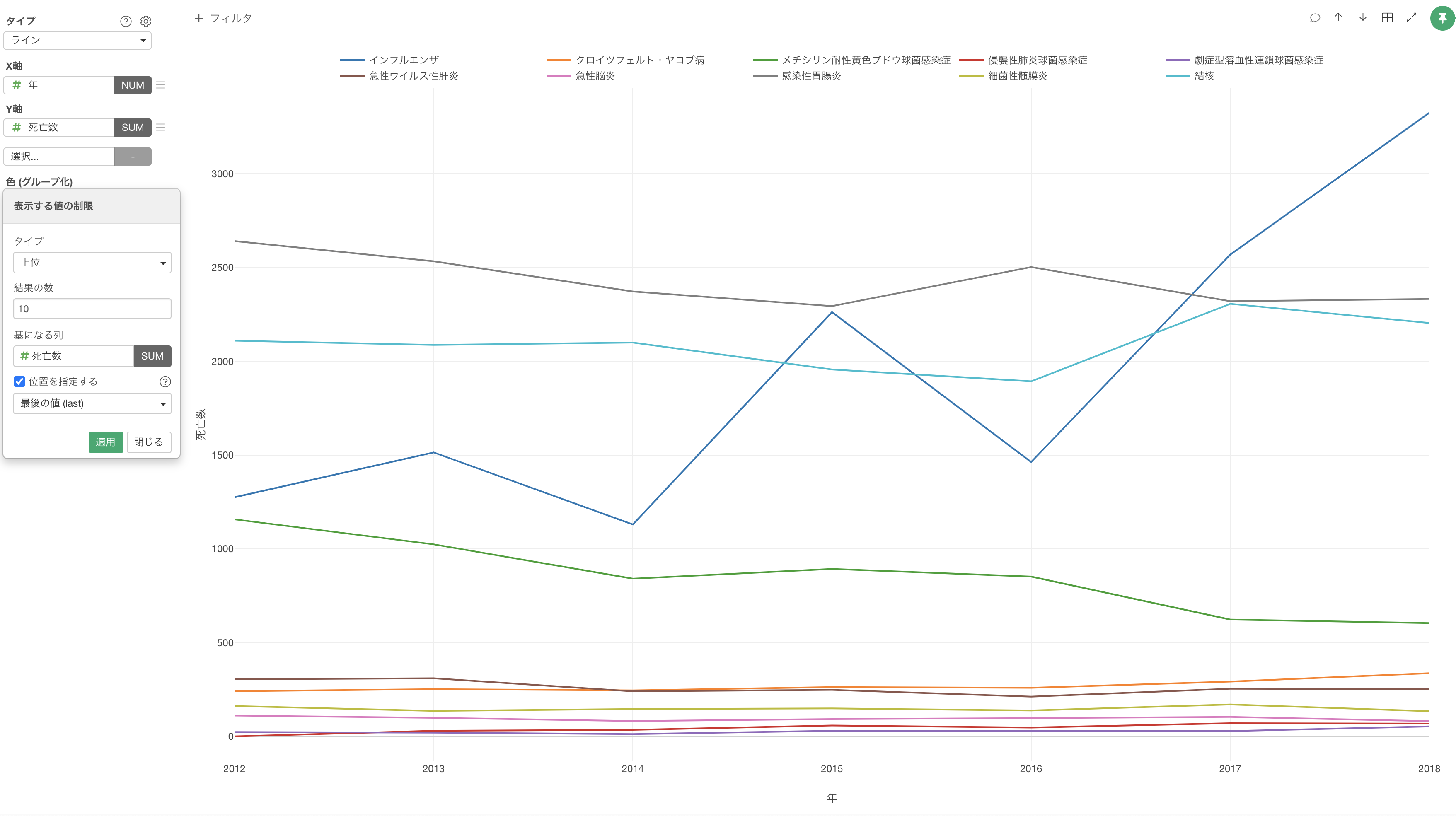
そこで、最後の年(2018年)の死亡者数が多い上位10の感染症のみを可視化するために、表示する値の制限という機能を使います。
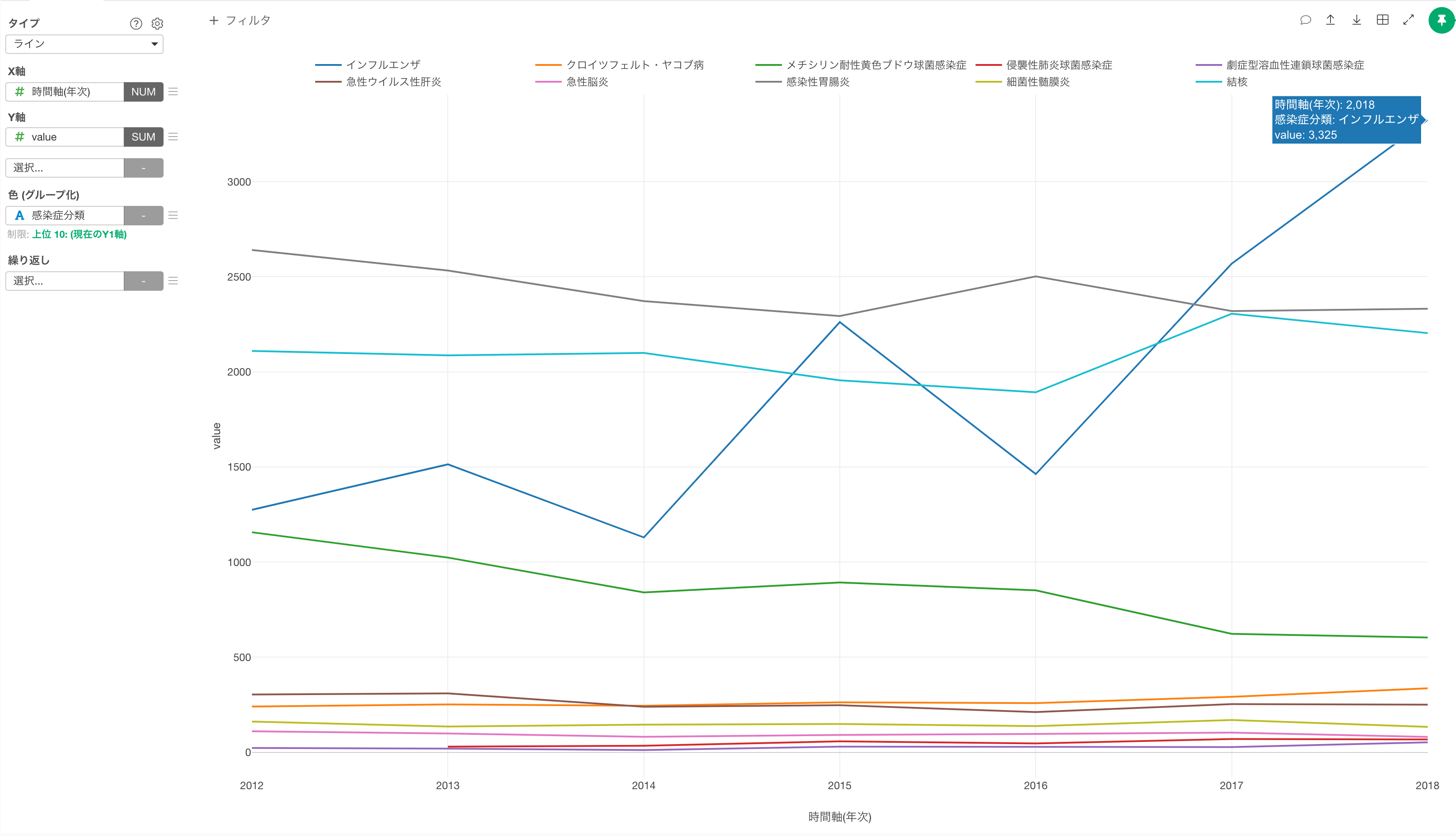
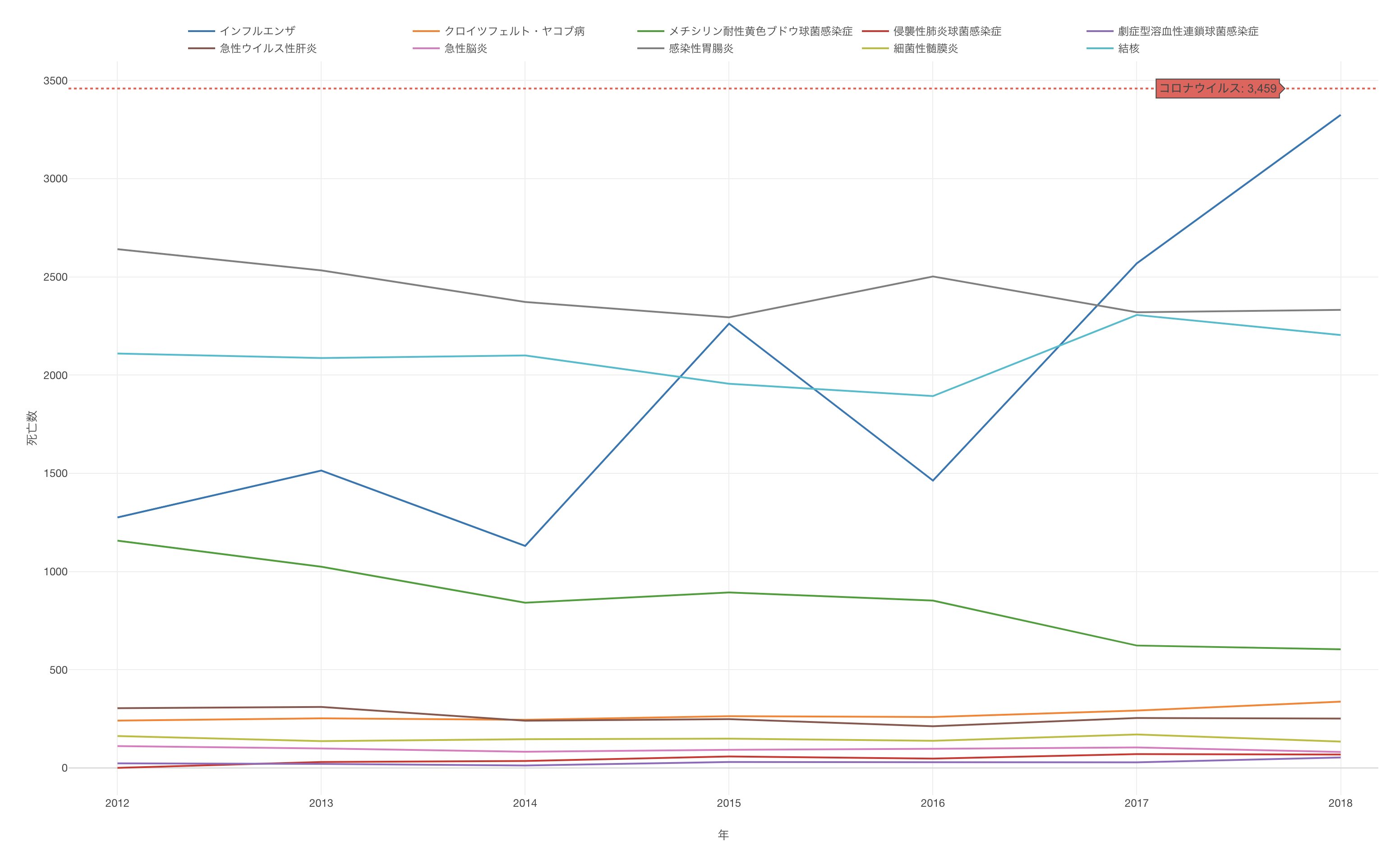
これにより、死亡者数の多い上位10の感染症の推移を可視化することができました。
インフルエンザによる死亡者数は時期にもよって変動するようですが、それでも毎年1000人以上は報告されており、2018年には死亡者数が3,300人と多いことが確認できます。他にも、胃腸炎と結核での死亡者数が毎年2,000人程いるということは驚きでした。
日本のコロナウイルスのデータと比較してみます。
データは厚生労働省のこちらから取得したデータになります。私たちの方でもデータを扱いやすい形で加工して公開しているので、興味がある方はこちらからデータをダウンロードしてみてください。
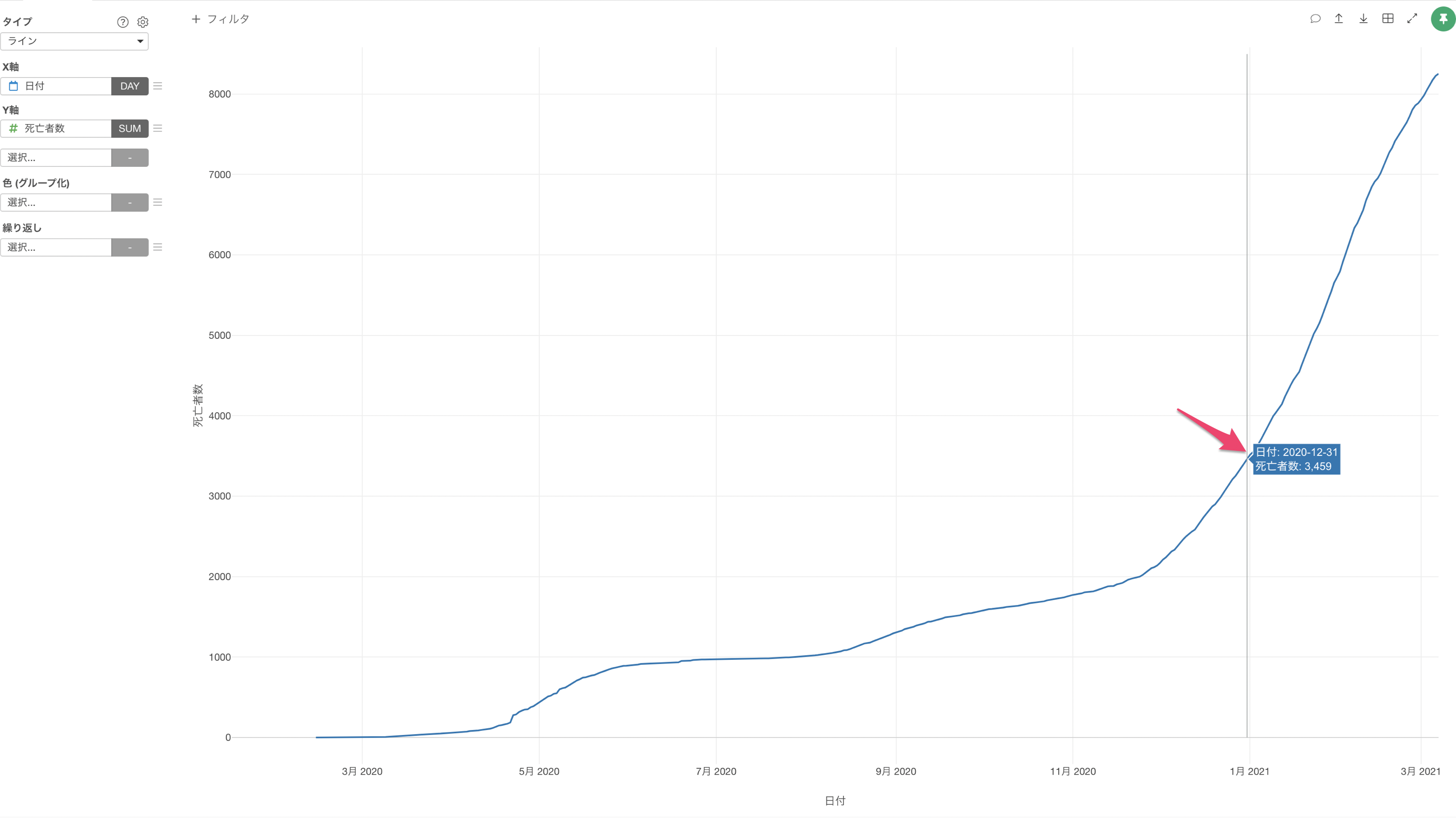
コロナウイルスのデータでは、何故か年が変わっても死亡者数は繰り越されて報告されることがあるため注意が必要です。2020年でみた時には、死亡者数は3,459人になります。
先程の感染症ごとの死亡者数の推移に、2020年のコロナウイルスの死亡者数を線で引いてみると2018年度のインフルエンザと100人ほどしか違いがないようです。
余談ではありますが、厚生労働省はインフルエンザが流行するシーズンに毎週、インフルエンザだと診断された人の数をレポートしていますが、今シーズンはこれまでに無いくらい異常な値がレポートされています。
通常であれば、1週間あたり数万人がインフルエンザにかかったと報告され、ピーク時には10万人を超えますが、今回は100人を超える報告はありませんでした。
今シーズンのインフルエンザの報告者数に関しては、こちらをご覧ください。
「コロナウイルスが流行したからインフルエンザが無くなった」ということはあり得ないので、推測ではありますが、本来はインフルエンザだったにもかかわらずコロナウイルスの陽性者だと判定され、そしてコロナウイルスで死亡してしまったと判定されている可能性があるかもしれません。
まとめ
今回は、政府の統計データの窓口であるe-Statから「感染症による死亡者数」のデータを、estatapiというRパッケージを使って簡単にとってきて、さらに最後に感染症ごとに死亡者数の推移の可視化まで行ってみました。
最近はe-Statのような政府系のデータサービスだけでなく、多くの企業もたくさんデータを「オープンデータ」として公開しています。さらに、estaapiのようなパッケージのおかげでそういったデータを取得するのも簡単になり、さらにその取得自体も自動化しやすくなりました。
しかし、こうした時代の一番の問題はそういったデータを取得することではなく、きれいにしたり加工したりすることで、使えるようにすることです。
一度使えるように形を整えてあげれば、後は可視化することでデータから直接世の中のことが見えるようになるのですが、残念ながらこの「データの加工」の部分ができないばかりに、多くの人たちがデータから役に立つ知識を得ることができないという状況を多く目にします。
これが「データの分析の80%の時間はデータの加工に費やされる」と言われる所以です。
そこで、私達の方でも、もっと多くの人たちがデータを自由自在に操り、効率よくデータを加工していけるようにということで、データラングリングのトレーニングをやっていたりします。
もし興味のある方は以下にリンクがある詳細ページを見てみて下さい。
それでは!
データラングリング・トレーニング、5月開催決定!
データラングリング(データの加工)の手法を1から体系的に、そして効果的に身に着けていただくために、データラングリング・トレーニングを提供しています。
データを自由自在に操れることで、実は思った以上に役に立つデータが身の回りにあるということに気づかれるはずです。そして思った以上にデータを使って答えることのできる質問がこの世の中には多くあるということにも気づいていただけると思っております。
ぜひこの機会に参加をご検討ください!
開催要項
- 日時 : 2021年 5月13日(木),14日(金) 9:00-17:00
- 会場: オンライン (参加者には事前にZoomのURLが送付されます。)
詳細はこちらのページにあります。