微生物解析パイプライン「QIIME2」を使った解析で、公式のチュートリアルでは、fastqファイルから門や属レベルのカウントデータは得ることができるが、OTUのカウントデータの取得方法が載っていない。
しかし近年はOTUレベルでの解析も盛んに行われており、必要だと思うので、調べてみた。
【利用ソフト(OS)】
VirtualBox
QIIME2 2019.10 (VirtualBox用のもの) 詳しくは→Installing QIIME2
(Dockerが流行ってるので今後はVirtalBoxからDockerにシフトする可能性はある。。。)
サンプルデータの取得
前提条件として、公式のチュートリアルに従って、table.qzaは持っているものとします。
サンプルデータは以下のリンクから取得してください。
download table.qza
OTUデータのエクスポート
.biomという形式のファイルに出力します。"feature-table.biom"というファイルが同じディレクトリに生成されます。
qiime tools export --input-path table.qza --output-path ./
テキストファイルへの変換
biomファイルのままでは普通の解析(OTUの名前とカウントが載ったテーブルを用いた解析)ができないので、biomをtsvファイル(.txtのようなテキストファイル)に変換
biom convert -i feature-table.biom -o feature-table.tsv --to-tsv
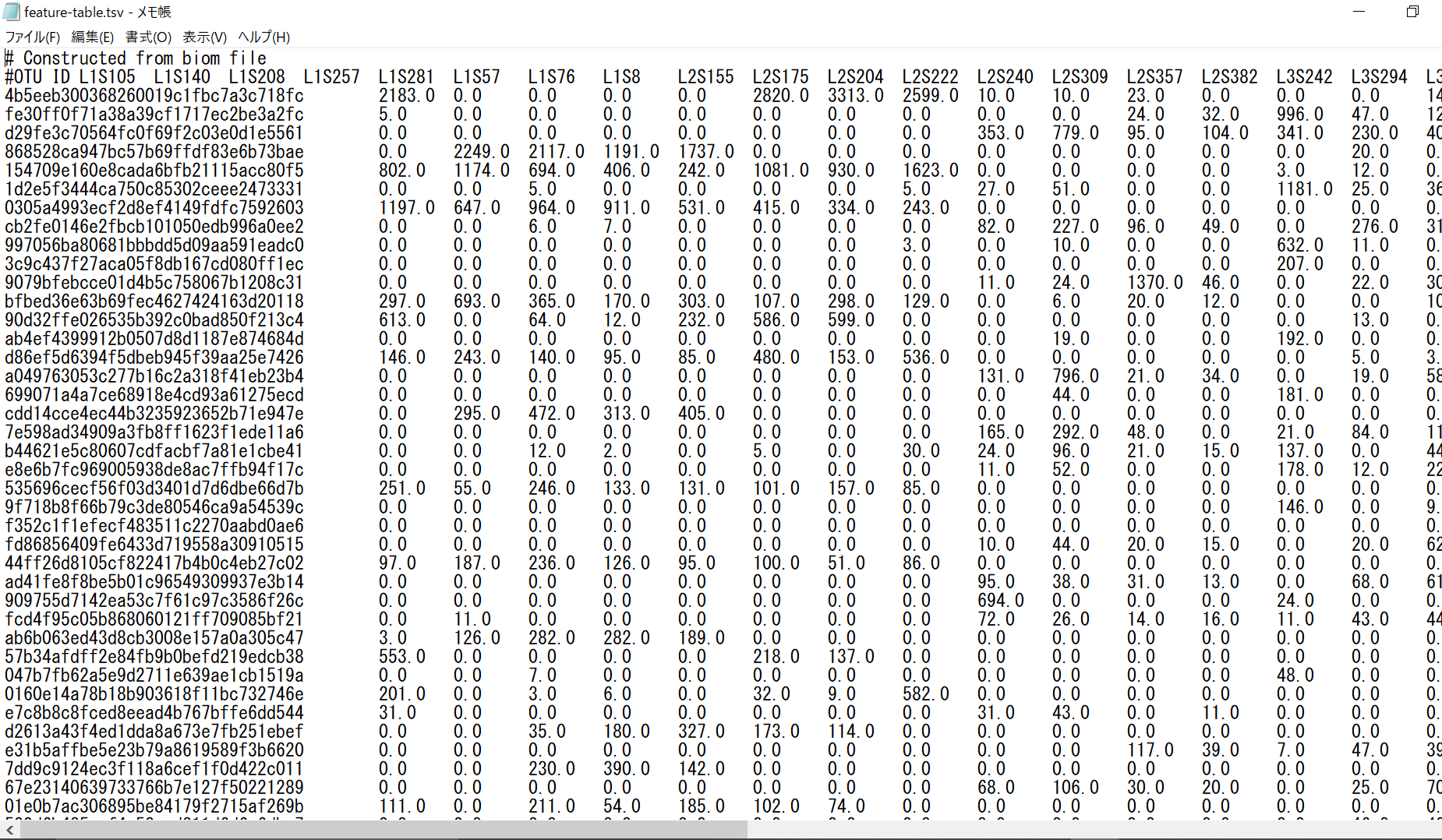
これで、以下のようなテキストファイルが得られます。
1列目がOTU名(ID)、2行目がサンプル名ですね。
データフレーム処理をするのに1行目は邪魔だと思うので、ここは手動で消しちゃってもいいと思います。
今後はこれを用いた解析のやり方をどんどん上げていきます。