0. はじめに
kaggleのPANDAという医療画像コンペで銅メダル~~(85 place, top 9%)~~を取ったのでその参加記を投稿します。
※7/25追記:final standingで(79 place, top8%)に変わりました
このコンペに参加するに当たって、メモをレポート形式で書いていたので、長ったらしいですがその辺を書いていきます。
今からPANDAコンペ復習する!という人には結構有意義な内容だと思いますが、そうでない人には興味のない部分も多くあると思いますので、そういうところは飛ばして頂いて構いません。
この記事ではある程度機械学習, Kaggleについての知識を前提としていますが、ニュアンスだけでも読めますので軽く読み流して頂けると幸いです。
また、本格的に画像コンペに取り組んだのは今回が初めてなので、手法的な部分で参考になるようなものは無いかと思われます。予めご了承ください。
0-1. ざっくり結論
・自分で考えた手法は何一つ精度の向上に寄与しなかった
・故に、典型的な手法を、幅広く試していくという検証方法が重要であると感じた
・画像コンペでのマシンパワーによる圧倒的な力量差を改めて感じた
・最終的なモデルはEfficientNet(b1)を13epochだけ学習させたもの単体で、まだまだ改善の余地が見られた
・完全にメダル圏外だと思われたが、140位もshake upしていて超ビビった
・Pytorchを用いてコンペに参加したのは初めてだったが、一通り使用方法を理解できた(今まではkeras)
・画像コンペの基本的な取り組み方をある程度理解できた(はず)
・その他、手法的な部分の理解度が大幅に上昇した
・メダル取れてめっちゃ嬉しかった
1.コンペについて
1-1.コンペ概要
PANDAコンペとは、Kaggleで行われたProstate cANcer graDe Assessment(前立腺がんステージ評価) Challangeの略で、前立腺から採取した組織を顕微鏡を用いて観察した時に、その細胞の形や大きさによって5つのグレードに分類したものを予測するという画像コンペです。この分類の仕方をグリソン分類(Gleason Score)といいます。グリソン分類は分類の手法を指し、グリソン分類によって算出されたグレードのことをISUPグレードといいます。(算出法については後述します)
よって、このコンペのタスクをまとめると、与えられた前立腺組織の画像に対して、その画像のグリソン分類を機械学習を用いて行い、ISUPグレードを提出することで、正解ラベルとの誤差を最小化すること、になります。
1-2.評価指標
1-1で正解ラベルとの誤差を最小化、と言いましたが、実は5段階の分類の中でも、誤差が少ないほど良いという仕組みになっています。
これを実現する評価指標がquadratic weighted kappaです。(cohen's kappaとも呼ばれるらしく、pytorchにはこちらの名称で実装されています)
この評価指標は、値こそ離散的であるけれども、その関係は連続的であるという場合に用いられます。
評価指標は簡単に、各予測に対して以下のようになっています。
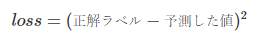
※これだとただのRMSEに見えますが、ざっっっっくりとこんな感じ。
例えば正解ラベルが4であったとして、
3と予測した時:(4-3)^2 = 1
2と予測した時:(4-2)^2 = 4
と、より離れた予測をしてしまった場合にペナルティが大きくなります。
よって、ある意味マルチクラス分類のタスクに対して、「間違っても良いので、できるだけその間違いは小さいようにしろ」というタスクであるとも言えます。
1-3.データセット
データセットは384GBと膨大でした。それもそのはず。1画像のピクセルサイズが(30000*30000)など超でかい。
・train_images
学習用画像が入っている。10616枚。tiff形式。ピクセルサイズは統一されておらずとにかくでかい。3チャンネル(RGB画像)。ヘッダーにISUPグレード,グリソンスコアなどの情報も入っている。
・train_label_masks
学習用画像に対する、マスク画像が入っている。具体的に、背景、正常組織、グリソンスコア3~5などの情報がマスクとして与えられる。
・train.csv
画像idに対する、その画像を解析した研究所、ISUPグレード、グリソンスコアの3つの情報が入っている
研究所:"karolinska"と"radboud"の2つが存在する。データ比はほぼ半々
ISUPグレード:0~5の6種類。0は前立腺がんが認められない場合に用いられる
グリソンスコア:0+0~5+5の組み合わせと、0+0と同義の"negative"の11種類のデータが存在する
その他、test.csv,sample_submission.csvがある。(どちらも提出時のデータフォーマットが正しいか確認するもの)
2.方針
このコンペを走り切るに当たって、最初に以下の方針を立てました。
1.モデルはEfficient Net一本に絞る
2.モデルの分割、学習データの分割の工夫を行う
3.Data Augmentationを工夫して行う
結論から言うと、最終的には1,部分的に3しか達成できませんでした。
というのも、画像コンペにおいて何が大事なのかを当時理解しておらず、とりあえず方針は立てておこうというということでやる気だけで適当に決めたからです。
それぞれ順を追って説明します。
2-1 1.モデルはEfficient Net一本に絞る
画像コンペでは様々な手法を試すのが良いとされているのですが、参加し始めたのがコンペ終了16日前と相当終盤だったので、ある程度モデルと手法を決め打ってやることにしました。
Efficient Netに決め打った理由は、学習時間に対する精度(GFLOPS)が優秀であることと、前に参加した画像コンペで上位日本勢の方が用いていたモデルがEfficient Netだったからです。(あとSE-ResNextだったかな?)Fine-Tuningとは学習済みモデルを用いて再学習させることを指しますが、これも短時間で高精度を出すことができます。
(EfficientNetについては3.理論に説明があります)
2-2 2.モデルの分割、学習データの分割の工夫を行う
ここでいうモデルの分割とは、k-fold法を用いたクロスバリデーションを指すのですが、これは単純に時間的な問題で無理でした。(1つのモデルの学習に10時間以上かかり、最終的な方針が決まった時にはそんな時間はありませんでした)
学習データの分割は、テーブルデータのコンペではしばしば有効です。今回の場合、画像データを提供している研究所ごとに学習するというのが一番良さそうに感じますが、資料18のDiscussionに、「研究所ごとの学習は精度の向上に寄与しなかった」と、既に検証結果を示してくれている方がいました。これは、学習用の画像枚数が約10000枚と母数が少ない中で、それを半分にして学習させて研究所ごとに特化させるメリットよりも、できるだけ母数の多い状態でモデルに多様性を求めるメリットのほうが勝っていたということが考えられます。
※但し、コンペ終了後の上位解法を見たところ、施設ごとにquadratic weighted kappaのしきい値を替えるというのは有効なアプローチだったようです。
よって、この方針は結果的に実現しませんでした。
2-3 3.Data Augmentationを工夫して行う
※(Data Augmentationについては3.理論に説明があります。)
Data Augmentationは行いましたが、工夫する事はありませんでした。結局、Rotate,Flip,Transposeのシンプルなものを使用しました。(4-6で独自のAugmentationを行いましたが、精度の向上には寄与しませんでした。でも面白いと思うので見てみてください)
今回のコンペでは、画像の分類のポイントとなるのが物体の外観や色ではなく、細胞の"顔つき"という少し特殊なものなので、下手にボカしやブラーなどを入れるとかえって精度の低下を招きます。これは、通常の画像に比べて、細胞の画像は高周波数成分の重要度が高いからであると推測できます。ボカしを入れたりすると、高周波数成分の情報は一気に消し飛ぶので、学習に必要な情報が無くなるということになります。
実際にコンペ終了後の上位解法でも、これ以外のData Augmentaitonを行ったものは私が確認した限りではありませんでした。(そもそも、他コンペに比べてData Augmentationについて述べているものが少ないので、回転・反転程度で留めるのが前提なのかも)
3.理論
3-1,3-2では、臨床的な理論について述べます。3-3~3-5では、機械学習の手法に関することについて述べます。
3-1.前立腺癌
前立腺癌は尿道から離れた辺縁部に発生する事が多い為、排尿障害などの臨床症状があまり現れません。一方で、前立腺癌は血行を伝って骨(特に脊椎、骨盤)などに転移しやすく、転移先の症状で初めて見つかる場合も少なくない癌です。その他、浸潤的に精嚢・膀胱にも転移します。
前立腺癌の診断は、
・直腸から触診による診断
・血液検査(腫瘍マーカーPSA値の測定)
・超音波検査
・生検(前立腺の組織を採取して顕微鏡で細胞の形を診断)
があります。最終的な確定診断は生検によって行い、そのままステージの判別を行います。(そのため、特に生検の画像に対する機械学習は重要であると言えます)
前立腺がんの治療は、基本的には手術・放射線治療によって行います。(遠隔転移がある場合にはその限りではありません。)特に前立腺がんにおける放射線治療の成績は良く、密封小線源治療や、IMRT(強度変調放射線治療)によって治療を行います。前立腺の真後ろには放射線に弱い直腸が存在するので、気を使ってやる必要があったりします。
3-2.グリソンスコア
グリソンスコアは生検で採取した前立腺の細胞の"顔つき"をその悪性度に分けて5段階に評価するものです、が、実際には良性度の高いものは0としてしまい、3以上の数字が使用されることが多いです。
グリソンスコアは、最も面積を多く占める細胞のグレードと、その次に面積を多く占める細胞のグレードの2つを用います。これを、3+4などと表します。
日本ではこのまま二数の和をもって診断に用いますが、海外ではここから更にISUPグレードというものに変換するそうです。変換表は図3-1に示したとおりになります。
今回のコンペは、このISUPグレードを予測するのがタスクとなります。(ISUPをそのまま予測するか、グリソンスコアを用いて間接的に予測すべきか…など、色々なアプローチがありますね)
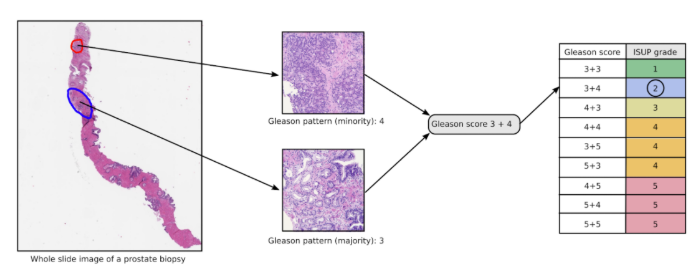
図3-1:グリソンスコアとISUPグレード(資料7より引用)
グリソン分類図(分類の目安となる図)が、下記論文のFigre12にあります。その他、ネットで「グリソン分類図」と調べても同じようなものが出てきます。
URL: https://pubmed.ncbi.nlm.nih.gov/16096414/
直接リンク: http://nefrouro.patologi.org/files/ISUP2005konsensusProst.pdf
図で見ると分かりそうですが、実際にはそんなに単純ではないため、素人目には何が何やらわかりません。
3-3.Fine-Tuning
Fine Tuningとは、既存の学習済みモデルを再学習させることで、全く重みを持たない状態からの学習よりも高精度に、かつ早く学習する事ができる手法のことです。入力層および出力層付近のみではなく、中間層も再学習させるという点で転移学習とは異なります。但し、CNNの特徴として入力層付近では画像の大まかな特徴を捉え、出力層付近で細かな特徴を捉えるという性質があるので、一般的に入力層付近の学習率は低く(もしくは学習せず)、出力層付近の学習率を高く設定します。
画像コンペでは基本的にはこの手法を用います。
3-4.EfficientNet
使用するモデルについてです。有名なCNNモデルとして、VGG16やVGG19、それを一気に出し抜いたResNet,そこから派生したDenseNetやResNextなどがありますが、2019年にSOTA(その時点で一番性能が良いもの)となったのがEfficientNetです。多くの新しいモデルはResNetから派生したものですが、EfficientNetもResNetから派生(スケールアップ)したモデルであると言えます。
特に以下の点で優れています
・パラメータ数が従来のモデルに対してかなり少ない(=計算時間の短縮)
・にもかかわらず、精度は非常に高い。(多くのデータセットに対してSOTAを更新した)
・転移学習モデルとして適している。
早い・強い・使いやすいが揃ったEfficient Netは、kaggleの画像コンペでもしばしば使用されるモデルです。
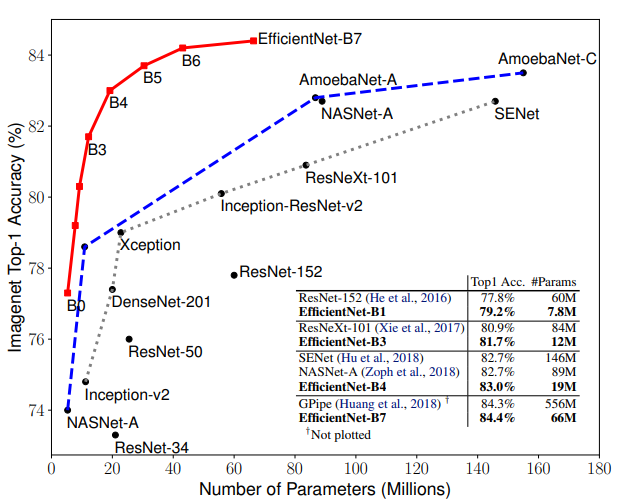
図3-2:EfficientNet(赤線)のパラメータ数に対する精度(資料5より引用)
3-5.Data Augmentation
Data Augmentation(データ拡張)とは、データの水増しをすることにより過学習の抑制及び精度の向上を目的とした、feature engineering(特徴量作成)の一種です。特に画像系のデータに対して有効です。
KerasのImage Data Generatorでできる簡単なData Augmentationには以下のようなものがあります。
水平・垂直に画像をシフトする
水平方向・垂直方向に画像を反転させる
回転させる (回転角度はランダムのケースもある)
明度を変える
ズームインする・ズームアウトする
画像の一部をくり抜く、削除する
背景色を変える
背景を置き換える
また、近年有効とされる特徴的な手法として、以下のようなものがあります。(図3-3)
ResNet-50と書いてあるのがベースラインとなるスコアです。
Mixupは複数の画像を重ね合わせる手法です。
Cutoutはデータの一部を欠落させる手法です。
CutMixは複数のデータを矩形領域で合成する手法です。正解ラベルはその画像の面積によって分けます。すべての評価指標に対して精度が向上しているのが分かります。
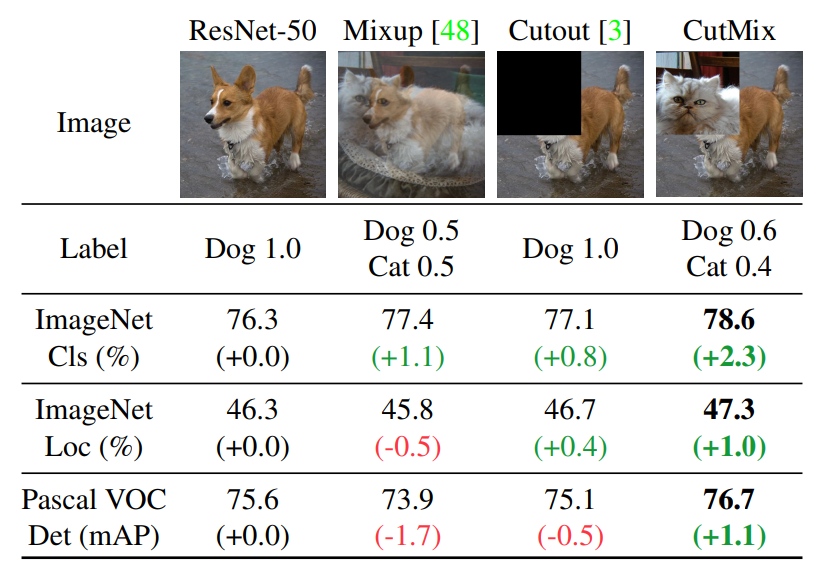
図3-3:Data Augmentation(Mixup,Cutout,CutMix)(資料6より引用)
但し、このような新しい手法はライブラリとして存在しない場合が多く、自作するか他の人のものを探す必要があります。
また複数正解ラベルが存在するという処理が面倒であることがデメリットです。
4.検証
4-1.EDA
まず最初に行ったことは、EDA(探索的データ分析)です。
データの特徴を知らないことには、モデルの工夫も何もできません。
多くは資料4のnotebookを見て真似たものですが、データの主要な特徴について駆け足で分析していきます。
EDAを行ったnotebook
↑に、より詳細に記載しています。(超お粗末ですが…)
まず正常組織と悪性度の高い組織について見比べました。
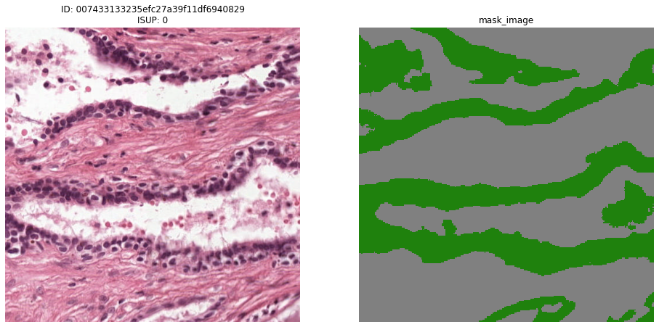
図4-1-1:正常組織とそのマスク画像(緑部分が診断に用いる領域)
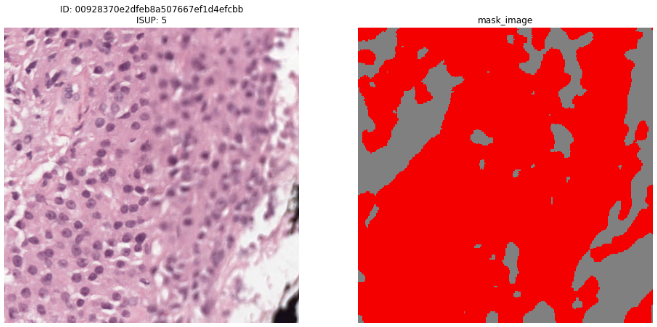
図4-1-2:悪性度の高い組織(ISUPグレード5)とそのマスク画像(赤い部分がグリソン分類5)
上に表示しているのは最大倍率の画像ですが、色々な視点で2画像を比較した時に、以下のような特徴が見えてきました。
正常細胞:
・細胞核が密集しているのは組織表面である。
・上皮性組織において、比較的細胞核の配置が規則的である。
・細胞核の形・大きさが一定である。
・マスク画像を確認した時に、多くの場所は診断に関係ない非上皮性組織である。
悪性度の高い細胞:
・組織全体に細胞核が密集しており、画像を細胞核が確認できるぐらいの倍率で見た時に、明らかにISUPグレードの高い画像のほうが細胞核の数が多い。
・細胞核の配置がまばらである。
・細胞核の形や大きさがおかしい。
・マスク画像を確認した時に、ほとんどの部分を悪性の上皮性組織が占めている。
あと思ったのは、これは関係あるかわからないので上のリストには入れなかったのですが、正常な細胞は水っぽいのに対して、悪性の細胞は粘液性っぽいなと思いました。
集合体恐怖症の僕には結構辛い作業でした。
次に、テーブルデータ(csvファイル)から得られる情報について調べました。
データ提供施設(data_provider)は2つで、データ提供割合は以下のようになっていました。
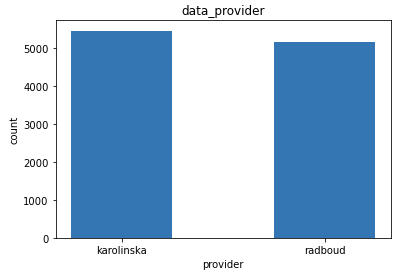
図4-1-3:データ提供割合(karolinska:51.39 % , radboud:48.61 %)
ほとんど1:1のデータ割合となっていることが分かりました。なお、このデータはテーブルデータの中で唯一学習に用いることが可能です。(他の2つは正解ラベルなので当たり前ですが)
ISUPグレードの分布は以下のようになりました。
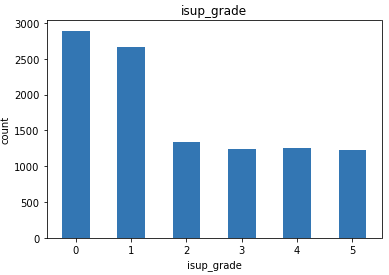
図4-1-4:ISUPグレードの割合
陰性を示す0と早期癌を示す1が多めの割合をとり、2~5がほとんど同じ割合であることが分かりました。(あまりにも割合が等しいので、そのように調整されているのかな?と思いました。)
その他、施設ごとのISUPグレードの割合が相当違うことが分かったりしました。(気になる人はnotebookを見てみてください)
これで、データの特徴や偏りが一通り分かりました。
4-2.データの加工
trainデータとして与えられる画像はサイズが大きく、またその中でも組織が写っている部分は画像の半分未満です。よって、効率的に学習を行うために関心領域(ROI)を設定し、画像の一部を抽出するという作業が必要になります。この作業は資料8からコードを丸パクリすることによって省略しました。(EDAによって色々アイデアが浮かんだだけに、ちょっと丸パクリするのはもったいない気もしましたが、一通り学習が終わってから時間があればやろう…と諦めました。)
概要は以下の図4-2-1のような感じです。
CNNにおけるROIは基本的に矩形領域で設定することから、単純にこれをやろうとすると画像で言う左下ー右上の端で画像を抽出することになります。しかし、今回のデータの場合にはそれでは無駄な背景部分が多く、効率的でありません。
そこで、この細胞の全体像の形には意味がないという特性を利用して、学習に適している領域のみをタイル状にピックアップします(図4-2-1右)
これを実装する関数にはタイル数と各タイルの画素値を設定できる引数が用意されているため、自由にカスタマイズすることができました。
なお、この加工方法で学習を行った他の方(この関数を作った方とは別)のnotebookで、LB0.87という7/6時点で銅メダル圏内のスコアを出している人がいました。
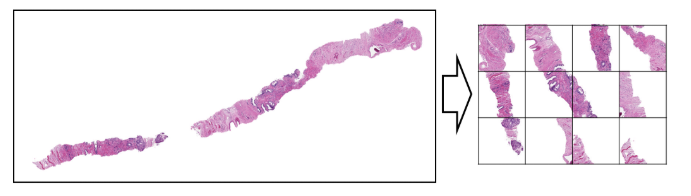
図4-2-1:tiling(資料8より引用)
4-3.ベースライン
ベースラインとして、以下のモデルを組みました。
name : panda-tachyon-baseline
model:Efficient Net b0 (fine tuning)
batch:2
epoch:10
criterion:CrossEntropy
optimizer:Adam
lr:0.01
data:1000
preprocess:tiling method (36,256,256)
train_test_split:single fold(t:v = 3:1)
data augmentation:none
Public LB score:0.19 (place 798)
ところが何故か非常に低いスコアしか出ず、lossも安定せず困ってたところ、他の人のカーネルを見た時にlearning rateが高すぎることが原因であることに気が付きました。(経験上lr=0.01で大きすぎるなんてことは無かったので、学習率なんて疑いもしませんでした…)
ということで、ちょっと改良して以下のモデルに組みました。
name : Panda-tachyon-v1
model : Efficient Net b0 (fine tuning)
batch : 2
epoch : 12
criterion : CrossEntropy
optimizer : Adam
scheduler : ReduceLROnPlateau
lr : 3e-3 ~ 1e-5
data : 2000
preprocess:tiling method (36,256,256)
train_test_split:single fold(t:v = 3:1)
data augmentation:Transpose,Flip (no TTA)
Public LB score:0.80 (place 567)
学習率に慎重で無ければいけないことが分かったので、この段階でスケジューラーを入れることにしました。同時に過学習抑制も早めに組み込んだほうが良さそうなので適当なData Augmentationを入れました。
これでおおよそ正しいスコアが得られるようになりました。
最後にひと押し、学習時間との兼ね合いを考慮して、最終的に以下のモデルに落ち着きました。
name : Panda-tachyon-v1-long
about : データサイズをちょっと変えてlrも微調整
model : Efficient Net (fine tuning)
batch : 4
epoch : 20
criterion : CrossEntropy
optimizer : Adam
lr : 1e-3 ~ 1e-6
scheduler : ReduceLROnPlateau
data : 3000
preprocess : tiling method (16,256,256)
train_test_split : single fold(t:v = 3:1)
data augmentation : transpose,flip (no TTA)
Public LB score: 0.81 (place574)
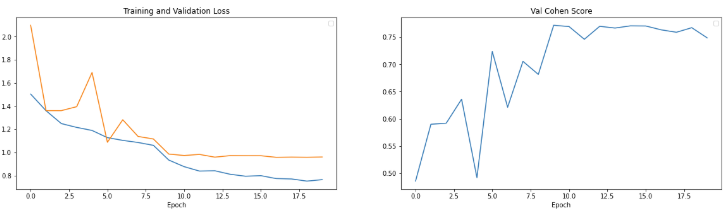
図4-3-1:learning curve (Panda-tachyon-v1-long)
4-4.過去のコンペからヒント探し
一通りちゃんとしたモデルを組み終えたところで、過去の似たようなコンペの上位ソリューションを読むことにしました。(スコアを上げるための常套手段だそうです)
医療系の画像コンペであり、評価指標がquadratic weighted kappaであるコンペとして、APTOS 2019 Blindness Detection(資料9)がありました。
このコンペは眼の画像から糖尿病性網膜症の有無・程度を求めるというコンペで、画像の性質が違えど今回のコンペとよく似ています。
ということで、このコンペの上位ソリューションを一読しました。(上位の方たちは難しい手法を試している場合が多く、よくわからない単語も出てきたため苦労しました)
1位ソリューション(資料10):
・画像の前処理は行わず、サイズの変更のみだった(画像の性質という意味ではかなり異なるので、これは参考外)
・Inceptionやse-resnextの合計8個のモデルの平均を最終的な提出に用いた
・但し、時間がなかったからEfficientNetを使わなかっただけで、あと2週間あったら実装したかった
・入力サイズは極端に大きくした(public LBではあまり効果を見受けられなかったがprivate LBでは恩恵を受けられる可能性があったため)
・損失関数は nn.SmoothL1Loss() のみを使用
・Data Augmentationは色んな手法を用いた
・最後のpooling層では、従来のaveragepoolingではなくgeneralized mean poolingのほうが良かった
・データの偏りをもとに、qwkの重みを調整した ←重要!
4位ソリューション(資料11):
・Densenet,se-resnextの表現力が乏しかった反面、efficientnetは良いスコアが出た。そこでb3~b5のefficientnetを用いた。画像サイズは
B3 image size: 300
B4 image size: 460
B5 image size: 456
とした。
・画像の前処理は最終的には行ってないものを提出した。
・Data Augmentationは多くのライブラリを使用して行った。
・trainingは複数モデルで行い、上位のepochをピックアップ。それらを単純に平均化したものを用いた。
・TTAとしてflipのみを用いた。
・使用できるデータはテストデータでも(trainingに)使用した。
7位ソリューション(資料12):
・Inception,se-resnextを用いた。EfficientNetは上手く行かなかった。
・TTAには水平flipだけ用いた。
・回帰モデルと分類モデル両方を試した
・data augmentationはたくさん入れた。減らしても良い結果は出なかった。
・optimizerはRAdamが一番良かった(RAdamはpytorchには実装されていない。資料13を参照)
・最終的には4つのモデルの平均値を用いた。
4-5.discussionで得た知見
過去コンペのみならず、PANDAコンペ自体でのdiscussionも有益な情報が得られるため、よく確認しました。特に以下の情報に注目しました。
・バッチサイズが3より小さいと、バッチ更新の際のノイズがあまりにも大きい。(この意見は多く、メモリと相談してできれば大きめにしたほうが良さそう。)
・研究所ごとの学習には意味がなさそうである。
・250万ピクセルを超える画像を用いた学習には意味がなさそうである(それ以上は精度の向上に寄与しないという意味で)。sqrt(2500000) = 1580x1580 …これは私のやり方でギリギリくらいです。6*256=1536
・パラメータ数が多すぎるモデルはオーバーフィットする。(EfficientNetでは、b0からb7にかけてパラメータ数が増加していきます)
・スケジューラーにGradualWarmupScheduler、CosineAnnealingWarmRestarts, CosineAnnealingLR, OneCycleLRなどを試している人がいる。
・criterionにCrossEntropyではなく、BCEwithlogitlossを使っている人が多いように見える。(onehotな値のみでなく、少数を扱えるなどのメリットが有る)
4-6.改善案(初期)
やったことをズラッと並べます。
・Criterion(損失関数)をCrossEntropy→BCEwithlogitlossに変更
・BCEwithlogitlossのままbinningを適用
binningとは
正解ラベル:[0, 0, 0, 1, 0, 0]→grade4
予測:argmax([0, 0, 0.5, 1, 0.5, 0])→grade4
としていたものを、
各グレード以上を満たすという考え方で、
正解ラベル:[1, 1, 1, 1, 0]→grade4
予測:sum([1, 0.9, 0.8, 0.6, 0.2])→round(3.5)→grade4
※配列長が5になっているのは、grade0を[0,0,0,0,0]として表せるようになったため。
というように予測する手法です。今回のISUPgradeのような、値同士が連続的な関係になっている場合に用いられます。
・マスク画像を用いて、診断領域外の部分を黒塗りにする
いいアイデアじゃないかな~と思ったのですが、むしろ精度が下がりました。(但し、trainingのlossの減少が早くなったのでそういう意味では効果はあるのかなと思います)
具体的には下図のような感じです。
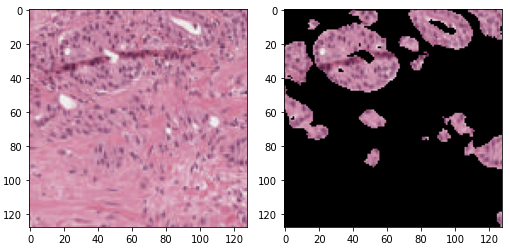
図4-6-1 : マスク前(左)とマスク後(右)
実際に学習に用いる際には、このマスク画像を適用する確率を指定して用いました。結果的に確率0の場合が一番精度が出ました。(コード書くの大変だったのでちょっとショックでした)
・schedulerをwarmup→CosineAnnealingLRに変更。
ちょっと最後までwarmupを使う理由が分かりませんでした。普通にCosineAnnealingLRだけでいい気がする。(となってくると、LambdaLRでも別にいい気がする…)
4-7.改善案2(1週間前~)
実は、4-6の改善案では何一つ改善している部分が存在しません。(コンペの経験が浅すぎる)
これなら、notebookに上がってるコードをパクって提出したほうがいいスコアが出ます。
コンペも残り7日と終了が迫ってきたところで、"メダルを取るための選択肢"を2つ考えました。
(1) 時間的・環境的に上位スコアを出せるモデルを作成できそうにないので、notebook(コンペサイト上で公開されているコード)の上位学習済みモデルを再学習させて改善を図る。
(2) 分類モデルではなく回帰モデルに今からシフトし、超特急で学習させる。
特に(2)の方は技術的にも実装できるか怪しく、比較的(1)が安全策ですが、この両者を並行して作成することにしました。
(1)はメモリコスト上、kaggleのnotebookでしか動作できないので、(1)をnotebookで、(2)をローカル環境で進めました。
そもそも、なぜ(2)案がいきなり出てきたかと言うと、quadric weighted kappaについて調べていた時に(資料17)の記事を見つけたからです。
この記事には、「評価指標がqwkの場合には、分類問題として解くのではなく、回帰モデルを作成してから分類の閾値を最適化するのが一般的だ(意訳)」と書かれています。
確かに、4-4の過去コンペの1位ソリューションも回帰モデルを作成してから分類、という手法でした。
これを試してみたくなりました。が、今まで散々分類モデルをやってきたので、いきなり回帰にできるかと言われると微妙なところです。賭けの部分が大きかったのですが、どうせ(1)案も学習時間が大半で時間は余るので、やるだけやってみることにしました。
が、1はある程度結果が出たのですが、2はほとんどダメでした。そもそも、binning処理が(合計を取るという意味で)結構回帰的な考え方に基づいていたので、わざわざ回帰モデルを作成する意味は無かったのかも…(めちゃくちゃ勉強不足です)
ここまでやった時点で、ほとんど手の打ちようがなくなったのでメダルは無理だろうと思っていました。
4-8.改善案3(1日前~)
コンペも終盤、ほとんどnotebookやdiscussionの動きが無くなった1日前に、資料19のnotebookを公開した人がいました。これは従来公開されていたnotebookのLBスコアを0.001だけ上回るもので、内容も資料15とほぼ同じものでしたが、モデルにEfficientNet b1 を用いているという点で異なっていました。これは、図3-2の通り、EfficientNetのパラメータ数がb0より少し増えたものです。注目すべきは、その学習したepoch数です。このモデルはENet b0で30世代かかって0.875のスコアを出していたものを、たった8世代で0.876に更新していました。
と、いうことは…?これをもうちょっと学習させればメダルまでは届かずとももうちょっとスコアが上がるよな…?
ということで、コンペ締め切り前日の夜になって急いでnotebookをパクって、再学習のコードを5世代回しました。(学習率は賭けで1e-5くらいに設定しました)
翌朝9時締切のものを8時半くらいにquadratic weighted kappaのしきい値を調整してから提出し**(この作業が実はめっっっっちゃくちゃ重要でした)**、LBで225位のスコアしか出なかったのを確認して、「やっぱダメか~」となっていました。
5.結果(4-8続き)
が、
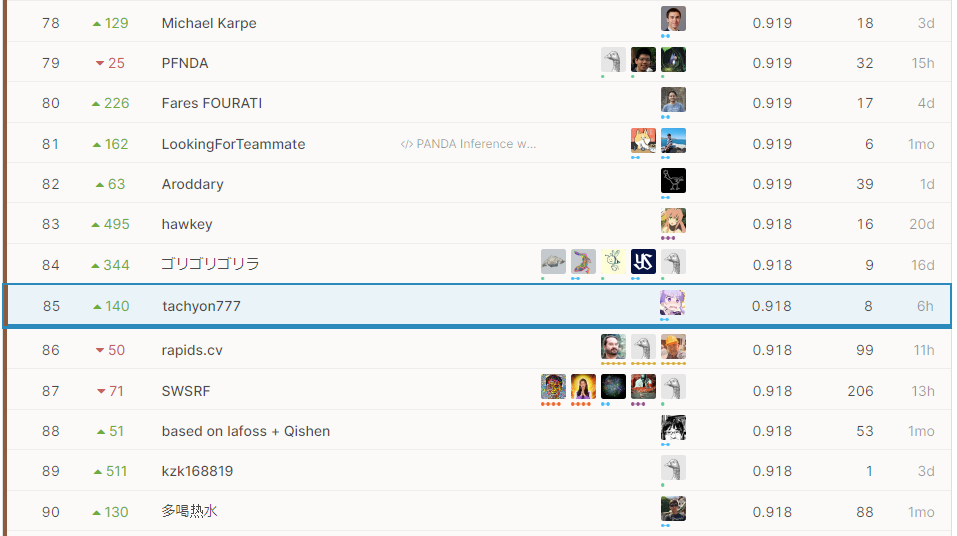
謎のshake upが発生し、Public LBに対して140位上昇した85位の結果となっていました。
Kaggleのサイトの仕様上、自分の順位-25位くらいの場所から表示されるので、60位くらいから表示されたときにはめちゃくちゃビビりました。
実は、銅メダル入賞圏内のスコアが0.917以上だったので、4-8にめちゃくちゃ重要と書いたしきい値の調整が無ければメダルは取れていませんでした。(実験的に、しきい値の調整は0.002程度スコアが上がることが分かっていたので)
結果的には人のモデルパクって入賞したわけですが、この2週間かなり注力してきた分、嬉しかったです。
ちなみに、その人は…
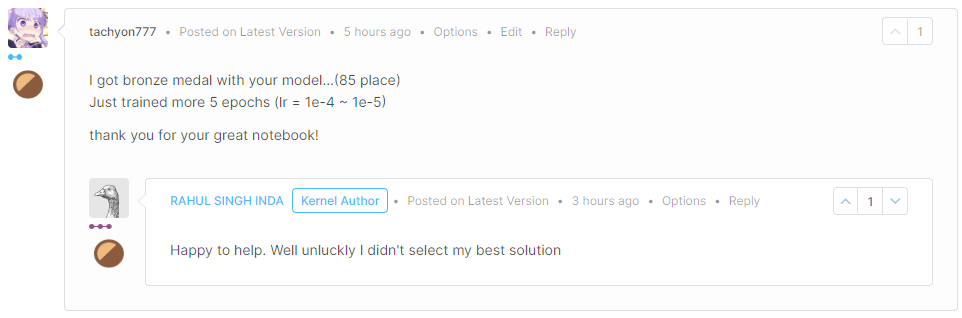
図5-1 : 資料19コメント(資料19より引用)
残念ながら入賞されなかったみたいです。(いつか恩返ししたい)
6.考察
1位のfam_taroさんの投稿されたDiscussion(資料21)を元に考察していきます。
まず、データのノイズ除去が重要だったそうです。どうやら事前に運営側から「ラベルは完全ではない」という情報が公開されていたようで(全く知りませんでした)、上位入賞の為にはこの処理を行う必要があったようです。1位解法では、このノイズ除去に、クロスバリデーションしたときの結果の差が大きいようなラベルのデータを取り除いて再学習するという方法を取ったようです。誤差が大きいようなラベルがtrainingデータに4%ほど含まれていたようです。勉強になります。
また、データの加工や画像サイズ、画像の前加工についてはおおよそ公開されているnotebook(主に資料8と資料15)を元にして作成したようです。正直、もっとグリソンスコアの方から分類したり、全く違うアプローチをしているのだろうなと考えていたので意外でした。但し、画像サイズはkaggleのnotebookのスペックでは実現不可能な大きさでの学習を行っている方もいました。
モデルとしてはEnet b0,b1,Resnext50の予測の平均を用いたということです。
元々quadric weighted kappaはshakeが起きやすい評価指標なので、特にshakeに強いモデルを最終的に選択したということでした。
今回の最終的なモデルの改善点として、もっと学習すれば更に精度の向上が期待できたと思います。理想的には30世代回したほうが良いと思うので、あと17世代程度改善の余地があったと考えられます。あと、時間的な都合上クロスバリデーションを行っていません。これができれば更に精度が上がったと思いますが、このコンペを通して一度もクロスバリデーションできていないので、どの程度良くなるのかは分かりません。更に、Enetb0よりもb1の方が良かったので、b2やb3も良かったのではないかと考えられます。但し、パラメータ数が増えると過学習しやすくなるため、ちょうど良いパラメータ数のモデルを模索する必要がありそうです。1位solutionの方がb1までを用いていることから、b2以降はあまり意味がなかったのかな?とも感じます。同じような意味で、他の構造のモデル(ResNextなど)も試すべきだったと思います。
全体を通して、もっと時間に余裕を持って参加すべきだったと思います。短期集中という意味では良かったですが、時間がないと試せることも限られてきてしまう事が分かりました。(特に高速な学習環境が無いので…)
7.終わりに
結果的に銅メダルが取れた訳ですが、実際に自分で考えて作ったモデルではないのでまっったく実力によるものではないです。(努力賞ということで…)
但し、今回初めて本格的にコンペに参加したことで、画像コンペに対する取り組み方が一通り分かりました。
次のコンペとして、CT画像から肺機能の低下度合いを検出するコンペ(資料20)を考えています。
今回の反省を活かして、次は自力でメダルを取れるよう頑張りたいです。
8.参考文献
1.門脇大輔 阪田隆司 保坂桂佑 平松雄司 Kaggleで勝つ データ分析の技術 技術評論社 2019
2.小川祐太郎 つくりながら学ぶ!Pytorchによる発展ディープラーニング マイナビ出版 2019
3.笹野公伸 岡田保典 安井弥 シンプル病理学 改訂第7版 南江堂 2015
4. https://www.kaggle.com/rohitsingh9990/panda-eda-better-visualization-simple-baseline
5.https://arxiv.org/abs/1905.11946
6. https://arxiv.org/abs/1905.04899
7. https://www.kaggle.com/c/prostate-cancer-grade-assessment
8. https://www.kaggle.com/iafoss/panda-16x128x128-tiles
9. https://www.kaggle.com/c/aptos2019-blindness-detection/overview
10. https://www.kaggle.com/c/aptos2019-blindness-detection/discussion/108065
11. https://www.kaggle.com/c/aptos2019-blindness-detection/discussion/107926
12. https://www.kaggle.com/c/aptos2019-blindness-detection/discussion/108058
13. https://github.com/jettify/pytorch-optimizer
14. https://pytorch.org/docs/stable/nn.html#smoothl1loss
15. https://www.kaggle.com/haqishen/train-efficientnet-b0-w-36-tiles-256-lb0-87
16. https://github.com/albumentations-team/albumentations_examples/blob/master/notebooks/showcase.ipynb
17. https://qiita.com/kaggle_master-arai-san/items/d59b2fb7142ec7e270a5#qwk
18. https://www.kaggle.com/c/prostate-cancer-grade-assessment/discussion/165632
19. https://www.kaggle.com/rsinda/panda-inference-efficientnet-b1
20. https://www.kaggle.com/c/osic-pulmonary-fibrosis-progression/overview
21. https://www.kaggle.com/c/prostate-cancer-grade-assessment/discussion/169143