はじめに
本記事では、医療画像を用いた研究を行う方、自作した深層学習モデルを臨床に導入しようと考えている方、あるいはコンペティションでDICOMデータを取り扱う必要のある方向けに、病院内ネットワークからDICOMデータを取り出すための匿名化処理から、深層学習モデルの予測などをDICOM形式で出力する方法まで、DICOMデータをpythonで扱うにあたって必要になるであろう情報に絞って紹介していきます。
特に後半部分では、CT画像と、その輪郭データ(RT Structure)について扱いますが、他モダリティについても応用できる部分が多いかと思います。
正しい情報の記述を心がけますが、間違いに気づかれた方はコメントで教えていただけますと幸いです。
前提知識:
Python
DICOMとはなんぞや
目次
- pydicomを使おう
- 匿名化をしよう
- 画像データを出力しよう
- テーブルデータを出力しよう
- RT Structureを画像化・出力しよう
- 推論したセグメンテーションデータをRT Structureに出力しよう
1. pydicomを使おう
pydicomは、dicomデータを扱うためのpythonライブラリです。インストール方法は以下のとおりです。
pip install pydicom
pydicomを使えば、dicomデータの全てにアクセスすることができますが、dicomデータはツリー構造になっており、かなり泥臭い作業を行うハメになります。
そこで、いつでも簡単にdicomヘッダや画像データを確認できるようにdicompylerというソフトをPCに入れておくのがオススメです。
僕は、dicomタグを扱う作業をpydicomで行う際は、同じデータをdicompylerで開いておいて、dicomタグをすぐ調べられるようにしています。
余談ですが、線量分布・コンツーリングデータなどの放射線治療分野のデータを併せて表示したい場合は、3D Slicerがめちゃオススメです。
では、pydicomを扱う練習として、kaggle notebookを用いてRSNA-MICCAI Brain Tumor Radiogenomic Classificationの適当なdicomファイルを読み込んでみましょう。
notebookはこちらで公開しています。
必要なモジュールをimportします。
import numpy as np
import cv2
import matplotlib
import matplotlib.pyplot as plt
%matplotlib inline
import pydicom
次に、適当なdicomファイルを読み込みます。
#dicomファイル読み込み
file_path = "/kaggle/input/rsna-miccai-brain-tumor-radiogenomic-classification/train/00000/T1w/Image-10.dcm"
dicom = pydicom.read_file(file_path)
dicomヘッダ一覧を表示するためには、dicom.dirを用います。
#dicomタグ一覧の表示
dicom.dir
出力には、左からTag, Name, VR, Valueが表示されています。
※VR(value representation) ... データ型を2文字で表すもの。
<bound method Dataset.dir of Dataset.file_meta -------------------------------
(0002, 0010) Transfer Syntax UID UI: Implicit VR Little Endian
-------------------------------------------------
(0008, 0005) Specific Character Set CS: 'ISO_IR 100'
(0008, 0008) Image Type CS: ['DERIVED', 'SECONDARY']
(0008, 0016) SOP Class UID UI: MR Image Storage
(0008, 0018) SOP Instance UID UI: 1.2.826.0.1.3680043.8.498.19923580803426667111874409007572708600
(0008, 0050) Accession Number SH: '00000'
(0008, 0060) Modality CS: 'MR'
(0008, 103e) Series Description LO: 'T1w'
(0010, 0010) Patient's Name PN: '00000'
(0010, 0020) Patient ID LO: '00000'
(0018, 0023) MR Acquisition Type CS: '2D'
(0018, 0050) Slice Thickness DS: '5.0'
(0018, 0081) Echo Time DS: None
(0018, 0083) Number of Averages DS: '1.0'
(0018, 0084) Imaging Frequency DS: '127.742365'
(0018, 0085) Imaged Nucleus SH: '1H'
(0018, 0086) Echo Number(s) IS: '1'
(0018, 0087) Magnetic Field Strength DS: '3.0'
(0018, 0088) Spacing Between Slices DS: '1.0'
(0018, 0091) Echo Train Length IS: '1'
(0018, 0093) Percent Sampling DS: '100.0'
(0018, 0094) Percent Phase Field of View DS: '80.0'
(0018, 0095) Pixel Bandwidth DS: '122.07'
(0018, 1094) Trigger Window IS: '0'
(0018, 1100) Reconstruction Diameter DS: '240.0'
(0018, 1310) Acquisition Matrix US: [0, 320, 224, 0]
(0018, 1312) In-plane Phase Encoding Direction CS: 'ROW'
(0018, 1314) Flip Angle DS: '73.0'
(0018, 1316) SAR DS: '1.74313'
(0018, 5100) Patient Position CS: 'HFS'
(0020, 000d) Study Instance UID UI: 1.2.826.0.1.3680043.8.498.21557373119637153130481842401167746353
(0020, 000e) Series Instance UID UI: 1.2.826.0.1.3680043.8.498.10088481353579602776972549292627734001
(0020, 0011) Series Number IS: '8'
(0020, 0013) Instance Number IS: '10'
(0020, 0032) Image Position (Patient) DS: [-117.109, -142.138, -26.3763]
(0020, 0037) Image Orientation (Patient) DS: [1, -0, 0, -0, 1, 0]
(0020, 0060) Laterality CS: ''
(0020, 1040) Position Reference Indicator LO: ''
(0020, 1041) Slice Location DS: '-26.37625122'
(0020, 9057) In-Stack Position Number UL: 10
(0028, 0002) Samples per Pixel US: 1
(0028, 0004) Photometric Interpretation CS: 'MONOCHROME2'
(0028, 0010) Rows US: 512
(0028, 0011) Columns US: 512
(0028, 0030) Pixel Spacing DS: [0.468800008296967, 0.468800008296967]
(0028, 0100) Bits Allocated US: 16
(0028, 0101) Bits Stored US: 16
(0028, 0102) High Bit US: 15
(0028, 0103) Pixel Representation US: 1
(0028, 1050) Window Center DS: '1854.0'
(0028, 1051) Window Width DS: '3709.0'
(0028, 1052) Rescale Intercept DS: '0.0'
(0028, 1053) Rescale Slope DS: '1.0'
(0028, 1054) Rescale Type LO: 'US'
(7fe0, 0010) Pixel Data OW: Array of 524288 elements>
dicomヘッダの特定のデータにアクセスする方法は、よく使う方法としては2つあります。
1つ目は、 Name(Attribute Name) を用いてアクセスする方法です。例えば、Patient IDにアクセスする場合、
#Nameを用いてデータにアクセス
#dicom.dirでは、"Patient ID"ですが、スペースは省略します。
dicom.PatientID
#Out [5]:'00000'
とします。
ここで、dicom.dirで表示されるNameと変数名でアクセスする際のNameが多くの場合異なります。
例えば、"Patient's Name"は、"PatientName"になっていたり。
変数名でアクセスする際のName一覧は、dicom.dirを関数で呼び出すことでリスト型で得られます。
#関数として呼び出すとAttribute Nameのリストが得られる
dicom.dir()
['AccessionNumber',
'AcquisitionMatrix',
'BitsAllocated',
'BitsStored',
'Columns',
'EchoNumbers',
'EchoTime',
...]
Nameでアクセスする方法は可読性が高く便利ですが、上記のNameの違いがバグの原因になりやすいので、僕がオススメするのはもう1つの方法です。
もう1つは、Tagでデータにアクセスする方法です。
Nameでのアクセスと比較してスペースや文字大小を考えなくて良い分、こちらのほうが安全かもしれません。
16進数でのアクセスのため、最初に0xをつけます。
例えば、Patient's NameのTagは(0010, 0020)なので、
#Tagを用いてデータにアクセス
dicom[0x0010,0x0010].value
#Out [6]:'00000'
となります。
最後に、画像データへのアクセスを見ておきましょう。
画像データは、dicom.pixel_arrayでnumpy形式の配列を得ることができます。
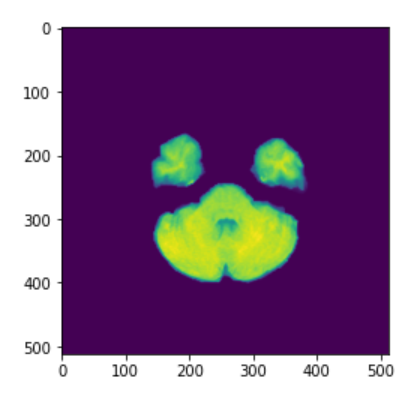
plt.imshow(dicom.pixel_array)
ひとまず、こんなところでpydicomの基本的な使い方は終わります。
2. 匿名化をしよう
自施設のデータを用いた研究を行おうとする時、まずデータの取り出しから入るはずです。この際、必ず必要になる作業が、倫理委員会への申請・承認と、患者個人情報の匿名化になります。
通常、患者データの匿名化を手作業で行うと、膨大な時間を要しますが(経験上)、プログラムを書いておけば一瞬で終わらせることができます。
ここでは、pydicomを用いた患者個人情報の匿名化を行うプログラム例を紹介します。
なお、このプログラムを走らせるPCは院内ネットワークへの接続を承認されているものとします。
DICOMヘッダに記載される、匿名化する必要のある個人情報は以下の3つになります。(患者年齢や、撮影日時まで匿名化することもあります)
| 内容 | Name | Dicom Tag |
|---|---|---|
| 患者名 | PatientName | (0010, 0010) |
| 患者ID | PatientID | (0010, 0020) |
| 患者生年月日 | PatientBirthDate | (0010, 0030) |
さて、匿名化作業を始めるにあたって、想定されるディレクトリ構成を以下のようにします。
今回は、患者IDを示すディレクトリ下にCT画像のdicomファイルが複数入っていることを想定します。
.
├── patient_info.csv #匿名化前と、後の患者情報を管理するcsvファイル
├── anonymization.py #匿名化を行うpythonプログラム
├── input_dir #dicomデータが入っている入力ディレクトリ
| ├── 1000000 #患者ID、このディレクトリ下に、CT画像が入っている
| | ├── CT0001.dcm #ID1000000の患者のCT画像の1枚目
| | ├── CT0002.dcm #ID1000000の患者のCT画像の2枚目
| | └── ...
| ├── 1000001
| | └── ...
| └── ...
├── anonymized_dicom_dir #ここに匿名化後のデータが出力される
patient_info.csv
patient_info.csvは、匿名化前と、後の患者情報を管理するcsvファイルです。匿名化後の患者IDは各施設で命名規則があると思いますので、よしなにしてください。
このファイルは、匿名化前後のデータの紐づけを行う唯一の情報になりますので、必ず院内ネットワーク側に保存し、持ち出さないようにします。
今回はディレクトリ名を患者IDと同じにして、input_dir_name=PatientID_before, output_dir_name=PatientID_afterとしました。
| input_dir_name | PatientID_before | PatientID_after | output_dir_name |
|---|---|---|---|
| 1000000 | 1000000 | 0001 | 0001 |
| 1000001 | 1000001 | 0002 | 0002 |
| ... | ... | ... | ... |
anonymization.py
これを踏まえて、匿名化のプログラムは以下のようになります。
import os
import datetime
import numpy as np
import pandas as pd
from tqdm import tqdm
import pydicom
input_dir = "./input_dir" #入力ディレクトリパス
output_dir = "./anonymized_dicom_dir" #出力ディレクトリパス
#patient_info
df = pd.read_csv("patient_info.csv")
for patient_path in tqdm(os.listdir(input_dir)):
for i in range(len(df)):
if str(df["input_dir_name"][i]) == patient_path:
sr_info = df.iloc[i]
break
#出力先ディレクトリを先に作成
opath = os.path.join(output_dir,sr_info["output_dir_name"])
if not os.path.exists(opath):
os.mkdir(opath)
#全ファイルのdicomヘッダを匿名化
for file_path in os.listdir(os.path.join(input_dir,patient_path)): #file_path:入力ファイル名(Slice001.dcm)はそのまま
ipath = os.path.join(input_dir,patient_path,file_path)
dicom = pydicom.read_file(ipath)
#匿名化
dicom.PatientName = "Anonymous"
dicom.PatientID = str(sr_info["PatientID_after"])
dicom.PatientBirthDate = datetime.datetime.now().strftime("%Y%m%d") #患者誕生日は匿名化日にでもしておきましょう
#保存
dicom.save_as(os.path.join(opath,file_path))
ここで、入力ファイル名(CT0001.dcm)はそのままで出力していますが、システムによってはファイル名に患者IDを入れる機種があったりするので、その際はまた適当に番号振るなりして匿名化ください。CTデータのようにスライス番号がある場合は、InstanceNumber (Tag : (0020,0013))をファイル名にしてしまうのがオススメです。
3. 画像データを出力しよう
画像が欲しいなら最初から画像ファイルとして取り出せば面倒な匿名化もいらないじゃん!と思うかもしれませんが、なんだかんだdicomヘッダの情報はあったほうが良いです。ぱっと思いつく範囲で、以下のような理由があります。
- CT画像において、寝台の移動方向(INTO GANTRY,OUTOF GANTRY)を示すヘッダが無いと、後からスライスの方向が上下逆で手動で修正する羽目になる。
- セグメンテーション出力をRT Structureに戻す際に、dicomファイルが無いと点群座標が合わせられない(後述)。
- 実空間距離を用いた評価を行う際、Pixel Spacingが無いと、1ピクセルあたりの距離を求めることができない。スライス厚、空間分解能も求められない。
- 患者の年齢や性別の分布といった情報が無いと、論文書く時困る。
- 複数の撮影機器を用いていた場合、機器間の変動(ドメインシフト)を後から検証できない。
これらの理由から、下図に示すように dicomファイルのまま 匿名化→画像やテーブルデータを出力、という形式を取っています。
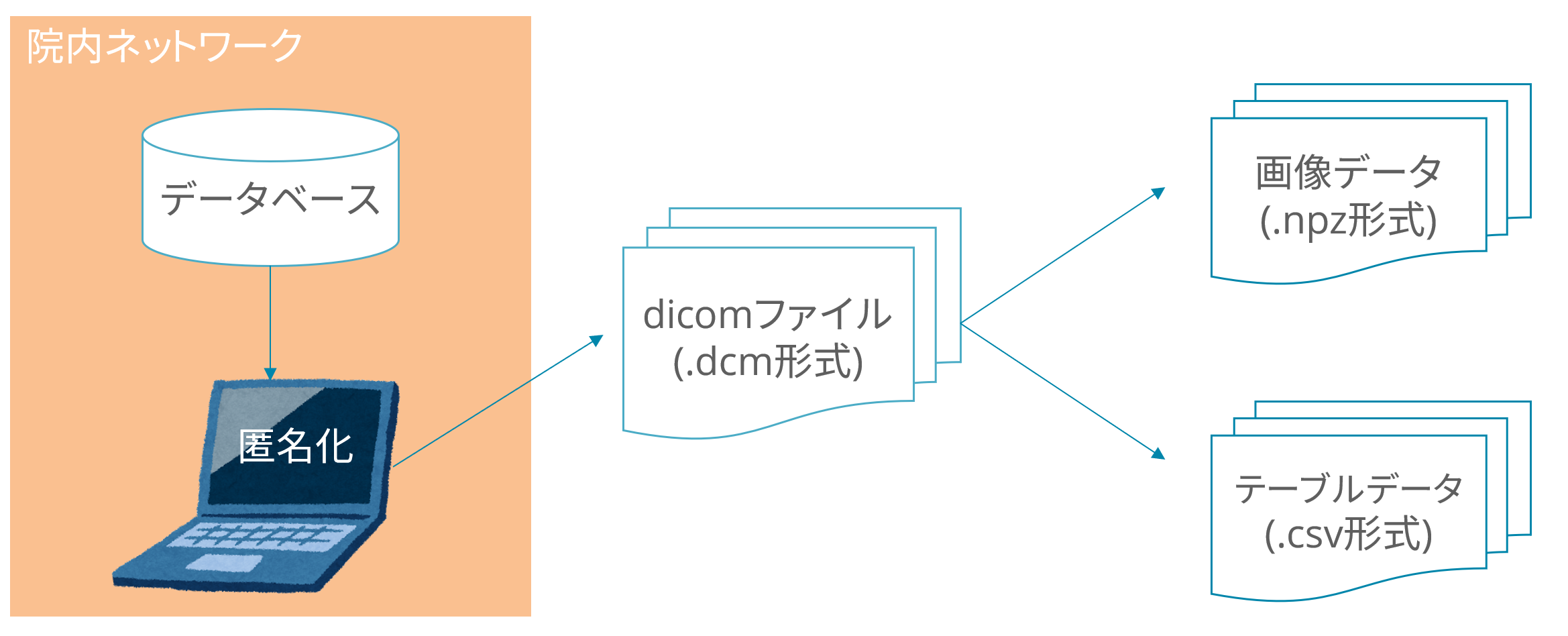
そんなわけで、匿名化の終わったdicomファイルから画像データを取り出してnumpyの可逆圧縮形式である.npzに出力したいと思います。
pngでも良いかもしれませんが、画像読み込み時にcv2やpillowを通すのが嫌とか、間違いなく生値で保存できるとかそんな理由でnpzを使うことが多いです。(ここは僕の好みですのでおまかせします)
先程匿名化を行ったanonymized_dicom_dirの中身を、院内ネットワークから取り出して計算用PCに取り込んだとして、新たに作成したimage_dirに患者毎にディレクトリを作成し、画像データを保存していきましょう。
.
├── anonymized_dicom_dir #匿名化されたディレクトリ
| ├── 0001 #匿名化された患者ID、このディレクトリ下に、CT画像が入っている
| | ├── CT0001.dcm #ID0001の患者のCT画像の1枚目
| | ├── CT0002.dcm #ID0001の患者のCT画像の2枚目
| | └── ...
| ├── 0002
| | └── ...
| └── ...
├── dicom2npz.py #画像出力を行うpythonプログラム
└── image_dir #npz形式で出力した画像データを保存するディレクトリ
dicom2npz.py
import os
import datetime
import numpy as np
import pandas as pd
from tqdm import tqdm
import pydicom
def make_image_dataset(input_dir,output_dir,patient_id):
if not os.path.exists(os.path.join(output_dir,patient_id)):
os.mkdir(os.path.join(output_dir,patient_id))
for path in os.path.join(input_dir,patient_id):
dicom = pydicom.read_file(os.path.join(input_dir,patient_id,path))
slice_num = dicom.InstanceNumber #ここでは、出力ファイル名をスライス番号にしてしまいます
img = dicom.pixel_array
if img.dtype == np.uint16: #uint16にして保存する機器があったりしますが、CT値は-1000が最小値なので、int16にします。
img = img.astype(np.int16)
img += int(dicom.RescaleIntercept) #RescaleInterceptを足してCT値に変換
np.savez_compressed(os.path.join(output_dir,patient_id,str(slice_num)+".npz"),img)
if __name__ == "__main__":
input_dir = "./anonymized_dicom_dir"
output_dir = "./image_dir"
for patient_id in tqdm(os.listdir(input_dir)):
make_image_dataset(input_dir,output_dir,patient_id)
途中で、型をuint16からint16に直したり、RescaleInterceptで画像全体に値を足している処理があります。
CT値の最小は-1000ですが、ここに1000を足してやれば符号なし(uint型)で保存する事ができます。CT装置側でこの形式で保存されることが多いです。
このため、予めこの-1000(稀に-1024)をdicomヘッダにRescaleInterceptという名前で保存しておくことで、画像をuint16で保存して、後からこの分だけ足すことで正しいCT値に戻すようになっています。
他のモダリティではこのあたりの処理は違ってくると思うので、データに合った処理をしてください。
4. テーブルデータを出力しよう
dicomデータから画像を取り出すことができたので、次はdicomヘッダのデータを取り出して、csv形式で保存しましょう。
今は使わないと思っても、後々必要になるデータもあるので、できるだけ多くのデータを取り出しておきましょう。(意味がわからないものも含めて全部取り出してしまってもいいかもしれません)
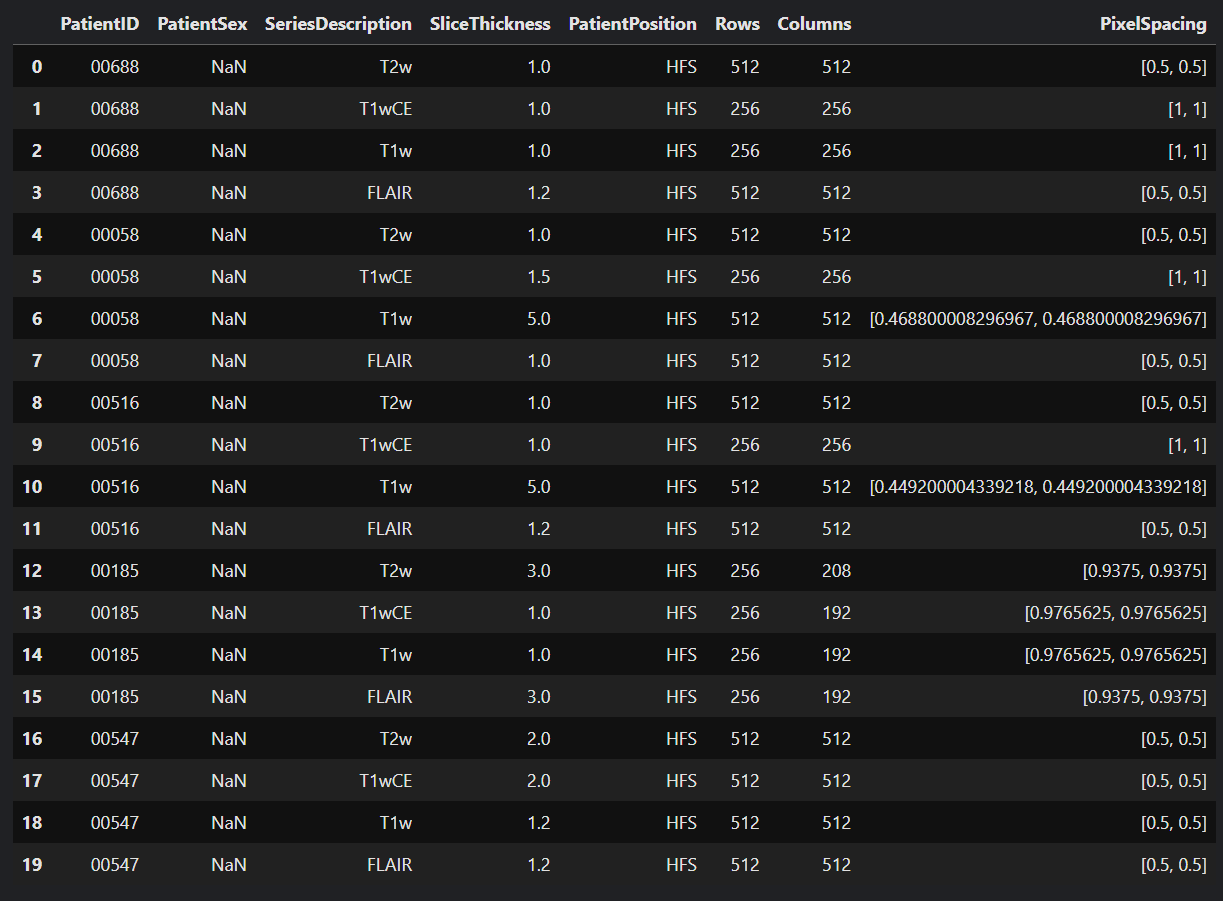
ここでは、先程も用いたRSNA-MICCAI Brain Tumor Radiogenomic Classificationのdicomデータを使ってpandasのDataFrameにし、出力してみます。
notebookはこちらで公開しています。
特に説明することもないので、一気に載せて出力を見てみます。
import os
import numpy as np
import pandas as pd
import cv2
import matplotlib
import matplotlib.pyplot as plt
%matplotlib inline
import pydicom
df = pd.DataFrame(columns = [
'PatientID',
'PatientSex',
'SeriesDescription',
'SliceThickness',
'PatientPosition',
'Rows',
'Columns',
'PixelSpacing',
])
base = "/kaggle/input/rsna-miccai-brain-tumor-radiogenomic-classification/"
for patient_dir in os.listdir(os.path.join(base,"train"))[:5]: #お試しなので5人分だけ
for series in os.listdir(os.path.join(base,"train",patient_dir)):
#適当に1画像読み込む
file_path = os.path.join(base,"train",patient_dir,series,os.listdir(os.path.join(base,"train",patient_dir,series))[0])
dicom = pydicom.read_file(file_path)
df = df.append({
'PatientID':dicom.PatientID,
#'PatientSex':dicom.PatientSex, #性別はこのdicomデータには無い
'SeriesDescription':dicom.SeriesDescription,
'SliceThickness':dicom.SliceThickness,
'PatientPosition':dicom.PatientPosition,
'Rows':dicom.Rows,
'Columns':dicom.Columns,
'PixelSpacing':dicom.PixelSpacing,
},ignore_index = True)
output_path = "/kaggle/working/" #出力パス
df.to_csv(os.path.join(output_path,"metadata.csv"),index=False)
これで、テーブルデータの出力が完了しました。後々、これらの情報が必要になったときにpandasで読み込んで、検証することができます。
5. RT Structureを画像化・出力しよう
ここからはコアな内容になるのでオマケ的な感じになりますが、特に放射線治療分野の研究をされている方には役に立つと思います。
RT Structureとは、放射線治療において臓器ごとに投与線量を管理するためのCT画像上における臓器のセグメンテーションデータです。
1つのdicomファイルとして、セグメンテーションデータは3次元の点群座標で与えられます。
ここでは具体的なdicomファイルの内容は省略し、一気にRT Structureの画像化コードを見てみましょう。大体今まで説明した感じで分かるはずです。
ディレクトリ構成ですが、治療計画装置からRTStruct.dcmを取り出して、患者ごとのCTファイルと同じディレクトリ下にあるものとします。
.
├── anonymized_dicom_dir #匿名化されたディレクトリ
| ├── 0001 #患者ID
| | ├── CT0001.dcm #ID0001の患者のCT画像の1枚目
| | ├── CT0002.dcm #ID0001の患者のCT画像の2枚目
| | ├── ...
| | └── RTStruct.dcm #RTStructure
| ├── 0002
| | └── ...
| └── ...
├── dicom2npz_rtstruct.py #rtstructから画像出力を行うpythonプログラム
└── mask_dir #rtstructの画像データを保存するディレクトリ
ここで新たに、skimageというライブラリが必要になるので、インストールしていない場合は以下のコマンドでインストールしましょう。
pip install scikit-image
本コードでは画像のマトリクスサイズが512x512となっていることを仮定していますが、もしバラバラな画像サイズが入る場合にはshapeを得るなどして対処してください。
セグメンテーションのラベルにはマルチクラスとマルチラベルがあり、フラグで選べるようになっています。
なお、コードは一部AAPMから引用しています。
import sys
import random
import os
import shutil
import numpy as np
import pandas as pd
import cv2
from skimage.draw import polygon
from tqdm import tqdm
import warnings
warnings.filterwarnings('ignore')
import pydicom
#organ_dict : rtstructureについている臓器ラベルを管理するためのdictです。表記ゆれがあるため、セグメンテーション対象の臓器をこのように管理しています。
#ここでは、例として前立腺癌治療の際にセグメンテーションする臓器を挙げています。
organ_dict = {
"BLADDER":["BLADDER","BLADDER_CONTRAST"],
"RECTUM":["RECTUM","RECTUM2"],
"PROSTATE":["PROSTATE","PROSTATE1","Prostate"],
"SEM_VES":["SEM_VES","SV"],
"FEMUR_HEAD_L":["FEMUR_HEAD_L","Femur_L"],
"FEMUR_HEAD_R":["FEMUR_HEAD_R","Femur_R"],
}
#続いて、インデックスで管理できるように辞書をもう1つ作成します。
organ_index_dict = {k:idx for idx,k in enumerate(organ_dict.keys())}
organ_dict_inv = {} #逆引きは表記ゆれに対してそれぞれkeyを割り振る
for k,v in organ_dict.items():
for s in v:
organ_dict_inv[s] = k
#http://aapmchallenges.cloudapp.net/forums/3/2/
def read_structure(structure,patient_path):
contours = []
for i in range(len(structure.ROIContourSequence)):
contour = {}
#contour['color'] = structure.ROIContourSequence[i].ROIDisplayColor
#contour['number'] = structure.ROIContourSequence[i].RefdROINumber
contour['name'] = structure.StructureSetROISequence[i].ROIName
#assert contour['number'] == structure.StructureSetROISequence[i].ROINumber
try:
contour['contours'] = [s.ContourData for s in structure.ROIContourSequence[i].ContourSequence]
contours.append(contour)
except:
print(f"patient:{patient_path} organ:{structure.StructureSetROISequence[i].ROIName} is empty.")
pass
return contours
def get_mask_from_RTStruct(patient_path):
"""
pathent_path : include slices,RTStructure dcm file
if "CT~.dcm" read as CT data, elif "RT~.dcm" read as RTStructure data.
return (S, C, H, W) shaped mask array Corresponds to each slices.
"""
#dicomファイルの先頭2文字が"CT"ならCT画像のファイル、"RT"ならRT Structureのファイルと区別しています。
lst = []
for i in os.listdir(patient_path):
if i[:2]=="CT":
lst.append(i)
elif i[:2]=="RS":
rt_path = i
lst2 = []
for path in lst: #ファイル名でスライス番号を管理していたが、一致しない場合があるためInstanceNumberで管理
dicom = pydicom.read_file(os.path.join(patient_path,path))
num = dicom.InstanceNumber -1 #zero index
lst2.append([num,dicom.ImagePositionPatient,dicom.PixelSpacing])
lst2.sort()
z = [i[1][2] for i in lst2] #z軸のindexでのアクセスをできるようにしておく
#Read RTStructure, correct the coordinates, and create the mask image.
structure = pydicom.read_file(os.path.join(patient_path,rt_path))
contours = read_structure(structure,patient_path)
arr = np.zeros((len(lst2),len(organ_dict),512,512),dtype=np.uint8)
for con in contours:
if con["name"] in organ_dict_inv:
organ_index = organ_index_dict[organ_dict_inv[con["name"]]]
for c in con['contours']:
nodes = np.array(c).reshape((-1, 3))
if len(nodes) == 1:continue #たまに1点だけのnodeがある
nodes[(nodes>-0.0001) & (nodes<0.0001)] = 0 #変な値修正
assert np.amax(np.abs(np.diff(nodes[:, 2]))) == 0 #z軸の値がスライス内で一定
z_index = z.index(nodes[0, 2])
pos_r,pos_c,spacing_r,spacing_c = lst2[z_index][1][1],lst2[z_index][1][0],lst2[z_index][2][1],lst2[z_index][2][0]
r = (nodes[:, 1] - pos_r) / spacing_r
c = (nodes[:, 0] - pos_c) / spacing_c
#cv2.polylinesで使用できるように変形
rr, cc = polygon(r, c)
arr[z_index,organ_index,rr,cc] = 1
return arr
def output_mask_from_array(masks_array,patient_id,multi_label=True):
"""
masks_array :
get_mask_from_RTStruct() ret array (S,C,H,W)
multi_label :
if False :
output array is (H, W),pixel values(0-255) represent organ classes.
For **Multi Class Segmentation**.
if True :
output array is (H, W, Class),pixel value is 0-1(OneHot).
For **Multi Label Segmentation**.
pathent_id :
ex.) "0001"
"""
output_path = os.path.join("./mask_dir",patient_id)
if not os.path.exists(output_path):
os.mkdir(output_path)
#multiclassの場合は、1つの2次元配列(H,W)に臓器indexを埋め込む
if not multi_label:
arr = np.zeros((masks_array.shape[0],512,512),dtype=np.uint8)
for slices in range(masks_array.shape[0]):
for label in range(masks_array.shape[1]):
mask = masks_array[slices,label,:,:]
arr[slices,mask==1] = label
for slices in range(masks_array.shape[0]):
np.savez_compressed(os.path.join(output_path,str(slices+1)+".npz"),masks_array[slices]) #0->1 indexed
#multilabelの場合は、1つのスライスのアウトプットが(C,H,W)
else:
for slices in range(masks_array.shape[0]):
np.savez_compressed(os.path.join(output_path,str(slices+1)+".npz"),masks_array[slices]) #0->1 indexed
if __name__ == "__main__":
input_dir = "./anonymized_dicom_dir"
output_dir = "./mask_dir"
for patient_id in tqdm(os.listdir(input_dir)):
mask = get_mask_from_RTStruct(os.path.join(input_dir,patient_id))
output_mask_from_array(mask,patient_id,multi_label=True)
以上でRT Structure -> マスク画像への変換が行えたかと思います。
6. 推論したセグメンテーションデータをRT Structureに出力しよう
今までの章で作成したデータセットを用いて深層学習モデルの学習・評価を行い、臨床上十分な恩恵が得られる結果が出たとしましょう。
そうしたらいよいよ臨床実装です!臨床実装に際しては病院にかけあって推論専用のPCを一台買ってもらうのが良いです。マシンパワーが必要な推論を行う場合はGPUを積んだ良いマシンを買ってもらいましょう。
回帰・分類モデルであれば推論したPCで結果をそのまま表示したり、以下のように推論したdicomデータにプライベートタグとして推論結果を追加して、dicomヘッダが表示できるソフトウェアで見れば良いです。(そう単純では無いかもしれませんが一つの方法として...)
import pydicom
from pydicom.dataelem import DataElement
dicom = pydicom.read_file("image.dcm")
prediction = model(dicom) #推論(適当な疑似コードです)
#プライベートタグの追加(タグ番号は適当に決めています。標準タグと同じ番号は使わないようにしましょう。)
dicom.add(DataElement("0xffff0001","DS",str(prediction)))
#-> (ffff, 0001) Private Creator DS: '0.5'
dicom.save_as("image_pred.dcm")
しかし、セグメンテーションの推論を放射線治療の治療計画に用いたい場合などは、RT Structureのファイル形式に戻してやる必要があります。
実装としてはRT Structure -> マスク画像の逆操作をしてやれば良いわけですが、マスク画像を入れると自動でRT Structureに変換してくれるrt_utilsという素晴らしいライブラリがあります。今回はこちらを使いましょう。
インストールは以下のとおりです。
pip install rt_utils
ディレクトリ構成は以下のとおりです。
prediction_dirに、推論したセグメンテーション出力をスライスごとに保存してあります。
セグメンテーションの出力は、各スライスにつき(C,H,W)のnpz形式で保存してあるものとします。※Cはクラス数
また、RT Structureとして出力する際にCTのdicomファイルを参照する必要があるので、匿名化する際に作成したoutput_dirを使います。
.
├── prediction_dir #セグメンテーション出力を保存してあるディレクトリ
| ├── 0101 #患者ID
| | ├── MASK0001.npz #ID0101の患者のマスク画像の1枚目
| | ├── MASK0002.npz #ID0101の患者のマスク画像の2枚目
| | └── ...
| ├── 0102
| | └── ...
| └── ...
├── anonymized_dicom_dir #匿名化されたdicomファイルのディレクトリ
| ├── 0101
| | ├── CT0001.dcm
| | ├── CT0002.dcm
| | └── ...
| ├── 0102
| | └── ...
| └── ...
├── dicom2npz_rtstruct.py #rtstructから画像出力を行うpythonプログラム
├── mask2rtstruct.py #推論したマスク画像からrtstructureへの出力を行うプログラム
└── rtstruct_eval_dir #出力先ディレクトリ
import sys
import random
import os
import shutil
import numpy as np
import rt_utils
from rt_utils import RTStructBuilder
from skimage.draw import polygon
import warnings
warnings.filterwarnings('ignore')
import pydicom
from dicom2npz_rtstruct import organ_index_dict
def contour_point_round(rtstruct):
for i in range(len(rtstruct.ds.ROIContourSequence)):
for j in range(len(rtstruct.ds.ROIContourSequence[i].ContourSequence)):
lst1 = [round(i,5) for i in rtstruct.ds.ROIContourSequence[i].ContourSequence[j].ContourData]
rtstruct.ds.ROIContourSequence[i].ContourSequence[j].ContourData = lst1
return rtstruct
def mask2dicom_save(masks,dicom_dir,output_dir,filename):
rtstruct = RTStructBuilder.create_new(dicom_series_path=dicom_dir)
masks = masks.astype(np.bool).transpose(1,2,3,0)[:,:,:,::-1] #(C,H,W,Z)にしたうえで、Zは降順
for k,v in organ_index_dict.items():
if masks[v].sum() != 0:
rtstruct.add_roi(mask=masks[v], name=k)
else:
print(f"organ not found:{k}")
#座標の桁数が多すぎると治療計画装置でエラーが出るので、小数点5桁に丸め込む
rtstruct = contour_point_round(rtstruct)
#コンツーリング全体の名前
rtstruct.ds.StructureSetLabel = "SegmentationInference"
#rtstruct.ds[0x0008,0x0013].value = str(int(float(rtstruct.ds[0x0008,0x0013].value))) #時間を少数→整数に変更
#rtstruct.ds[0x3006,0x0009].value = str(int(float(rtstruct.ds[0x3006,0x0009].value))) #時間を少数→整数に変更
#適当にCTを1枚読み込む
dicom_ct = pydicom.read_file(os.path.join(dicom_dir,os.listdir(dicom_dir)[0]))
Frame_of_Reference_UID = dicom_ct[0x0020,0x0052].value
#CTのFrame_of_Reference_UIDで書き換え(これがないと治療計画装置でエラー)
rtstruct.ds[0x3006,0x0010][0][0x0020,0x0052].value = Frame_of_Reference_UID
for i in rtstruct.ds[0x3006,0x0020]:
i[0x3006,0x0024].value = Frame_of_Reference_UID
rtstruct.save(f"{output_dir}/RS_{filename}.dcm")
if __name__ == "__main__":
input_dir = "./prediction_dir"
dicom_dir = "./anonymized_dicom_dir"
output_dir = "./rtstruct_eval_dir"
patient_id = "0101" #1~100までをtrainingに使用したことを想定して、101番目のpatient_idをevaluationに使います
mask = []
#推論したマスク画像の読み込み(スライスの順番)
#各マスク画像はnp.uint8のバイナリ値(0or1)で、(C,H,W)の順番
for i in range(len(os.listdir(os.path.join(input_dir,patient_id)))):
mask.append(np.load(os.path.join(input_dir,patient_id,"MASK"+str(i).zfill(4)))["arr_0"])
#ここで、mask画像のshapeは(Z,C,H,W)
mask = np.array(mask)
mask2dicom_save(mask,dicom_dir,output_dir,patient_id)
出力されたRT Structureのdicomファイルを、3D Slicerや治療計画装置から読み込めれば成功です。
おわりに
本記事では、実践向けのdicomデータの取扱いについて解説しました。
あとはGUIを実装すれば医療AIソフトウェアの完成ですね。pythonでは即席のものがpysimpleguiで作れるので、オススメです。
拙い文章&コードでしたが、以上となります。ありがとうございました。