本記事について
- 遺伝統計解析ソフトウェアPLINKを用いて主成分分析(Principal Component Analysis, PCA)を行なった結果を2次元平面上にプロットするスクリプトを書いた。
- スクリプトの入力・出力ファイルと実行方法を紹介する。
- スクリプトはこちら(GitHubへのリンク)
入力ファイルの準備
1. 主成分負荷量データの準備
ファイルフォーマット
1列目に家族ID, 2列目に個人ID, 3列目以降に主成分負荷量が入っているファイルを準備する。このようなフォーマットのファイルは、PLINKを用いて主成分分析を行うことで得られる。
# 1 FamID
# 2 Individual ID
# 3 PC1
# 4 PC2
...
PLINKによる主成分分析
遺伝統計解析ソフトウェアPLINKでは、主成分分析を行うことができる。
主成分分析とは、分散共分散行列あるいは相関行列の固有値分解に基づく次元削減手法で、一塩基多型(SNP)データの関連解析では、集団構造の可視化や、主成分負荷量を共変量とした交絡調整に用いられる。
$ plink --bfile ${bfile_name} --out ${outfile_name} --pca
PLINKによるPCAの出力結果として、${outfile_name}.eigenvecと${outfile_name}.eigenvalが得られる。
結果の図示には、${outfile_name}.eigenvec(各個人における各主成分の負荷量)を使用する。
2. 集団ラベルデータの準備
ファイルフォーマット
1列目に家族ID, 2列目に個人ID, 3列目に集団ラベル(人種など)が入っているファイルを準備する。(populations.txtとする。)
# 1 FamID
# 2 Individual ID
# 3 Group
スクリプトの実行方法
実行環境はPython3で、pandasとmatplotlibをインストールする。
実行は下記のオプションを指定して行う。
-
-eオプションには${outfile_name}.eigenvecファイルを指定 -
-pオプションにはpopulations.txtファイルを指定 -
-oオプションには出力されるディレクトリを指定
$ python plot_pca_gwas.py -e ${outfile_name}.eigenvec -p populations.txt -o ${output_directory}/
出力結果の確認
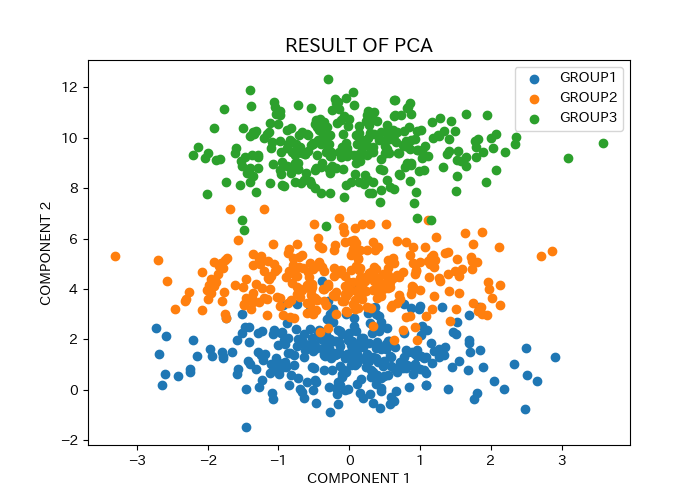
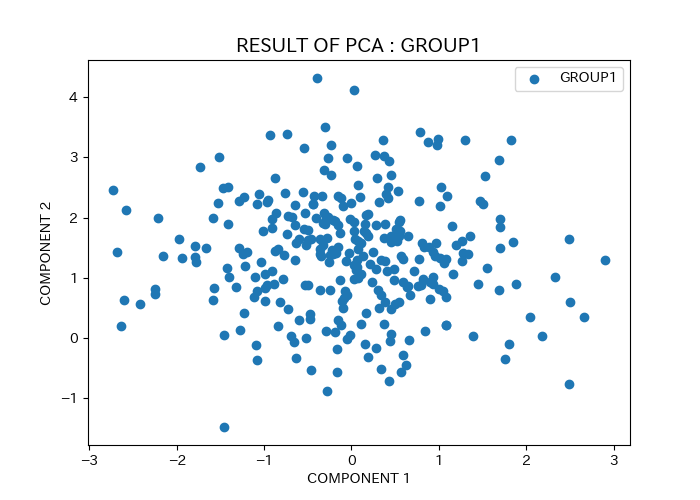
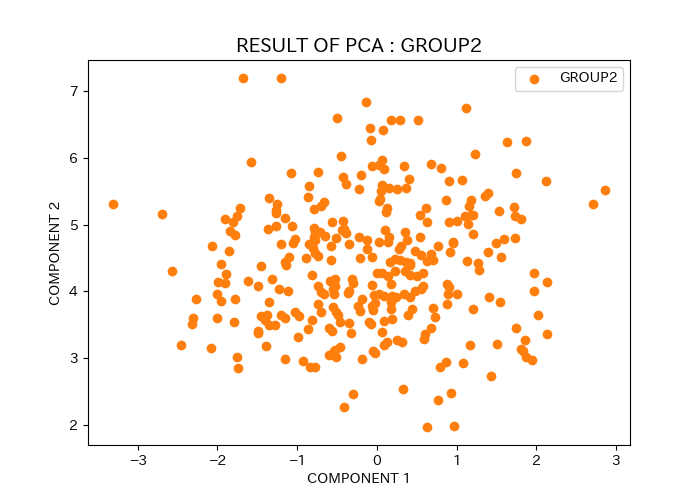
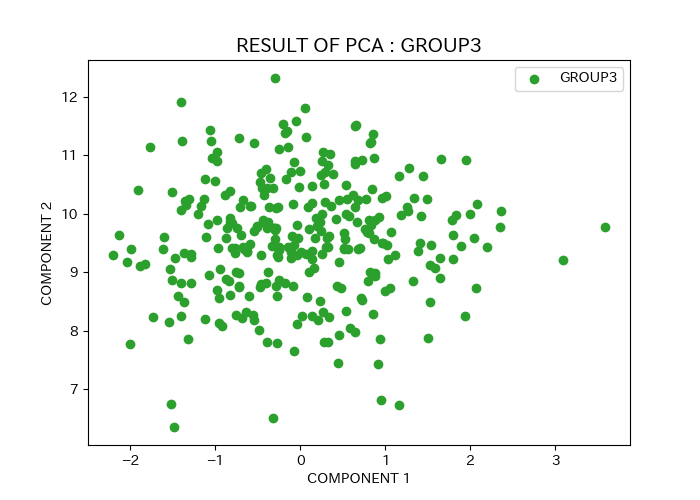
スクリプトの出力結果として、下記の画像が得られる。
-
pca.png:集団全体のプロット -
pca_{group}.png:各グループごとのプロット
実行例
入力ファイルとして、example.eigenvecとexample_population.txtを使用してスクリプトを実行した場合、以下の画像が得られる。