Bioinfo系の仕事をするにはMacOSかLinuxがやっぱり便利です。Windowsでも頑張れるんですが、pathのback slashがescapeプラス一文字と解釈されたりしてイヤですし、なんちゃってpermissionもたよりない。しかし、Windows 10でもできます、local blast。今回はそのお話です。
インストール
NCBIのftpサイトからインストーラーをダウンロードします。なんかたくさんあって、これかなと思える"win64"がついているものだけでもいくつもある。このあたりから不安になってしまうわけですが、
ncbi-blast-2.10.0+-win64.exe
をとりましょう。もう一つのほう、
ncbi-blast-2.10.0+-x64-win64.tar.gz
でもいいんです。*.tar.gzの展開がすぐできるなら。えっと、*.tar.gzの展開ってどうやるんだっけ、とgoogleするなら、.exeのほうを取りましょう。どちらのサイズもほとんど変わりませんし。
新しいバージョンがある場合には 2.10.0 の部分を読み替えてください。
さて、ファイルが取れてきましたら、ちょっと待って。ダブルクリックや展開の前に、正しいものが取れたか、ファイルが壊れたりしていないか、確認してみましょう。
ftpサイトに、今とったファイルの名前に.md5がついたファイルがあります。
ncbi-blast-2.10.0+-win64.exe.md5
または、
ncbi-blast-2.10.0+-x64-win64.tar.gz.md5
自分がとったほうのファイルに.md5がついたものを取ります。
中身はエディタで開くか、lessコマンドを使ってみ見てみると、
bc87568310e0878db73e177c15025d9c ncbi-blast-2.10.0+-win64.exe
あるいは
3f3ef65682be57bc3d2a39b25f1625a0 ncbi-blast-2.10.0+-x64-win64.tar.gz
となっています。左半分がその右にあるファイルのhash値です。チェックしてみましょう。
コマンドラインを開き、とったファイルに応じて、
certutil -hashfile ncbi-blast-2.10.0+-win64.exe md5
あるいは
certutil -hashfile ncbi-blast-2.10.0+-x64-win64.tar.gz md5
でmd5のhash値が得られます。先の.md5ファイルにあったものと一致していればファイルは壊れていません。もし違う場合にはもう一度ダウンロードしてみましょう。
上のcertutilは、md5のところをSHA256に変えればSHA256のhash値が得られます。SHA256のhash確認ファイルが配られている場合にもこのコマンドを使うことができます。
さて、インストールです。
.exeのほうは簡単で、ダブルクリックで一発、かと思いきや、
Windows protected your PCというメッセージが出ます。More Infoというのをクリックすると、あっさり"Run anyway"ボタンが登場して、先へ進めます。同意して、インストール場所を指定するだけ(upper pathが簡単には変わらないところを選びましょう。デフォルトではProgram Filesの下です)。このイントール場所、しっかり見て覚えておいてください。(search) pathを通すときに必要になります。
ちなみに、.tar.gzのほうをとった場合には、
tar zxvf ncbi-blast-2.10.0+-x64-win64.tar.gz
で展開できて、
ncbi-blast-2.10.0+
というフォルダができてきます。このフォルダをどこか好きな場所(ただし、そうそう簡単にupper pathが変わらないところ)に置きましょう。
pathが深すぎるとpath長すぎ、のようなエラーが出たり、日本語がpathの途中に含まれたり、スペースを含むdirectory(folder)があったりすると、本質的ではないエラーに悩まされたりすることがありますので、C:の直下、あるいはC:\bioとか、C:\localのようなフォルダを作ってそこをツール置き場にするのもいいかもしれません。
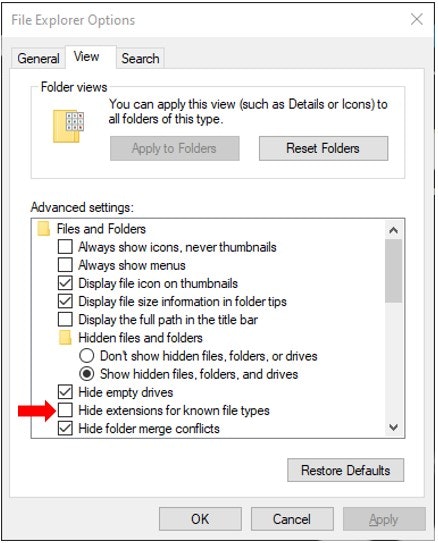
ちなみに、さっきから.exeとか、.tar.gzとか書いていますが、え?私のダウンロードファイルには.exeがない、あるいは.gzがない、という方は、表示の設定を変えましょう。拡張子は大切な情報です。Windowsはデフォルトでいくつかの拡張子を非表示にしてしまいます。困ります。こうします。
File Explorer Option(フォルダオプション)を開いて、
Hide extensions for known file types
のチェックを外してApply。
これで.exe、.gzが姿を現すはずです。
ここまでをまとめますと、インストールの結果はこうなっています。
.exeを使った場合
あなたがインストーラー実行中に選んだ場所/NCBI/blast-2.10.0+/bin --- (1)
の中にコマンド群があります。
.tar.gzを展開した場合
あなたがフォルダを置いた場所/ncbi-blast-2.10.0+/bin --- (2)
にコマンド群があります。
(コマンド群のありかへ)pathを通す
blastのコマンド群は、コマンドラインから使うもので、できれば、
blastn
と打つだけで使えるようにしたい。実際blastn.exeは、上の(1)か(2)にありますので、いまのままでは、
あなたがインストーラー実行中に選んだ場所/NCBI/blast-2.10.0+/bin/blastn
か、
あなたがフォルダを置いた場所/ncbi-blast-2.10.0+/bin/blastn
と打たなければ使えない。長い。コピペでこれを書くだけでも面倒です。
そこで、私のコマンドは、ここにある。いわなくてもまずはそこを探してください、を登録します。この登録しておく行為をパスを通す、と言います。
MacOSやLinuxでは.bashrc、.bash_profileなどに記載しますが、Windows 10ではこうやります。
System Properties(システムのプロパティ)> Advanced(詳細設定)tabを選び、Environment Variables(環境変数)ボタンをクリックして設定ウインドウを表示させます。User variablesのほうにあるPathをクリックして選択し、Edit(編集)ボタンをクリックします。
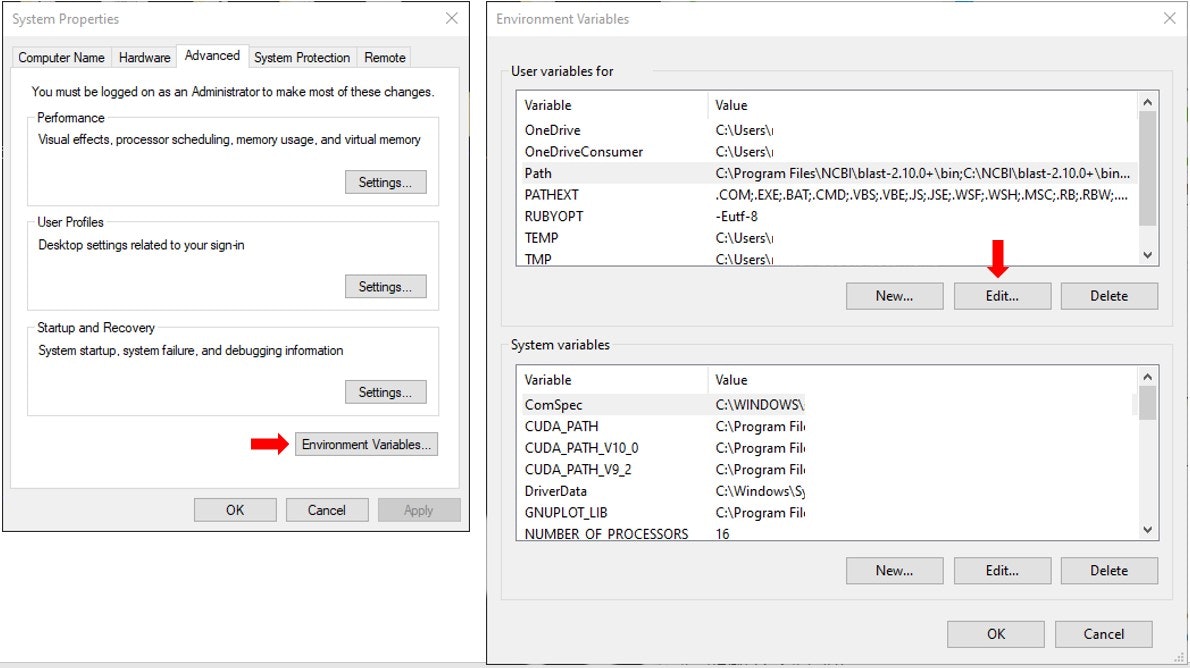
Edit environment variable(環境変数名の編集)ウインドウが開きます。New(新規)をクリックします。
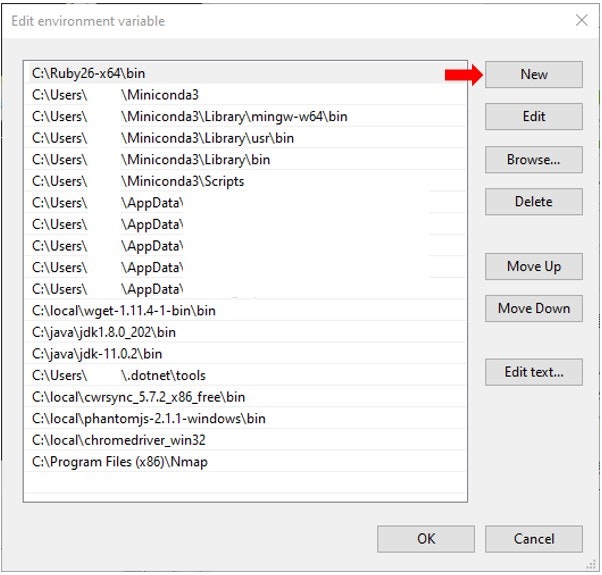
新しいパスの入力待ちになります。ここに登録したい(今回なら)binまでのパスを入力することになります。

さて、ここで、先ほどの(1)か(2)のbinフォルダを探し出して開いてください。このbinフォルダまでの(絶対)パスを取りたい。単純な場合は打てると思うんですが、単純でもC:のあとback slashいるんだっけ?とか思ってしまう。そういう場合には、下に示します、なんというんでしょうか、アドレスバー?のところの、binを右クリックして、copy address as textを選ぶと、binまでのabsolute path(絶対パス、Cからそこまでのパス)をコピーすることができます。
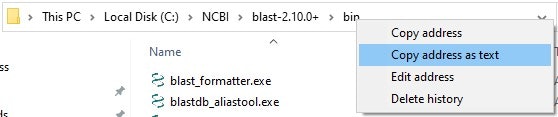
Edit Environment Variablesウインドウに戻って、たぶん、入力待ちだったところが消えてしまっていると思いますので、"New"をもう一度クリック。今コピーしてきたpathをペーストしてOKを押します。
これでpathの設定が完了しました。コマンドラインのウインドウを閉じて、新しく開きなおし(こうしないと新しいパス設定が読み込まれません)、次のようにタイプしてみます。
which blastn
あなたがインストーラー実行中に選んだ場所/NCBI/blast-2.10.0+/bin/blastn
か、
あなたがフォルダを置いた場所/ncbi-blast-2.10.0+/bin/blastn
のように表示されればインストールおよびpath通しが完了です。
使い方(blastn)
たいていは、インストール方法の紹介で終わるところなんですが、その先なんですよね、知りたいのは。しかもWindowsだと、MacOSやLinuxでは出ないエラーもでたりして、挫折しやすいんです。それでは行きましょう。
(基本はシンプルです。解析に必要なデータベースを作って、queryを用意して、blastn。走らせること自体は難しくありません。その後の解析、解釈が難しい。目的に応じたparameter設定も必要となる。でも、それは研究や目的の文脈によりますから、ここでは触れません。)
makeblastdb: データベースを作る
これです。作るんです。出来合いのモノを取ってくることもできますが、自分でデータベースを作ることができるところにlocalでやる意味があるんです。やってみましょう。
まずは、データベースに入れる配列たちを用意する必要があります。これは目的に応じて用意することになりますが、例を取り上げてみます。例えば、今(2020年3月)ならば、Coronavirusを取り上げるのはいいかもしれません。次の7つを取ってきてみましょう。
- SARS-CoV-2: NC_045512.2 (これがいま問題になっているものです)
- SARS Coronavirus: NC_004718.3(いわゆるSARS)
- MERS Coronavirus (England-1): NC_038294.1
- Human Coronavirus OC43: NC_006213.1
- Human Coronavirus HKU1: NC_006577.2
- Human Coronavirus 229E: NC_002645.1
- Human Coronavirus NL63: NC_005831.2
NCBIのページで上のaccessionを使えばすぐに到達できるはずです。fasta形式でファイルをとってきて、それぞれにわかりやすく名前を付けてください。作業用のdirを一つ作って(example)その中に保存した例が下。コマンドでの説明が一番なので、たどってみてください。
mkdir example
cd example
# fastaを取ってきたあとlsするとこうなっています。
ls
NC_002645.1.fa NC_005831.2.fa NC_006577.2.fa NC_045512.2.fa
NC_004718.3.fa NC_006213.1.fa NC_038294.1.fa
# 7つのファイルを一つにまとめておきます
cat *.fa > cov_7.mfa
# grepでid lineを見てみましょう。
grep ">" cov_7.mfa
>NC_002645.1 Human coronavirus 229E, complete genome
>NC_004718.3 SARS coronavirus, complete genome
>NC_005831.2 Human Coronavirus NL63, complete genome
>NC_006213.1 Human coronavirus OC43 strain ATCC VR-759, complete genome
>NC_006577.2 Human coronavirus HKU1, complete genome
>NC_038294.1 Betacoronavirus England 1, complete genome
>NC_045512.2 Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome
このように、データベースに格納する配列は、一つのfasta fileにまとめておくのが便利です。後々記録にもなりますし。もっと数がある場合には、
grep -c ">"あるいは、id lineが一つ前の配列の後についてしまうような失敗もあるかもしれないので、grep -c "^>"を使って、あるべき配列数がファイルの中にあることを確認しておきましょう。このあたり、面倒がらずにやる心が大切です。
格納する配列レコードの用意ができましたので、makeblastdbコマンドでblast databaseを作ります。いくつかのoptionを指定しますので、長ーいコマンドになります。まずは丁寧にタイプしてもいいのですが、ちょっとした間違いや、もう一度やり直したいというときに苦労します。
そこで、.shファイルにコマンドを書いて、それを実行することにします。こうすると、記録になりますし、少し変更して繰り返したり、誰かに渡して同様のことをしてもらうことも簡単です。
# いま、先ほど作ったexample dirにいることとします
# cov_7.mfaがほかのfasta fileとともにcurrent dirにある状態です。
# dirを作って整理しておきましょう
mkdir fasta_files
mv *.fa ./fasta_files
mv cov_7.mfa ./fasta_files
# 始めます
mkdir blast_db
cd blast_db
# makeblastdb_command.shを作ります。別名でもいいですよ。
touch makeblastdb_command.sh
# このファイルをエディタで編集します。
makeblastdb_command.shに以下の内容を記載して保存してください。最初はコピペせずに打ちましょう。そのほうが記憶に残ります。
# construct blast database
makeblastdb -in ../fasta_files/cov_7.mfa \
-dbtype nucl \
-parse_seqids \
-out corona_7 \
-title "7 Coronavirus sequences from NCBI" \
-logfile corona_7.log.txt \
-hash_index
説明です。
各行の最後にあるバックスラッシュ\は、見やすさのため改行しますが、コマンドとしての行はまだ続きますよ、というサインです。このあとにコメントを入れたりしないでください。"Too many positional arguments..."エラーがでます。やってみてください。たびたび出会うエラーですので。一度出会っておくと、あ、あれかな、とすぐに解決できるようになります。
makeblastdb -in ../fasta_files/cov_7.mfa
blast databaseを作るためのコマンド。-inオプションでinput fastaへのパスを指定します。
-dbtype nucl:今回は核酸の配列なので、argumentにnuclを指定します。
-parse_seqIds:これはargumentなしのオプション。
-out corona_7:これがデータベース名になります。短く、スペースなしで。
-title "7 Coronavirus sequences from NCBI":データベースのタイトル、ちょっとしたメモです。短めにかつ分かりやすくつけます。スペースが入ることになりますので、double quotesで挟むのを忘れずに。
logfile corona_7.log.txt:データベース構築のログを保存するファイル。私は-outのところで使ったデータベース名に.log.txtとつけています。ご自分でnaming ruleを作ってください。この作業は通常何度か行いますし、同じdirectoryにいくつものdatabaseを入れておくことにもなるかと思います。単にlogなどとしておくと書き換えが起こってしまうかもしれません(上書きされるかな?知りません、ごめんなさい)
hash_index:これもargumentなしのオプションです。
makeblastdb_command.shが用意できましたら、
bash makeblastdb_command.sh
で、データベースができてきます。
のはずなのに!
失敗しました。logファイルを開けてみると、
Building a new DB, current time: 03/01/2020 12:03:40
New DB name: C:\Users\xxxxxx\Desktop\example\blast_db\corona_7
New DB title: 7 Coronavirus sequences from NCBI
Sequence type: Nucleotide
Keep MBits: T
Maximum file size: 1000000000B
No volumes were created.
Error: mdb_env_open: There is not enough space on the disk.
だそうですが、今どきdisk spaceが不十分って何なんですかね。ここで心配になっていろいろごみ箱に捨てたり外部ディスクにファイルを移したりしないでください。次のようにすると解決します。
参考ページ:このページの一番最後
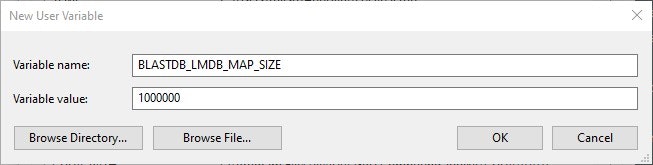
1つ新しい環境変数を作って値を指定するっていうことをします。
上でpathの設定をしたときに使った、Environment variables(環境変数)ウインドウを開きます。今回はNew(新規)ボタンをクリックします。
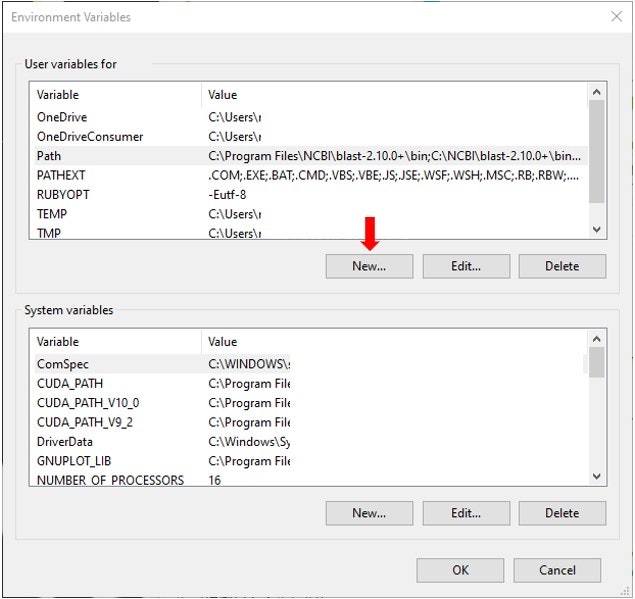
次のような小さなウインドウが表示されますので、下にあるように入力し、OKボタンをクリックします。二つ目は、ゼロ6個で。
コマンドラインウインドウをいったん閉じて、もう一度開き、example/blast_dbにcdして(cdするって変ですが、よくこういいます)、
bash makeblastdb_command.sh
どうですか?
今回はうまくいくはずです。まだ同じエラーがでますか?すみません、これ以上はお手伝いできませんので、なんとか調べて突破してください。
うまくいきますと、次のようなファイルたちができてきているはずです。
ls
corona_7.log.txt corona_7.nhi corona_7.nos corona_7.nto
corona_7.log.txt.perf corona_7.nhr corona_7.not makeblastdb_command.sh
corona_7.ndb corona_7.nin corona_7.nsq
corona_7.nhd corona_7.nog corona_7.ntf
logを開いてみると、先ほどのエラーの記録に続いて、今回のうまくいったログが記録されているはずです。
データベースができましたので、一つ上の階層(example)に移動しておきましょう。
cd ../
blastnを走らせる
いよいよです。まず、queryを用意しましょう。
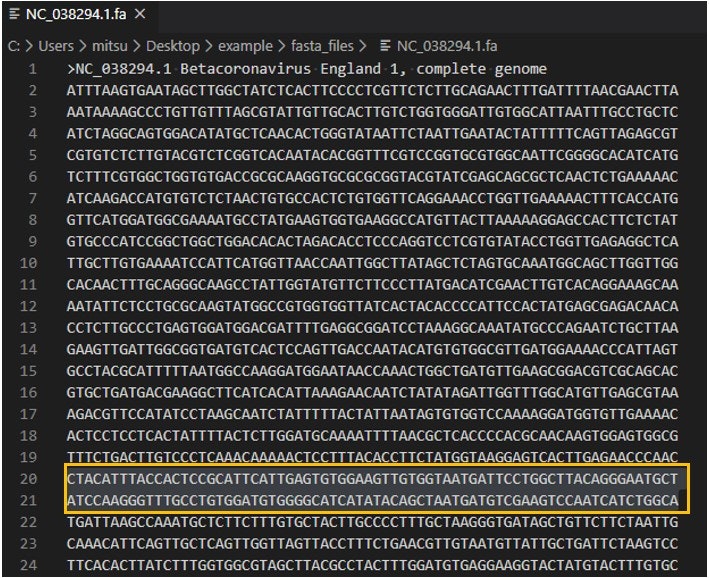
なんでもいいんですが、今回コロナウイルスの代表的7つをデータベースに入れていますので、そうですね、MERSの配列の一部をテキトーに切ってきて、それを使ってみましょう。
MERSの配列は、NC_038294.1.faにありますので、このファイルをエディタなどで開き、なんの意図もないので、あてずっぽうですが、ためしに20-21行目をコピーして、
下のような内容の、MERS_L20-21.faを作ってexample直下に保存してください。
>MERS_L20-21
CTACATTTACCACTCCGCATTCATTGAGTGTGGAAGTTGTGGTAATGATTCCTGGCTTACAGGGAATGCT
ATCCAAGGGTTTGCCTGTGGATGTGGGGCATCATATACAGCTAATGATGTCGAAGTCCAATCATCTGGCA
これをqueryとして使いましょう。
makeblastdbの時と同じく、blastnコマンドも複数のオプションをとり、一行で書いていくと非常に読みづらくなりますので、ファイルに保存してそれを実行することにしましょう。
run_blastn.shというファイルを用意し、以下の内容でexample直下に保存してください。
# run blast with format 0
blastn -query MERS_L20-21.fa \
-db ./blast_db/corona_7 \
-task blastn \
-outfmt 0 \
-out MERS_L20-21-fmt0.txt
説明です。
-query:query fileへのpathを書きます。
-db:先ほど作ったblast dbへのpathなのですが、拡張子より手前の部分のみ書きます。
-task:ここはblastn, megablast, blastn-shortから選ぶことができます。
-outfmt 0:今回0を指定してみます。あとでもう少し説明します。
-out:出力先。このオプションを省略するとSTDOUTへ結果が流れます。リダイレクト>を使ってファイルに保存しても同じことができますね。
さあ、走らせましょう。exampleがcurrent directoryであることを確認して、
bash run_blastn.sh
current directoryにMERS_L20-21-fmt0.txtが一瞬でできてきます。すごいですね。
結果を見てみましょう。
makeblastdbコマンドのところで指定した、database titleが見えます。こんな感じでtitleが表示されますので、databaseを作るときの-titleオプションargumentにはわかりやすいものを与えましょう。
Database: 7 Coronavirus sequences from NCBI
7 sequences; 205,302 total letters
Query= MERS_L20-21
Length=140
Score E
Sequences producing significant alignments: (Bits) Value
NC_038294.1 Betacoronavirus England 1, complete genome 253 1e-69
NC_006577.2 Human coronavirus HKU1, complete genome 25.6 0.90
NC_006213.1 Human coronavirus OC43 strain ATCC VR-759, complete g... 23.8 3.1
NC_004718.3 SARS coronavirus, complete genome 22.9 3.1
テキトーに切り取ったとはいえ、MERSの配列をqueryにしましたので、当然ですが、MERSに対してばっちりあたってきます。実験でいえば、positive controlですね。local blastではこういうものをきちんと含んで解析することが大切です。
>NC_038294.1 Betacoronavirus England 1, complete genome
Length=30111
Score = 253 bits (280), Expect = 1e-69
Identities = 140/140 (100%), Gaps = 0/140 (0%)
Strand=Plus/Plus
Query 1 CTACATTTACCACTCCGCATTCATTGAGTGTGGAAGTTGTGGTAATGATTCCTGGCTTAC 60
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1261 CTACATTTACCACTCCGCATTCATTGAGTGTGGAAGTTGTGGTAATGATTCCTGGCTTAC 1320
Query 61 AGGGAATGCTATCCAAGGGTTTGCCTGTGGATGTGGGGCATCATATACAGCTAATGATGT 120
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1321 AGGGAATGCTATCCAAGGGTTTGCCTGTGGATGTGGGGCATCATATACAGCTAATGATGT 1380
Query 121 CGAAGTCCAATCATCTGGCA 140
||||||||||||||||||||
Sbjct 1381 CGAAGTCCAATCATCTGGCA 1400
他はHKU1, OC43, SARSに対して短くしかあたってきませんでした。テキトーに切ったqueryでしたが、ここで面白いことに気が付きます。
databaseには7つの配列を入れた。一つには大当たり(当たりまえ)、あとの3つに少しだけ相同領域がある。(queryは140 base, hit stretchは100-140のあたり)
あれ?登場しなかった残りの3つは?
一つは(いま広がっている)SARS-CoV-2、後の二つは229E, NL63というstrainです。実は、今回データベースに入れた7つのstrainのうち、229EとNL63がalpha coronavirus、残りはbeta cronavirusというグループに区分されます。betaであるMERSの一部を使ってblastしたら、alphaはあたらず、betaではSARS-CoV-2があたってこなかった。だから、、、
こんな感じで解析結果を見ていろいろ考えます。blastはその名の通り、Basic Loacal Alignment Search Toolで、toolです。解釈は利用者がするものなので、blastでhitがなかったから大丈夫、とか、blastでhitしたから大丈夫、とかは文脈によりますし、よくよく考えないと勘違いします。
>NC_006577.2 Human coronavirus HKU1, complete genome
Length=29926
Score = 25.6 bits (27), Expect = 0.90
Identities = 15/16 (94%), Gaps = 0/16 (0%)
Strand=Plus/Plus
Query 123 AAGTCCAATCATCTGG 138
||| ||||||||||||
Sbjct 1401 AAGCCCAATCATCTGG 1416
>NC_006213.1 Human coronavirus OC43 strain ATCC VR-759, complete genome
Length=30741
Score = 23.8 bits (25), Expect = 3.1
Identities = 14/15 (93%), Gaps = 0/15 (0%)
Strand=Plus/Minus
Query 106 TACAGCTAATGATGT 120
||||||||||| |||
Sbjct 20779 TACAGCTAATGTTGT 20765
>NC_004718.3 SARS coronavirus, complete genome
Length=29751
Score = 22.9 bits (24), Expect = 3.1
Identities = 12/12 (100%), Gaps = 0/12 (0%)
Strand=Plus/Minus
Query 129 AATCATCTGGCA 140
||||||||||||
Sbjct 22737 AATCATCTGGCA 22726
余計な講釈たれるのはやめにして、output formatについて少し続けましょう。
先ほどのrun_blastn.shの-outfmtと-outを次のように変更して走らせてみてください。
-outfmt 3 \
-out MERS_L20-21-fmt3.txt
アラインメントの表示のされかたがずいぶん変わりました。
Query_1 1 CTACATTTACCACTCCGCATTCATTGAGTGTGGAAGTTGTGGTAATGATTCCTGGCTTAC 60
NC_038294.1 1261 ............................................................ 1320
Query_1 61 AGGGAATGCTATCCAAGGGTTTGCCTGTGGATGTGGGGCATCATATACAGCTAATGATGT 120
NC_038294.1 1321 ............................................................ 1380
NC_006213.1 20779 ...........T... 20765
Query_1 121 CGAAGTCCAATCATCTGGCA 140
NC_038294.1 1381 .................... 1400
NC_006577.2 1401 ...C............ 1416
NC_004718.3 22737 ............ 22726
先ほどのformat 0はpairwiseと呼ばれるもので、webでのデフォルト表示です。format 3は、flat query-anchored, show identitiesという設定です。一致したbaseをドットで表し、queryとは異なるところのみbaseが表示されます。これは異なるところをハイライトしたいときに便利な表示フォーマットです。
さらに、format 7ではテーブル表示が得られます。hitがいくつあったのか、query, subjectのhit start, hit endを取得してさらに次の解析に持ち込みたいときに便利なフォーマットです。同様にrun_blastn.shを変更して試してみてください。
blastdbcmdもやっぱり
databaseを作る目的はもちろんblastnを走らせるためなのですが、もう一つ便利なdatabaseの使い道があります。
blastdbcmdというコマンドを使うと、database中の配列を非常に便利に取り出すことができるのです。これは使わない手はありません。紹介させてください。
下に使い方の例を示します。
blastdbcmd -entry NC_038294.1 \
-range 1-10 \
-strand minus \
-db ./blast_db/corona_7 \
-outfmt %f
-db:data baseへのpath
-entry:取りたい配列のaccession id
-range:start-end
-strand:plus or minus
-out:出力ファイル。省略すればSTDOUTへ出ます
outfmt:%fはfasta
試しにNC_028294.1の1から10 baseを取ってみて、うまく取れているか確認するためのコマンドです。strand指定をminusにしたりして試してみてください。
おわりに
makeblastdb, blastn, blastdbcmdを紹介しました。オプションやargumentについては、それぞれのコマンドに-helpをつけてヘルプを参照してください。いろいろ発見があると思います。
また、本家のマニュアルも時間のある時に覗いてみてください。