はじめに
生存時間分析について学んでいくと、様々なアルゴリズムがあるのだなと非常に感心しています。
こちらのサーベイ論文では次のようにまとめられていました。
https://dmkd.cs.vt.edu/papers/CSUR19.pdf
今回は、パラメトリックの生存時間分析からブースティングによる拡張したモデルまでの様々なアルゴリズムについてをまとめます。
また、Rでの利用方法もまとめました。
※細部は結構雑です。中途半端な部分は加筆していきます。
生存時間分析
生存時間分析とは、イベントが発生するまでの時間とイベントとの間の関係に着目した分析です。
例えば、サービスのユーザーの離脱や、投薬後の患者の死亡の発生、機械の故障の発生といったもので扱われます。
生存関数とハザード関数
生存時間$t$を確率変数とした、確率密度関数$f(t)$を次のように表す。
f(t)=\lim_{\Delta t →0} \frac{Pr(t<T<t+\Delta t)}{\Delta t}
時刻$t$までにイベントが発生していないことを表す生存関数S(t)は次のような関係となる。
S(t)=Pr(T>t)=\int_t^{\infty}f(t)dt=1-F(t)
ここで、$F(t)$は累積確率密度関数である。
イベントがある時点t までに発生していないという条件の下で,次の瞬間にイベントが発生する瞬間死亡率を示すハザード関数は次のように表す。
h(t)=\left\{
\begin{array}{ll}
\lim_{\Delta t →0} \frac{Pr(t<T<t+\Delta t|T \geq t)}{\Delta t}= \frac{f(t)} {S(t)} (連続)\\
Pr(T=t|T\geq t)=\frac{f(t)}{S(t-1)} (離散)
\end{array}
\right.
ハザード関数と生存関数、そして累積ハザード関数の関係は次のようになる。
H(t)=\int_0^t h(t) dt = -log S(t)
また、ハザード関数と生存関数は次のような関係も存在する。
h(t)=- \frac{S(t)'}{S(t)}
打ち切り
イベントが発生するまでの観測を行う場合、イベントの発生によりデータが得られなくなる場合とイベントが発生せずに観測が終了になりデータが得られなくなる場合は別の現象として捉える必要がある。
そのため、イベントが発生せずに観測が終了する場合を打ち切りと呼ばれる情報として、イベント発生までの時間とともに生存時間分析に必要な情報となる。
なお、このような観測終了時に関する打ち切りを右側打ち切りと呼びます。
そのほかにも、観測開始時に関する打ち切りを左側打ち切り、特定区間内に打ち切りがあったかどうかだけわかる場合は区間打ち切り、右側左側共に打ち切りを考えるが場合は両側打ち切りと呼ばれている。
RではsurvivalパッケージのSurv関数を用いることで、打ち切り情報を付加したイベント発生までの時刻情報を形成することができる。
> library(survival)
> data("kidney")
> kidney %>% head
id time status age sex disease frail
1 1 8 1 28 1 Other 2.3
2 1 16 1 28 1 Other 2.3
3 2 23 1 48 2 GN 1.9
4 2 13 0 48 2 GN 1.9
5 3 22 1 32 1 Other 1.2
6 3 28 1 32 1 Other 1.2
> kidney.surv <- Surv(kidney$time,kidney$status)
> head(kidney.surv)
[1] 8 16 23 13+ 22 28
数字の後に+がついているものが、打ち切りがなかった(イベントが発生した)という情報になり、その他の無印は打ち切りあった (イベントが発生しなかった)ことを意味しています。
生存時間分析の主な種類
パラメトリック:ハザード関数に分布を仮定し、説明変数を用いない
セミパラメトリック:ハザード関数に分布を仮定せず、説明変数を用いる
ノンパラメトリック:ハザード関数に分布を仮定せず、説明変数も用いない
ノンパラメトリックモデル
確率分布を仮定せずに生存時間を推定する方法で、経験分布による推定法とハザード関数による推定法がある。
打ち切りがないデータであれば、生存関数は収集したデータから得られた累積分布関数$F(t)$を用いて推定する。
\hat S(t)=1-F(t)
打ち切りがある場合には、次のような定義により算出されるカプラン-マイヤー推定法により求める。
\hat S(t) = \prod_{t_i<t}(1-\frac{d_i}{r_i})
ここで、$d_i$は時刻$t_i$での死亡数、$r_i$は時刻$t_i-1$でイベントが発生する可能性がある観測対象の数である。
Rではsurvival::survfitにより求めることができる。
また、カプラン-マイヤー推定法により推定した生存関数より累積ハザード関数を求めることができる。
この推定量を、ネルソン推定量と呼ぶ。
\hat H(t)=-log \hat S(t) = \sum_{t_i<=t} \frac{d_i}{r_i}
パラメトリックモデル
生存関数に対して確率分布を仮定し当てはめます。
用いられる代表的な分布は、ワイブル分布と指数分布です。
ワイブル分布
生存関数と密度関数は次で表すことができる。
S(t)=e^{-(\lambda t)^p} f(t)=-\lambda p (\lambda t)^{p-1} e^{-(\lambda t)^p}
指数分布
生存関数と密度関数は次で表すことができる。
S(t)=e^{-\lambda t} f(t)=-\lambda e^{-\lambda t}
それほかの代表的な分布は次のようなものです。

<[引用元](https://www.slideshare.net/ChandanReddy4/machine-learning-for-survival-analysis)>
セミパラメトリックモデル
Cox比例ハザードモデル
説明変数がイベントまでの生存時間に影響を与えると考えるモデルです。
2つのハザード関数$h_a(t)$、$h_b(t)$が、$t>0$に対して次のように関係式2つのハザード関数は比例する場合を考える。
h_a (t ) = Ch_b (t )
ここで、Cは時間に寄らない比例定数である。
このような場合、あるハザード関数に対して、共変量$x$を条件づけた場合のハザード関数$h(t|x)$を次のように表す。
h(t|x)=h_0(t)exp(\beta x)
ここで、$h_0(t)$はベースラインハザード (baseline hazard) と呼び、共変量が全て0の場合のハザード関数である。
ベースラインハザードは、カプランマイヤー法を用いて推定する。
パラメーター$\beta$は次のような条件付き確率尤度関数$L$を最大にすることで推定する。
L(\beta)=\prod_{i=1}^M \frac{exp(\beta X_i)}{\sum_{k \in R(t_i)} exp(\beta X_k)}
ここで、$M$はイベントの数、$t_i$はイベント$i$の発生時間、$R(t_i)$は時刻$t$までにイベントが発生していない観測対象の集合である。
このようにベースラインハザードはノンパラメトリック、線形予測子部分はパラメトリックであるため、これらの掛け合わせであられるモデルであるためCox比例ハザードモデルはセミパラメトリックモデルと呼ばれている。
また、ある説明変数$x_i$において処置なしを$x_i=0$,処置ありを$x_i^*=1$として、そのほかの説明変数は変化しないと仮定する。
この時のハザードの比は次のようになる、
\frac{h(t|x_i^*,x_1,\dots,x_n)}{h(t|x_i,x_1,\dots,x_n)} =\frac{h_0(t)exp(\beta_1 x_1 + \dots + \beta_i x_i^* +\dots +\beta_n x_n)}{h_0(t)exp(\beta_1 x_1 + \dots + \beta_i x_i + \dots + \beta_n x_n)}=\frac{exp(\beta_i x_i^*)} {exp(\beta_i x_i)}=exp(\beta_i)
これは、ハザード比と呼ばれ、処置によるイベント発生への影響を表している。
この場合、処理を行うことによりイベントの発生確率が$exp(\beta_i)$倍になるといえる。
この時、ハザード比は共変量のみに依存し、時間依存しないという仮定のもとに成り立つことを注意しなければならない。
この仮定を比例ハザード性を呼ぶ。
survivalパッケージを利用してCox比例ハザードモデルをお試しです。
> library(MASS)
> library(survival)
> data("kidney")
> kidney.cox<-coxph(Surv(time, status) ~ sex+age+disease+frail, data=kidney) %>% stepAIC()
> summary(kidney.cox)
Call:
coxph(formula = Surv(time, status) ~ sex + age + disease + frail,
data = kidney)
n= 76, number of events= 58
coef exp(coef) se(coef) z Pr(>|z|)
sex -2.099844 0.122475 0.392654 -5.348 8.90e-08 ***
age 0.007714 1.007744 0.011907 0.648 0.517055
diseaseGN 0.130666 1.139587 0.436114 0.300 0.764471
diseaseAN 0.640906 1.898200 0.447886 1.431 0.152442
diseasePKD -2.168515 0.114347 0.648825 -3.342 0.000831 ***
frail 1.791873 6.000682 0.257639 6.955 3.53e-12 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
exp(coef) exp(-coef) lower .95 upper .95
sex 0.1225 8.1649 0.05673 0.2644
age 1.0077 0.9923 0.98450 1.0315
diseaseGN 1.1396 0.8775 0.48476 2.6790
diseaseAN 1.8982 0.5268 0.78904 4.5665
diseasePKD 0.1143 8.7453 0.03206 0.4079
frail 6.0007 0.1666 3.62158 9.9427
Concordance= 0.822 (se = 0.046 )
Rsquare= 0.595 (max possible= 0.993 )
Likelihood ratio test= 68.71 on 6 df, p=8e-13
Wald test = 60.01 on 6 df, p=4e-11
Score (logrank) test = 86.24 on 6 df, p=<2e-16
exp(coef)がハザード比であり、frailが1増えるとイベントが発生する確率が6倍になると言えます。
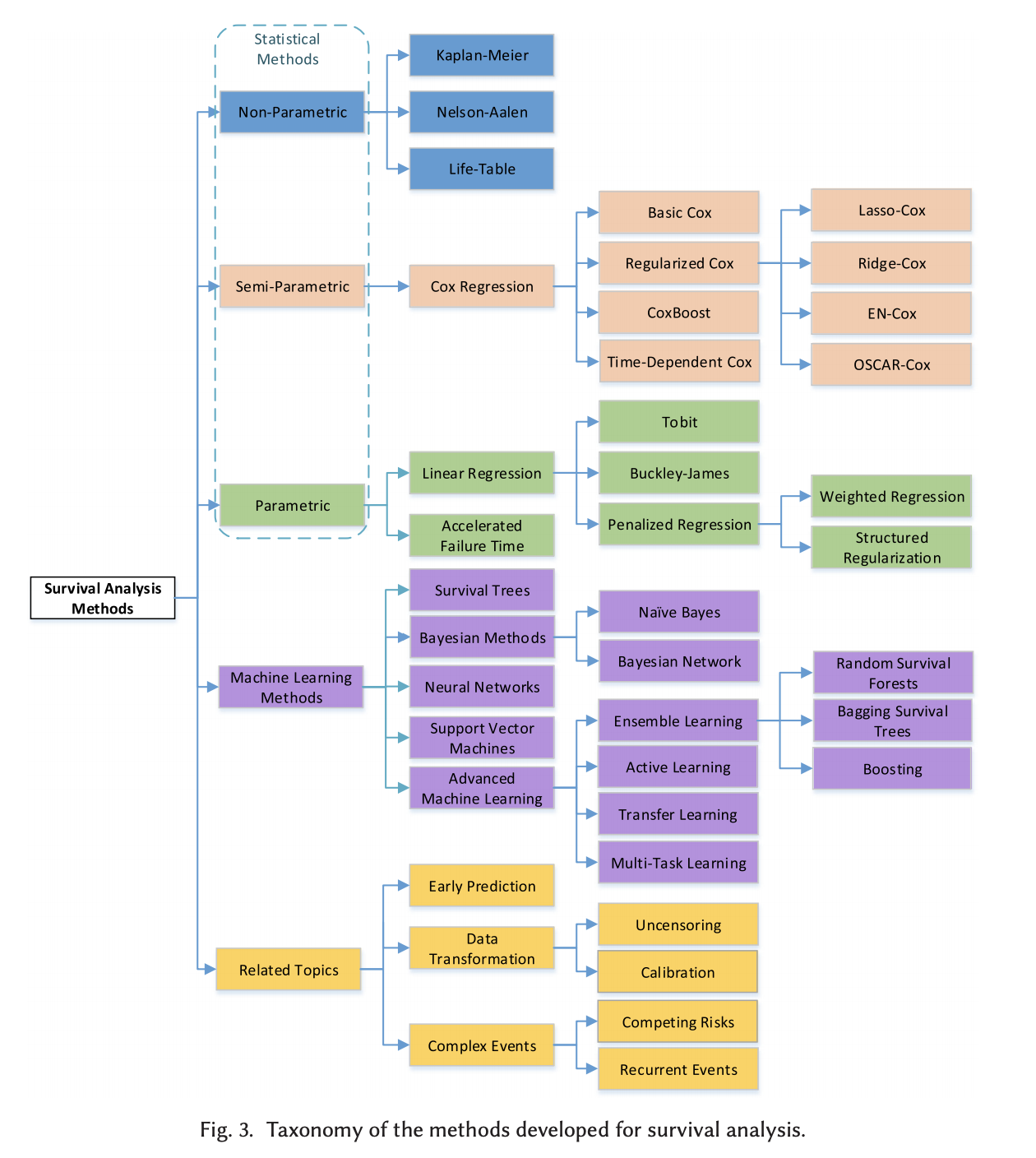
生存関数をplotしてみます。
kidney.fit<-survfit(kidney.cox)
plot(kidney.fit, main="Survival curve")
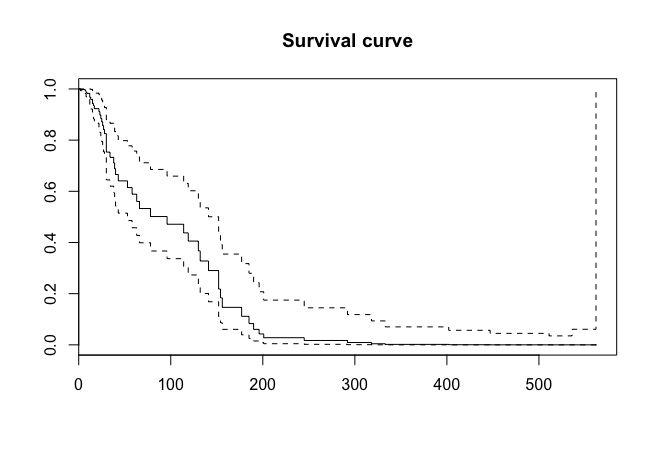
次にハザード比は時間変化しないという仮定を確認します。
p値が小さいほど、要素の影響が時間変化していると言えます。
> kidney.coxzph <- cox.zph(kidney.cox)
> print(kidney.coxzph)
rho chisq p
sex -0.04674 0.1476 0.7008
diseaseGN 0.00299 0.0005 0.9822
diseaseAN 0.06314 0.2142 0.6435
diseasePKD -0.14900 1.3569 0.2441
frail 0.26471 4.5880 0.0322
> par(mfrow=c(2, 3))
> plot(kidney.coxzph, df=2)
frailはp値とグラフから生存時間への影響が時間変化してしまっていると判断できます。
スパース cox回帰モデル
線型予測子の部分は、一般的な回帰分析と同様に対応する目的関数に正則化項を加えることで、スパース推定を行うことができます。
これにより、説明変数の数>サンプル数の多次元小標本の状態でもモデルを求めることが出来ます。
スパースモデリングの一般的な知識は、これらのサイト[1,2]から参照してください。
Lasso Cox 回帰
目的関数にL1正則化項を加えた回帰
変数選択を行うことができる。選択される変数は最大$n$である。
Ridge Cox 回帰
目的関数にL2正則化項を加えた回帰
変数選択は行うことが出来ない。
Elastic Net Cox 回帰
目的関数にL1,L2正則化項の両方を加えた回帰
変数選択を行うことができ、選択される変数は最大$p$となる。
また、選択される説明変数の一意性が生まれることになる。
これらのモデルは glmnet パッケージと fastCox パッケージで利用することができます。
個人的にはglmnetの方が、普通の回帰モデルと同様に扱うことができるので好みです。
> kidney.dummy <- kidney %>%
fastDummies::dummy_cols( select_columns = c("disease")) %>%
dplyr::select(age,sex,frail, starts_with("disease_")) %>%
as.matrix()
> kidney.Y = Surv(kidney$time,kidney$status) %>% as.matrix()
> lasso.model <- cv.glmnet(x = kidney.dummy, y = kidney.Y, family = "cox", alpha = 1)
> coef(lasso.model, s=lasso.model$lambda.min)
7 x 1 sparse Matrix of class "dgCMatrix"
1
age 0.007367912
sex -2.002408971
disease.Other -0.121695813
disease.GN .
disease.AN 0.476045680
disease.PKD -2.200370579
frail 1.717562319
> #ハザード比の算出
> coef(lasso.model, s=lasso.model$lambda.min) %>% exp()
7 x 1 Matrix of class "dgeMatrix"
1
age 1.0073951
sex 0.1350097
disease.Other 0.8854177
disease.GN 1.0000000
disease.AN 1.6096965
disease.PKD 0.1107621
frail 5.5709317
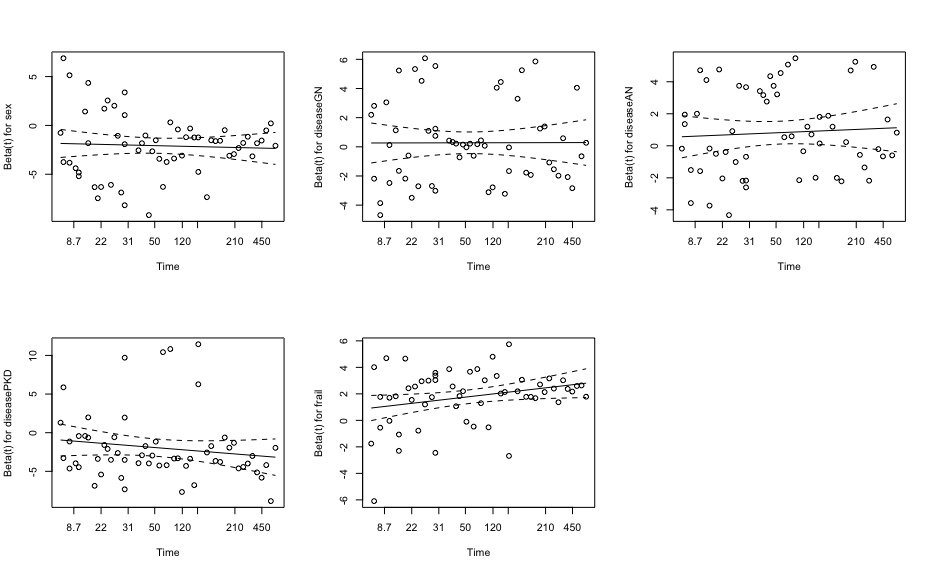
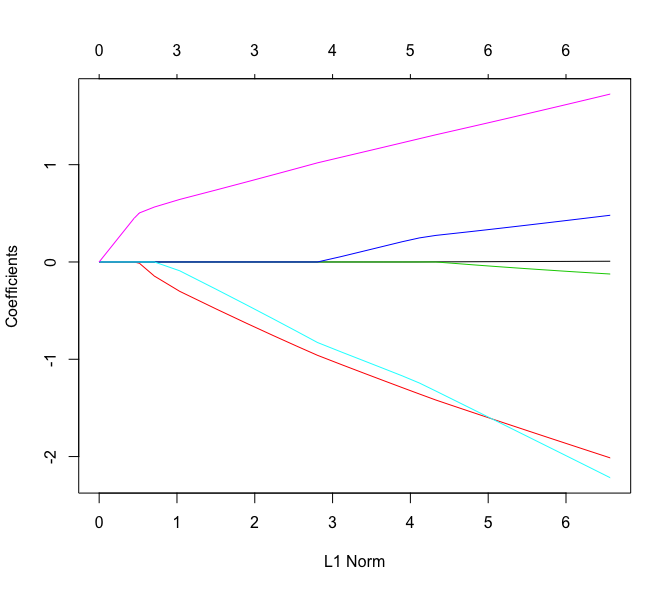
> plot(lasso.model)
lasso.model <- glmnet(x = kidney.dummy, y = kidney.Y, family = "cox", alpha = 1)
plot(lasso.model)
OSCAR Cox 回帰
OSCARは、回帰係数のスパース推定と同時に説明変数のクラスタリングを行うアルゴリズムです。
OSCARの詳しい解説は、川野先生の本をご覧ください。
このCox回帰を行うためにはGithub上の RegCox パッケージがありますが、現在は利用できないようです。
この他にも、Adaptive Lasso Cox や Adaptive Elastic Net Cox, Fused lasso Cox 等が存在するようです。
TD-Cox
Cox比例ハザードモデルは説明変数が時間変化しないという仮定の元に成り立つモデルでした。
しかし、多くの事象の場合で、説明変数は実験開始から変化してしまいます。
そのため、説明変数が時間変化する場合のモデルも考えられています。
説明変数が変化する場合、その時点で打ち切らずに観測が終了するとします。
次のステップで、説明変数が変化し、次の観測が続いていきます。
これを繰り返していくことで、説明変数が時間変化していく観測を表現することができます。
<後で追加>
層別比例ハザードモデル
例えば、性別が男女のように、ある共変量が階層的である場合があります。
その際は、モデルを階層化することで、説明しやすいモデルの形成やより精度の良い当てはまりを生み出すことができます。
Cox比例ハザードモデルも階層化することができ、次のように表します。
h_s(t| x)=h_{s0}(t)exp(\beta x)
ここで、$s=1,2,\dots,S$の層を表します。
共変量の階層ごとにベースハザード関数が存在し、線形予測子部分$exp(\beta x)$部分はどの階層でも共通です。
参考にしたテキストでは、共変量が実験デザインによって固定されている場合は最良であるとしている。
競合リスクモデル
<後で追加>
フレイルティモデル
生存時間分析の多くのモデルでは、各共変量とベースハザードが一意に決まっており、生存に影響する他の要因は存在しないという大きな仮定の元成り立っています。(当たり前のことですが)
しかし、実際には観測されていない共変量や個人差が存在している可能性も大いになり、それらの影響は無視することができない場合もあると考えられます。
この時、このような観測されていない個人間の差異を表現するために、フレイルティ(frailty)という概念が用いたれます。
個々の被験者に対して観測されていないランダムな効果をハザード関数に組み込むものであり、次のように表すことができます。
h_f(t|x)=z_i h_0(t)exp(\beta x)
ここで$z_i$は、個人$i$のフレイルティを表す変量である。
また、$z_i$は確率分布を仮定する場合が多く、ハザードは負になり得ないことから,平均1分散$\theta$のガンマ分布が最も用いられます。
また、上記の式を変換すると階層比例ハザードモデルの式が得られる。
適切なモデル構築を行うことで、両モデルの精度の差はなくなるとされている。
具体的には、階層が5以上の場合の結果は、ほとんど差がないとされているため、グループ数が5未満であると推定されるばいいに有効な手法であるともに呼ばれている。
関数データによるCox回帰
<あとで追加>
機械学習系モデル
機械学習系のモデルを拡張した生存時間モデルが開発されています。
Random Survival Forest
2008年にIshwaranがランダムフォレストベースの生存時間モデルを提案しています。(論文)
ノードの分割は、分割後の2群の差をlog-rank検定における$\chi^2$統計量であると定義し、これを最大にする変数の閾値を探索していきます。
すなわち、2群の期待生存数の差が最大になるように分割していきます。
それぞれの決定木ごとに累積ハザード関数を求めます。
この累積ハザード関数をアンサンブルをして、最終的な累積ハザード関数を求める形になります。
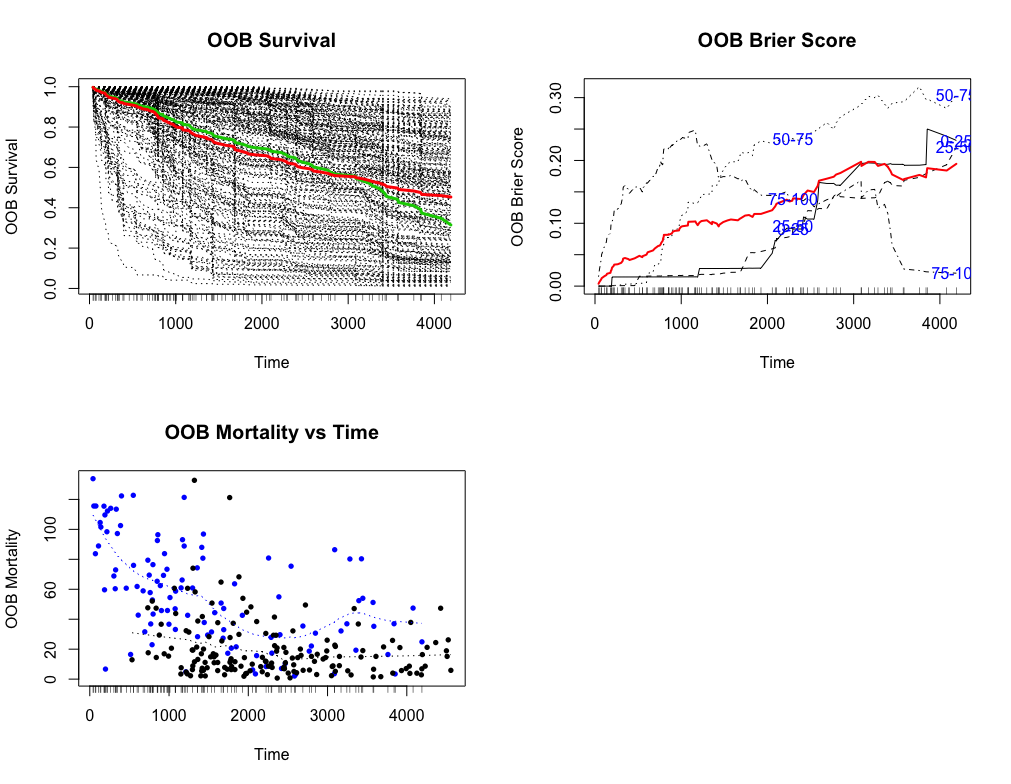
そして、OOBに対してアンサンブル累積ハザード関数を用いて予測誤差を算出してモデルの評価を行います。
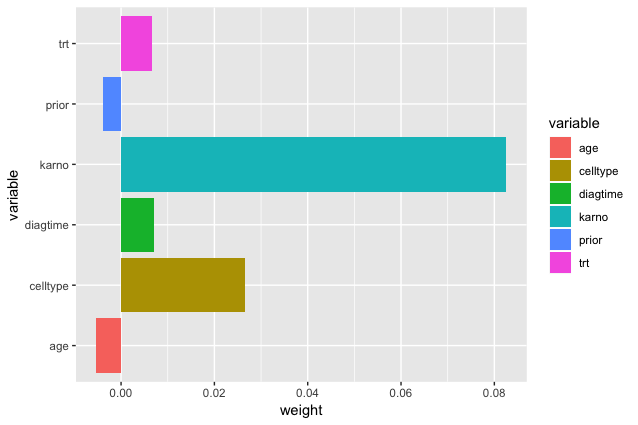
通常のランダムフォレスト同様に変数重要度を算出することができます。
library(randomForestSRC)
library(survival)
data("kidney")
data(veteran, package = "randomForestSRC")
modelRFSRC <- rfsrc(Surv(time, status) ~ ., data = veteran, ntree = 50)
plot(modelRFSRC)
# 変数重要を抽出
RSFimp<-vimp(modelRFSRC)$importance
library(ggplot2)
x <- data.frame(
variable = names(RSFimp),
weight = RSFimp
)
# 変数重要度を可視化
g <- ggplot(x, aes(x = variable, y = weight, fill = variable))
g <- g + geom_bar(stat = "identity")
g <- g + coord_flip()
plot(g)
Support Vector Cox 回帰
2008年にKhanにより提案されたSupport Vector Machineをベースにした生存時間分析モデルです。
時刻tにおいて、次のステップにイベントが発生するかしないかをSVMにより判別します。
そして全ての時刻において、SVMを適用する形です。
この際の損失関数は打ち切りを考慮して正の方向の罰則を少なくするように設計します。
Rではsurvivalsvmパッケージで実行可能です。
ですが、予測に特化しているようで、少し使いにくい印象です。
> require(survivalsvm)
> set.seed(123)
> n <- nrow(veteran)
> train.index <- sample(1:n, 0.7*n, replace = FALSE)
> test.index <- setdiff(1:n, train.index)
> survsvm.reg <- survivalsvm(Surv(veteran$diagtime, veteran$status) ~ .,
+ subset = train.index, data = veteran,
+ type = "regression", gamma.mu = 1,
+ opt.meth = "quadprog", kernel = "add_kernel")
> pred.survsvm.reg <- predict(object = survsvm.reg, newdata = veteran, subset = test.index)
> print(pred.survsvm.reg)
survivalsvm prediction
Type of survivalsvm : regression
Type of kernel : add_kernel
Optimization solver used in model : quadprog
predicted risk ranks : 14.8915.28 11.15 16.11 11.02 ...
survivalsvm version : 0.0.5
predicted risk ranksが生存時間の予測値に対応しているようです。
Generalized Linear Gradient Boosting Model
一般化線形モデル用いた勾配ブースティングモデルです。
mboost パッケージを利用しました。
インストール時に、libmpfr.4.dylibが無いとエラーが出ました。ここ を参照してリンクを作成して解決しました。
library(mboost)
fm <- Surv(time,cens) ~ MGEc.1 + MGEc.2 + MGEc.3 + MGEc.4 + MGEc.5 + MGEc.6 + MGEc.7 + MGEc.8 + MGEc.9 + MGEc.10 + IPI
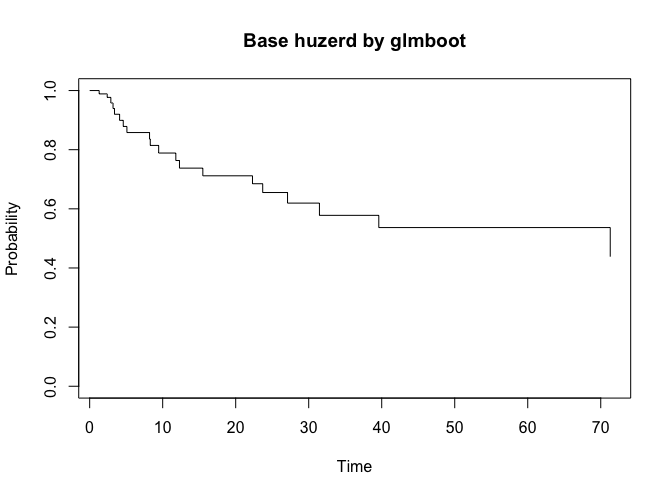
fit <- glmboost(fm, data = DLBCL, family = CoxPH(), control=boost_control(mstop = 500)) %>% step()
summary(fit)
S1 <- survFit(fit)
plot(S1, main="Base huzerd by glmboot")
時間依存の説明変数の場合のGLGBMも存在しているようです。
個人的には、これに注目したいです。
Boosting hazard regression with time-varying covariates
あとで追記(論文)
Boosting Survival Tree
あとで追記
Survival Causal Tree
あとで追記
その他
ここでは紹介にとどめますが、Deep Learingタイプのモデル、Active Learingタイプのモデル、malti Task Learingタイプのモデルもあるようです。
Convolutional survival analysisと呼ばれる、患部の画像からイベントの発生を予測するCNNベースのモデルも提案されているようです。
それぞれ、予測精度が良いようです。
まとめ
生存時間分析はイベントが発生するまで時間や確率に着目した分析です。
今までに統計的モデルから機械学習をベースにしたモデルまで様々提案されています。
統計的にどのパタメータがどのように作用して影響しているのかを知るためには、セミパラメトリックモデルの物が良いと感じます。
機械学習ベースモデルは、予測精度を求める場合には非常に役に立つかもしれません。
月並みな感想でしかありませんが...
log-rank検定の話とかしてないけどいいか〜
参考
・https://www1.doshisha.ac.jp/~mjin/R/Chap_36/36.html
・https://www.slideshare.net/ChandanReddy4/machine-learning-for-survival-analysis
・https://www.slideshare.net/takehikoihayashi/tricky
・http://smrmkt.hatenablog.jp/entry/2013/01/27/142815
・http://www.math.s.chiba-u.ac.jp/~wang/suvival.pdf
・http://terra58.hatenablog.com/entry/2015/03/23/190000
・https://www.elen.ucl.ac.be/Proceedings/esann/esannpdf/es2008-95.pdf