Kaggleコンペ「タイタニック」に挑戦するにあたって
- 筆者はeラーニングでディープラーニング講座修了しE資格取得後に実践的なプログラミングをしたいと思いkaggleに参加。自分の復習用として注釈も入れています。
- 基本的に「タイタニック」講座に即した内容で筆者が講座の内容だけではわからない部分を補強しながら作成。
提出したコードは以下
import pandas as pd
import numpy as np
from sklearn.ensemble import RandomForestClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from sklearn.model_selection import KFold
from sklearn.metrics import accuracy_score
import lightgbm as lgb
import random
import scipy.stats as stats
import os
# 乱数を固定
np.random.seed(0)
random.seed(0)
os.environ["PYTHONHASHSEED"] = "0"
train_df = pd.read_csv('/kaggle/input/titanic/train.csv')
test_df = pd.read_csv('/kaggle/input/titanic/test.csv')
sample_sub = pd.read_csv('/kaggle/input/titanic/gender_submission.csv')
all_df = pd.concat([train_df, test_df], axis=0).reset_index(drop=True)
all_df['Test_Flag'] = 0
all_df.loc[train_df.shape[0]:, 'Test_Flag'] = 1
all_df['Age'] = all_df['Age'].fillna(all_df['Age'].median())
all_df['Fare'] = all_df['Fare'].fillna(all_df['Fare'].median())
all_df['Embarked'] = all_df['Embarked'].fillna('NaN')
all_df['FareBand'] = pd.qcut(all_df['Fare'], 4)
all_df['AgeBand'] = pd.qcut(all_df['Age'], 4)
all_df['FamilySize'] = all_df['SibSp'] + all_df['Parch'] + 1
all_df['MedF'] = all_df['FamilySize'].map(lambda s: 1 if 2 <= s <= 4 else 0)
all_df['LargeF'] = all_df['FamilySize'].map(lambda s: 1 if s >= 5 else 0)
all_df['Alone'] = all_df['FamilySize'].map(lambda s: 1 if s == 1 else 0)
all_df['Title'] = all_df.Name.str.extract(' ([A-Za-z]+)\.', expand=False)
def prepro_name_title(Title):
if Title == 'Master':
return 0
elif Title == 'Miss':
return 1
elif Title == 'Mr':
return 2
elif Title == 'Mrs':
return 3
else:
return 4
all_df['Title_Encode'] = all_df['Title'].map(prepro_name_title)
all_df = pd.get_dummies(all_df, columns=["Sex", "Pclass"])
all_df = pd.get_dummies(all_df, columns=['AgeBand', 'FareBand', 'Embarked'])
train = all_df[all_df['Test_Flag'] == 0]
test = all_df[all_df['Test_Flag'] == 1].reset_index(drop=True)
target = train['Survived']
drop_col = [
'PassengerId', 'Age', 'Ticket', 'Title', 'Fare', 'Cabin', 'Test_Flag', 'Name', 'Survived'
]
train = train.drop(drop_col, axis=1)
test = test.drop(drop_col, axis=1)
cv = KFold(n_splits=3, random_state=0, shuffle=True)
train_acc_list = []
val_acc_list = []
lgb_params = {
"objective": "binary",
"metric": "binary_error",
"force_row_wise": True,
"seed": 0,
'learning_rate': 0.17722952605537962,
'num_leaves' : 35,
'min_data_in_leaf': 5,
'max_depth': 13
}
rename_dict = {
'AgeBand_(0.169, 22.0]': 'AgeBand_1',
'AgeBand_(22.0, 28.0]': 'AgeBand_2',
'AgeBand_(28.0, 35.0]': 'AgeBand_3',
'AgeBand_(35.0, 80.0]': 'AgeBand_4',
'FareBand_(-0.001, 7.896]': 'FareBand_1',
'FareBand_(7.896, 14.454]': 'FareBand_2',
'FareBand_(14.454, 31.275]': 'FareBand_3',
'FareBand_(31.275, 512.329]': 'FareBand_4'
}
for i, (trn_index, val_index) in enumerate(cv.split(train, target)):
print(f'Fold : {i}')
X_train, X_val = train.loc[trn_index].rename(columns=rename_dict), train.loc[val_index].rename(columns=rename_dict)
y_train, y_val = target.iloc[trn_index], target.iloc[val_index]
lgb_train = lgb.Dataset(X_train, y_train)
lgb_valid = lgb.Dataset(X_val, y_val)
model_lgb = lgb.train(
params=lgb_params,
train_set=lgb_train,
valid_sets=[lgb_train, lgb_valid],
verbose_eval=0,
early_stopping_rounds=10
)
print('-' * 10 + ' Start_rf ' + '-' * 10)
model_rf = RandomForestClassifier(
random_state=0, max_depth=15,
min_samples_leaf=5, min_samples_split=5
)
model_rf.fit(X_train, y_train)
print('-' * 10 + ' Start_rogi ' + '-' * 10)
model_rogi = LogisticRegression()
model_rogi.fit(X_train, y_train)
print('-' * 10 + ' Start_SVM ' + '-' * 10)
model_svm = SVC(random_state=0)
model_svm.fit(X_train, y_train)
train_pred = np.zeros((len(y_train), 4))
train_pred[:, 0] = np.where(model_lgb.predict(X_train) >= 0.5, 1, 0)
train_pred[:, 1] = model_rf.predict(X_train)
train_pred[:, 2] = model_rogi.predict(X_train)
train_pred[:, 3] = model_svm.predict(X_train)
train_acc = accuracy_score(
y_train, stats.mode(train_pred, axis=1)[0]
)
train_acc_list.append(train_acc)
val_pred = np.zeros((len(y_val), 4))
val_pred[:, 0] = np.where(model_lgb.predict(X_val) >= 0.5, 1, 0)
val_pred[:, 1] = model_rf.predict(X_val)
val_pred[:, 2] = model_rogi.predict(X_val)
val_pred[:, 3] = model_svm.predict(X_val)
val_acc = accuracy_score(
y_val, stats.mode(val_pred, axis=1)[0]
)
val_acc_list.append(val_acc)
print('-' * 10 + 'Result' + '-' * 10)
print(f'Train_acc : {train_acc_list}, Ave: {np.mean(train_acc_list)}')
print(f'Valid_acc : {val_acc_list}, Ave: {np.mean(val_acc_list)}')
# 提出ファイルを出力 (1.3.6)
sample_sub = pd.read_csv('/kaggle/input/titanic/gender_submission.csv')
test_pred = np.array([1, 0])
mode_pred = np.repeat(test_pred[np.argmax(np.bincount(test_pred))], len(sample_sub))
sample_sub["Survived"] = mode_pred
sample_sub["Survived"] = sample_sub["Survived"].astype('int8')
sample_sub.to_csv("submission.csv", index=False)
1 データの確認
1-1 データの読み込み
まずは用意されているデータセットのtrain.csv test.csv gender_submission.csvを読み込みます
train_df = pd.read_csv('/kaggle/input/titanic/train.csv') #訓練用データ
test_df = pd.read_csv('/kaggle/input/titanic/test.csv') # 評価用データ
sample_sub = pd.read_csv('/kaggle/input/titanic/gender_submission.csv') #提出用データ
1-2 データの型と中身を確認
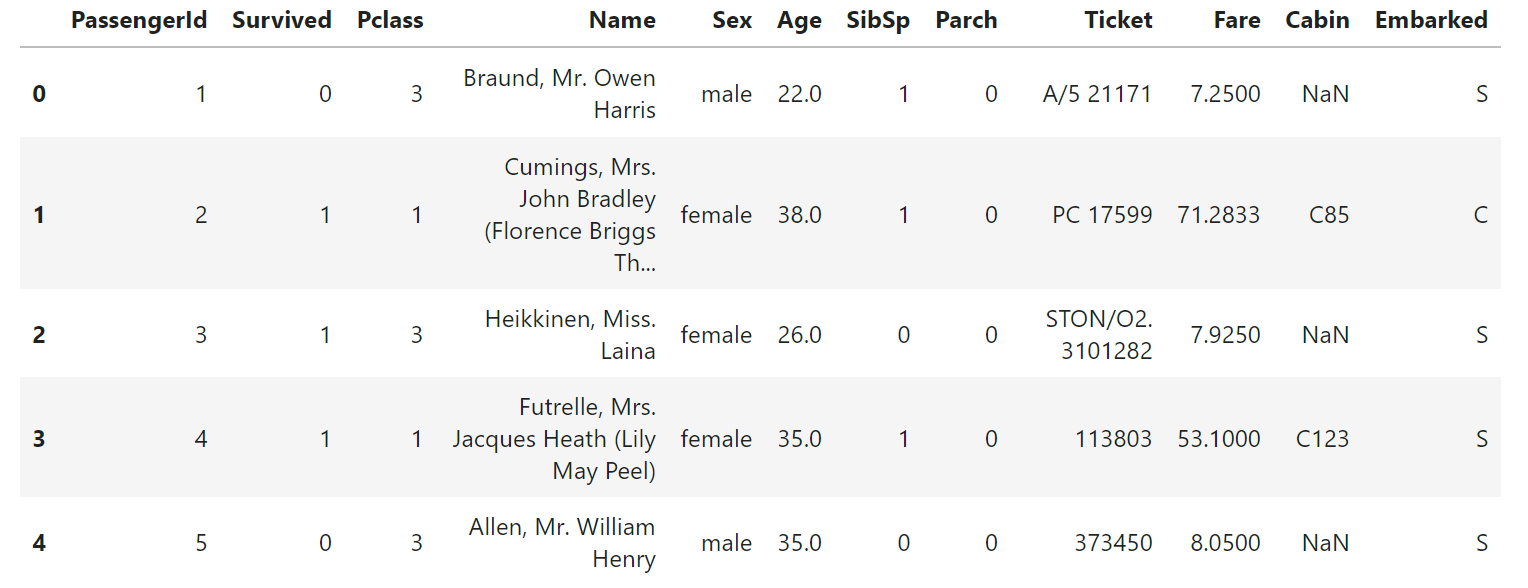
print(f'Train_df_shape : {train_df.shape}\n')
#Train_df_shape : (891, 12)
print(f'{train_df.dtypes}\n')
#PassengerId int64
Survived int64
Pclass int64
Name object
Sex object
Age float64
SibSp int64
Parch int64
Ticket object
Fare float64
Cabin object
Embarked object
dtype: object
shapeより891人の乗客データと12列のデータを確認。
dtypesによりint,object,float型を確認。
display(train_df.head())
1-3 統計量の確認
まずカテゴリカル変数を数値から文字列へ変更し数値データの統計量を表示します。
- カテゴリカルデータ:量的ではないデータでタイタニックのデータではName,Sex,Ticket等。カテゴリカル変数は、数値的な大小関係や算術演算が意味を持たないデータを表現するために使用される。例として今回なら客室クラス(Pclass)やPassengerId。これらには順序や大小関係は無いです。
train_df = train_df.astype({
'PassengerId' : str,
'Pclass' : str
})
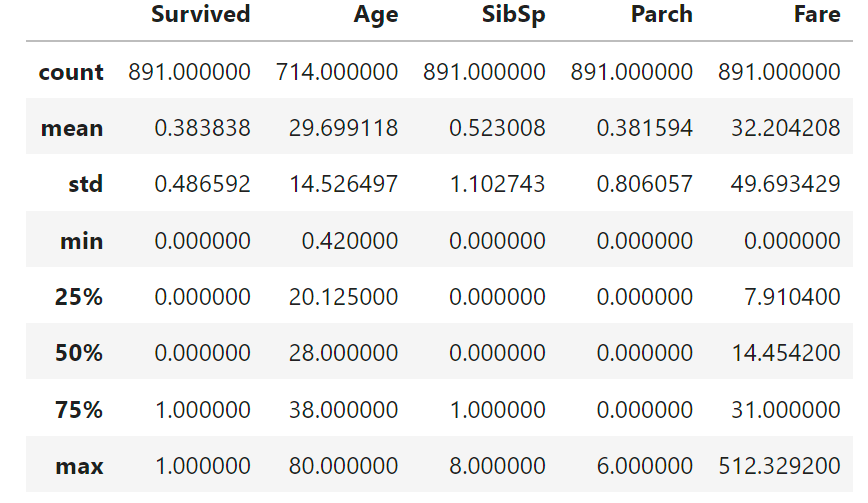
display(train_df.describe())
- describe()メソッド:データフレームの統計情報(平均や最大等)を抽出する。
- ()からカッコだと数値データのみ
- describe(exclude='number') 数値データ以外
- describe((include=’all’) 全てのデータ型
- describe(include=['O']) 大文字の’O’でオブジェクトデータ(str型等)に関する情報を表示
# カテゴリカルデータの統計量を表示
display(train_df.describe(exclude='number'))
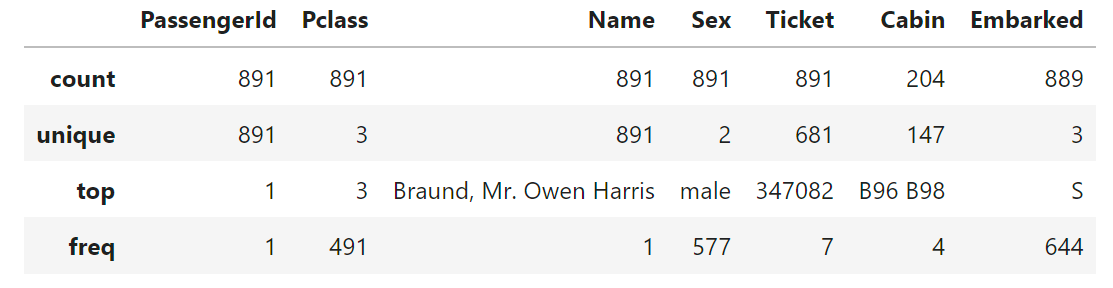
以上によりCabinやEmbarkedのcoountが891になっていないことで欠損値を、カテゴリカル変数のuniqueからPassengerID,Nameから重複のない変数であることを確認。
仮説として
- Survivedの平均が0.5より小さいので目的変数には偏りがあるのではないか。
- Age,Fareの最大値が平均値や中央値から大きくひらいており外れ値があるのではないか。
- SidSp,Parchの半分以上は0でこのため同乗していた家族の人数には偏りがありそう。
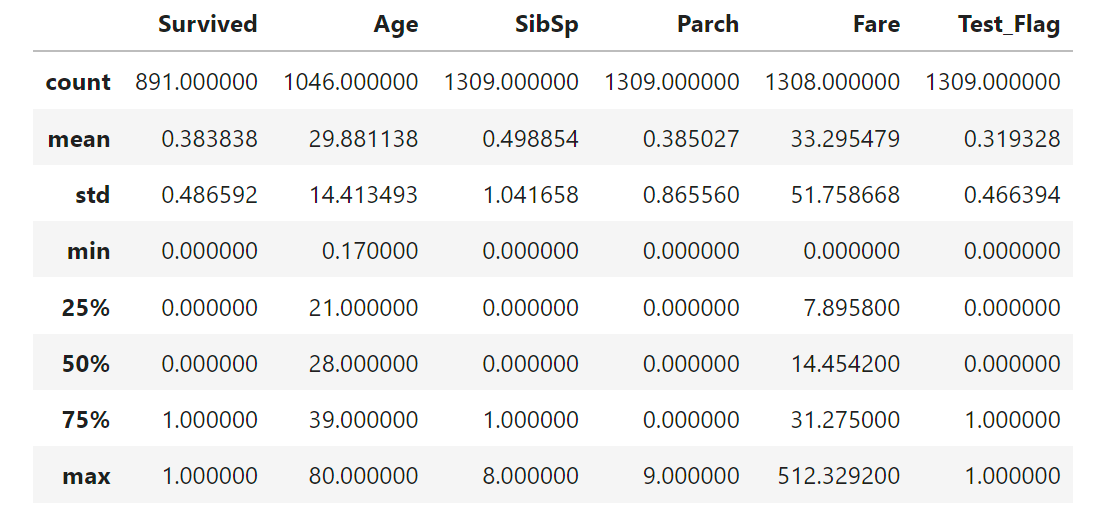
1-4 訓練データとテストデータを結合しall_dfを作成
all_df = pd.concat([train_df,test_df],axis=0).reset_index(drop=True)
# 訓練データとテストデータを判定するためのカラムを作成
all_df['Test_Flag'] = 0
all_df.loc[train_df.shape[0]:,'Test_Flag'] = 1
display(all_df.describe())
2 データの可視化
2-1 重複の確認
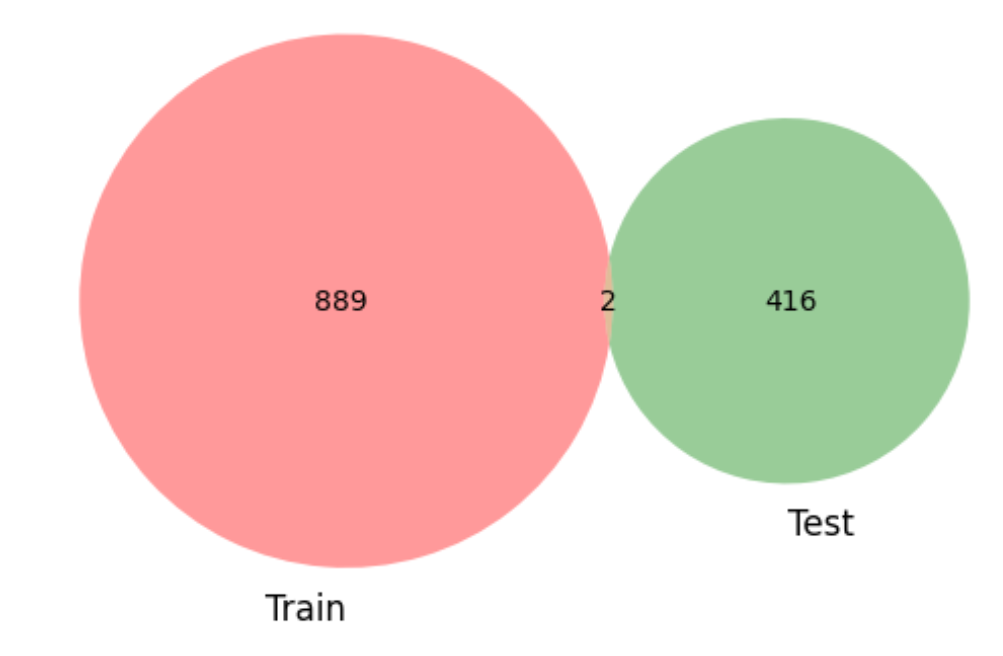
訓練データとテストデータの重複を確認します。
import matplotlib.pyplot as plt
from matplotlib_venn import venn2
fig ,axes = plt.subplots(figsize=(8,8),ncols=3,nrows=1)
for col_name,ax in zip(
['Name','Pclass','Cabin']
,axes.ravel()
):
venn2(
subsets=(set(train_df[col_name].unique()),
set(test_df[col_name].unique())),
set_labels=('Train','Test'),
ax=ax)
注)コード実行すると
/opt/conda/lib/python3.10/site-packages/scipy/init.py:146: UserWarning: A NumPy version >=1.16.5 and <1.23.0 is required for this version of SciPy (detected version 1.23.5
warnings.warn(f"A NumPy version >={np_minversion} and <{np_maxversion}"
がでますがエラーではなく、scipyのバージョンによる警告なので無視できます。
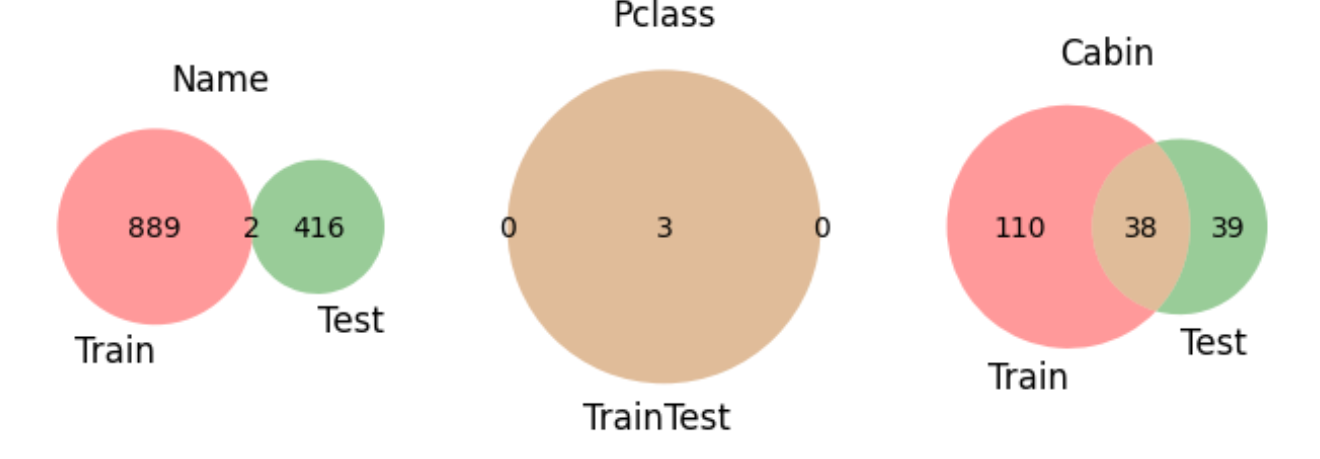
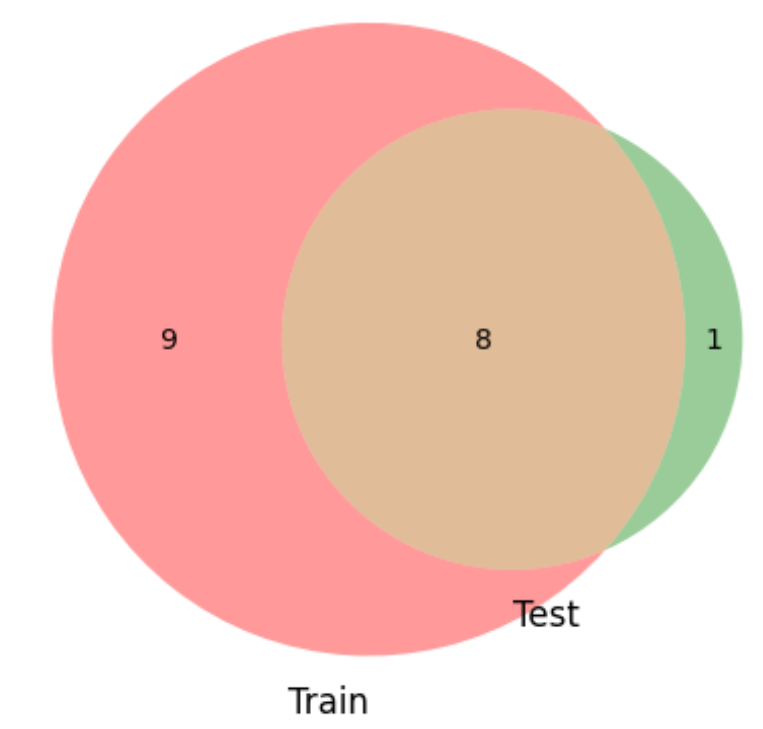
Nameから同じ名前は二人、Cabinには重なりがあるので家族が考えられる。
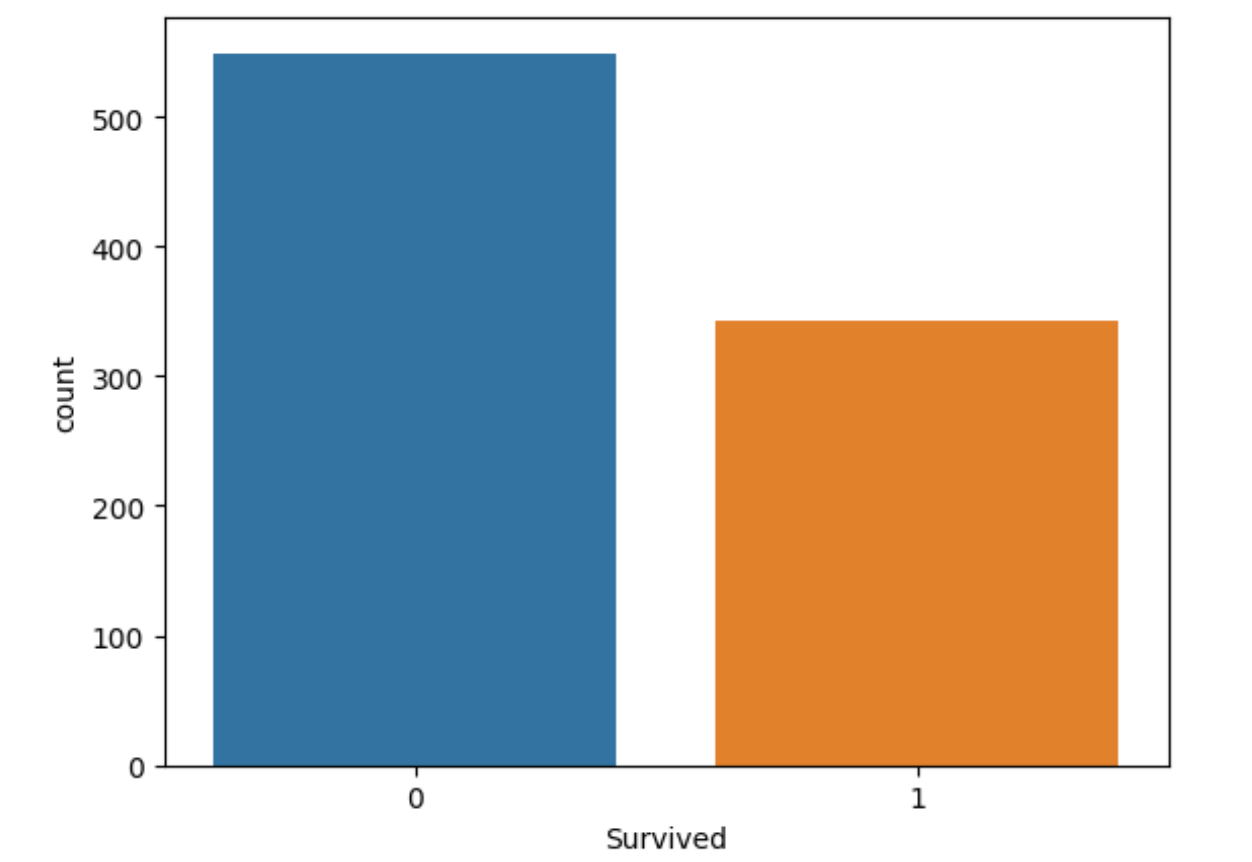
2-2 各カテゴリの分布の確認
Survived,Sex,Age,Fare,SidSp,Parchの分布の確認
import seaborn as sns
sns.countplot(x='Survived',data=train_df)
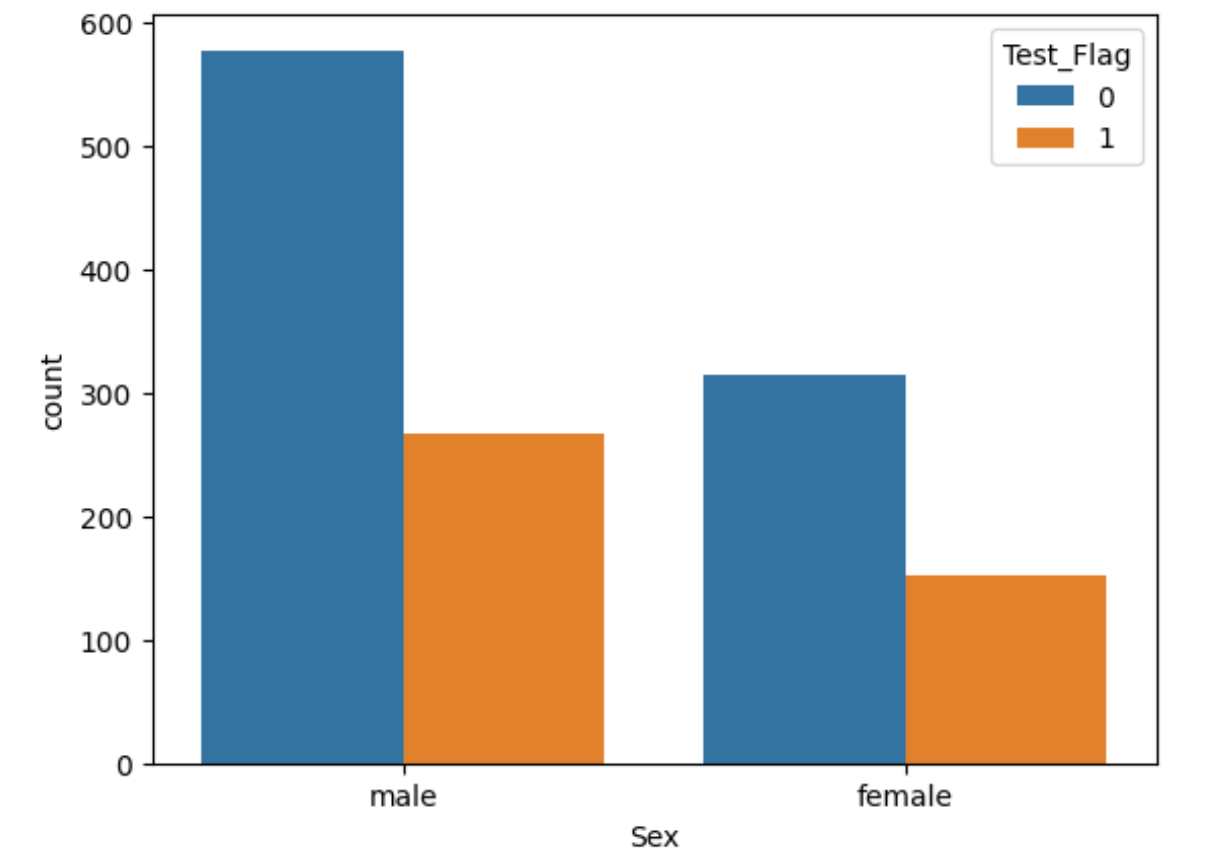
sns.countplot(x='Sex',hue='Test_Flag',data=all_df)
fig = sns.FacetGrid(all_df,col='Test_Flag',hue='Test_Flag',height=4)
fig.map(sns.histplot,'Age',bins=30,kde=False)
注)こちらも実行すると
/opt/conda/lib/python3.10/site-packages/seaborn/axisgrid.py:118: UserWarning: The figure layout has changed to tight
self._figure.tight_layout(*args, **kwargs)
と警告が出ますがこれもバージョンによるものでグラフが表示できれば特に問題はありません。
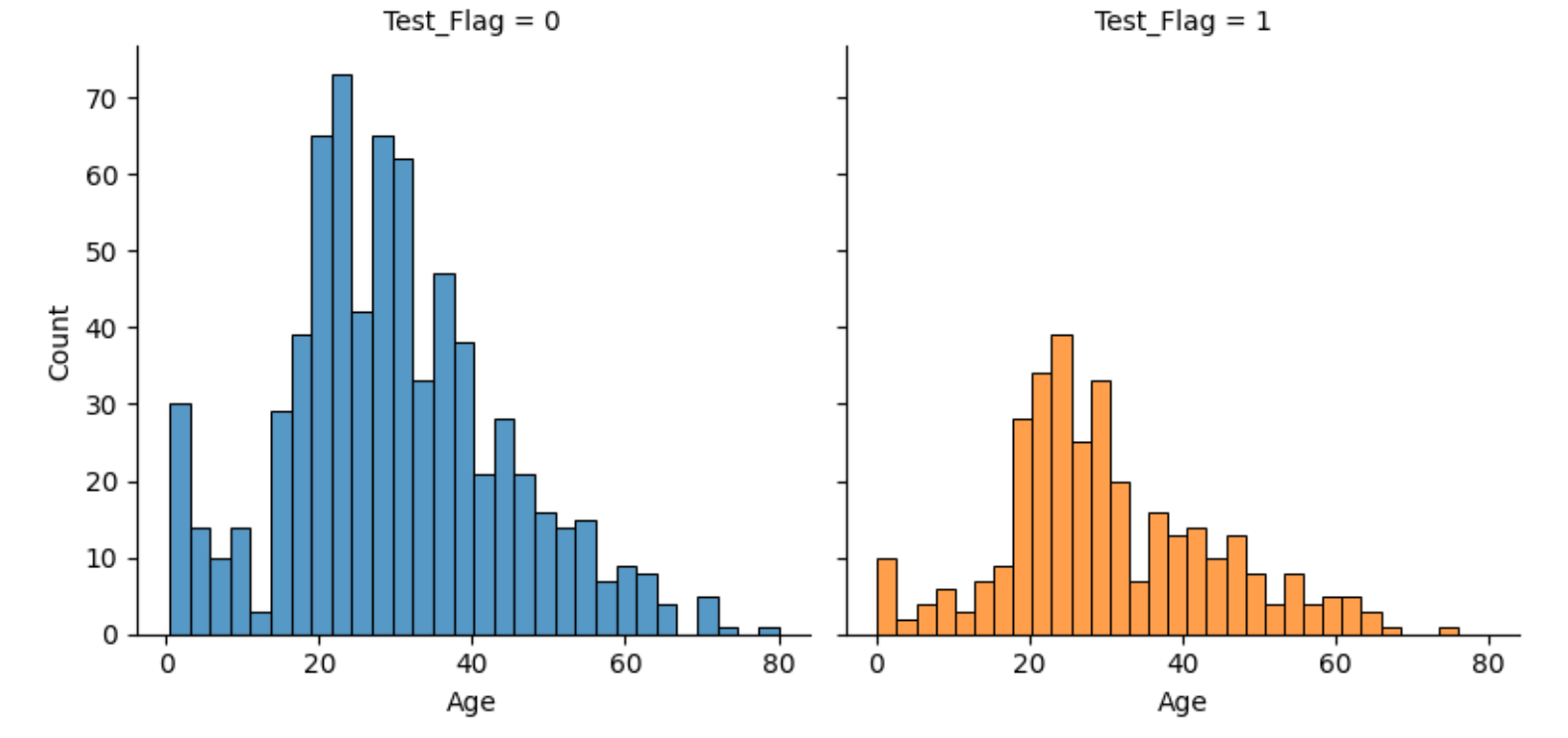
fig = sns.FacetGrid(all_df,col='Test_Flag',hue='Test_Flag',height=4)
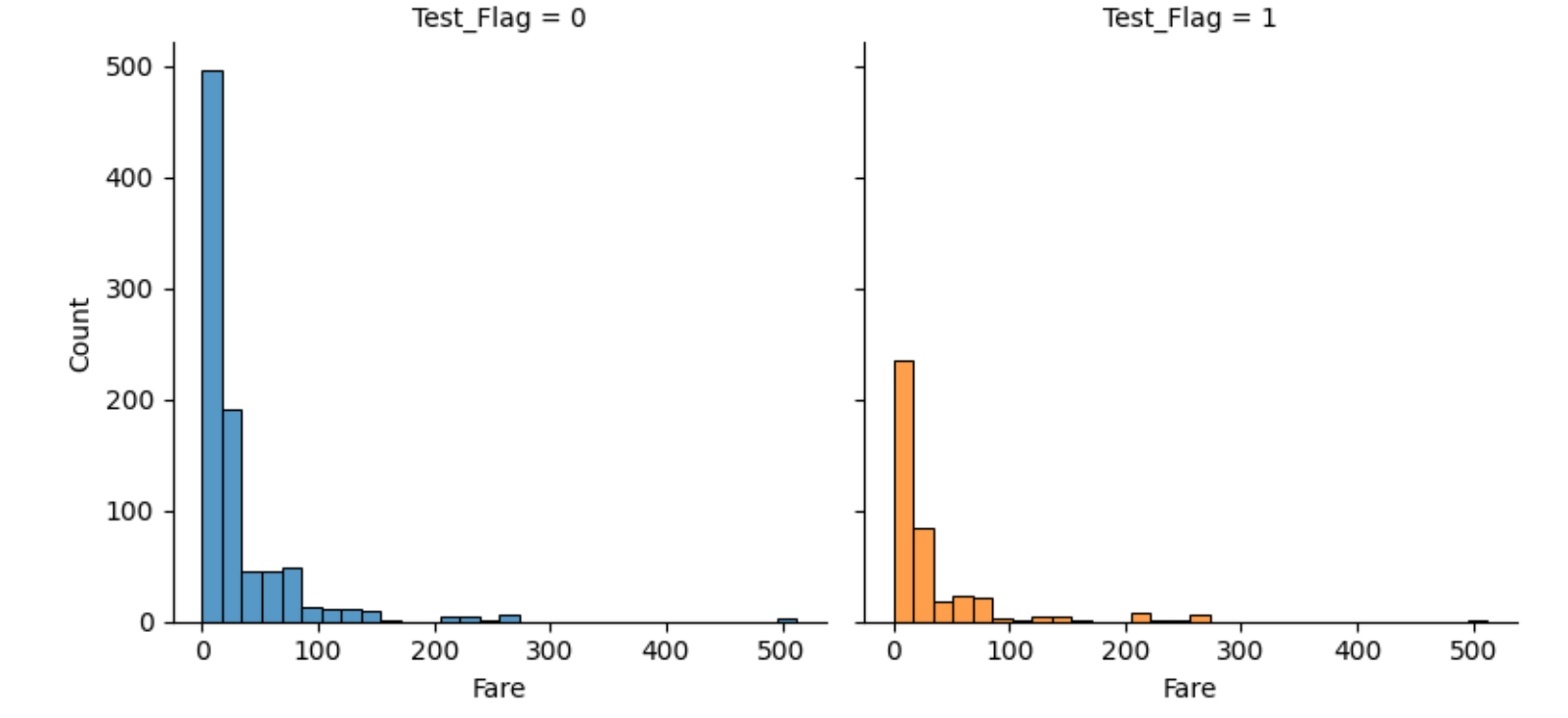
fig.map(sns.histplot,'Fare',bins=30,kde=False)
sns.countplot(x='SibSp',hue='Test_Flag',data=all_df)
plt.show()
sns.countplot(x='Parch',hue='Test_Flag',data=all_df)
plt.legend(title='Test_Flag',loc='upper right')
plt.show()
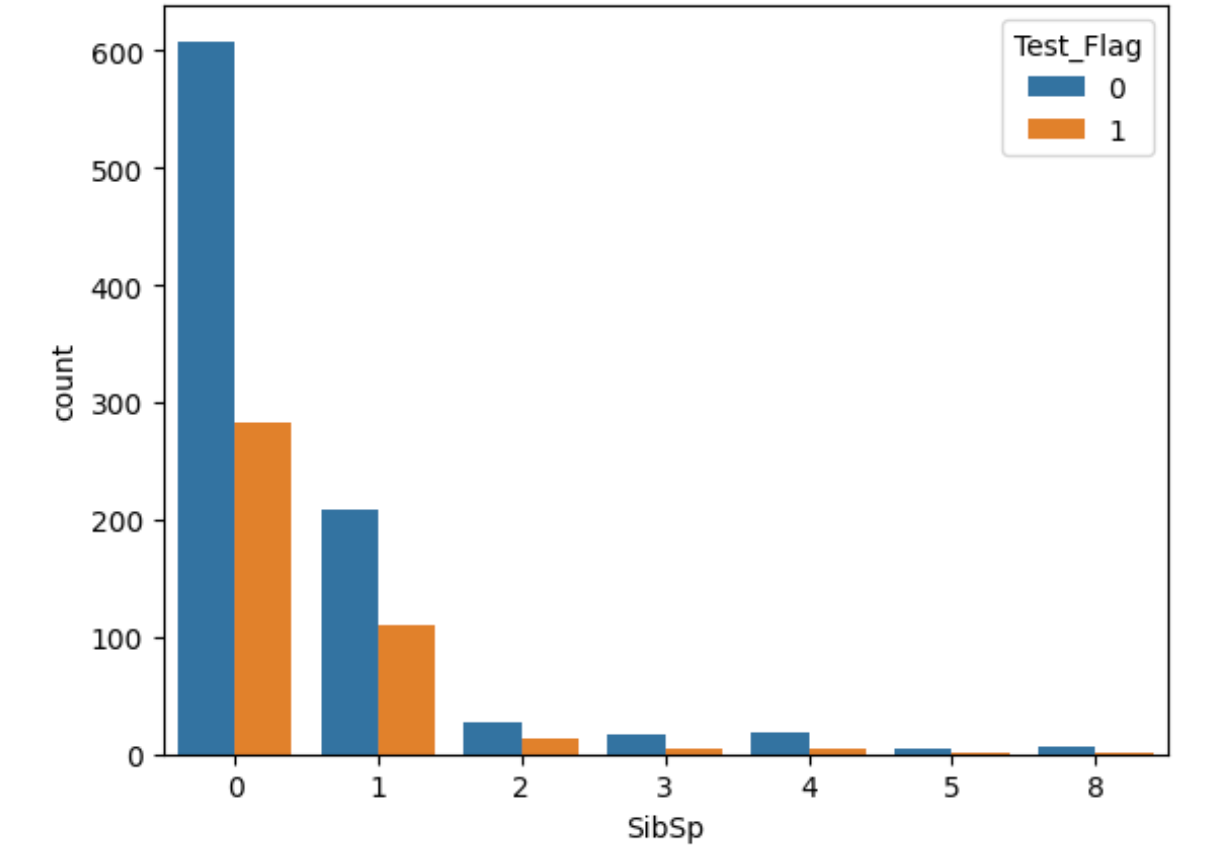
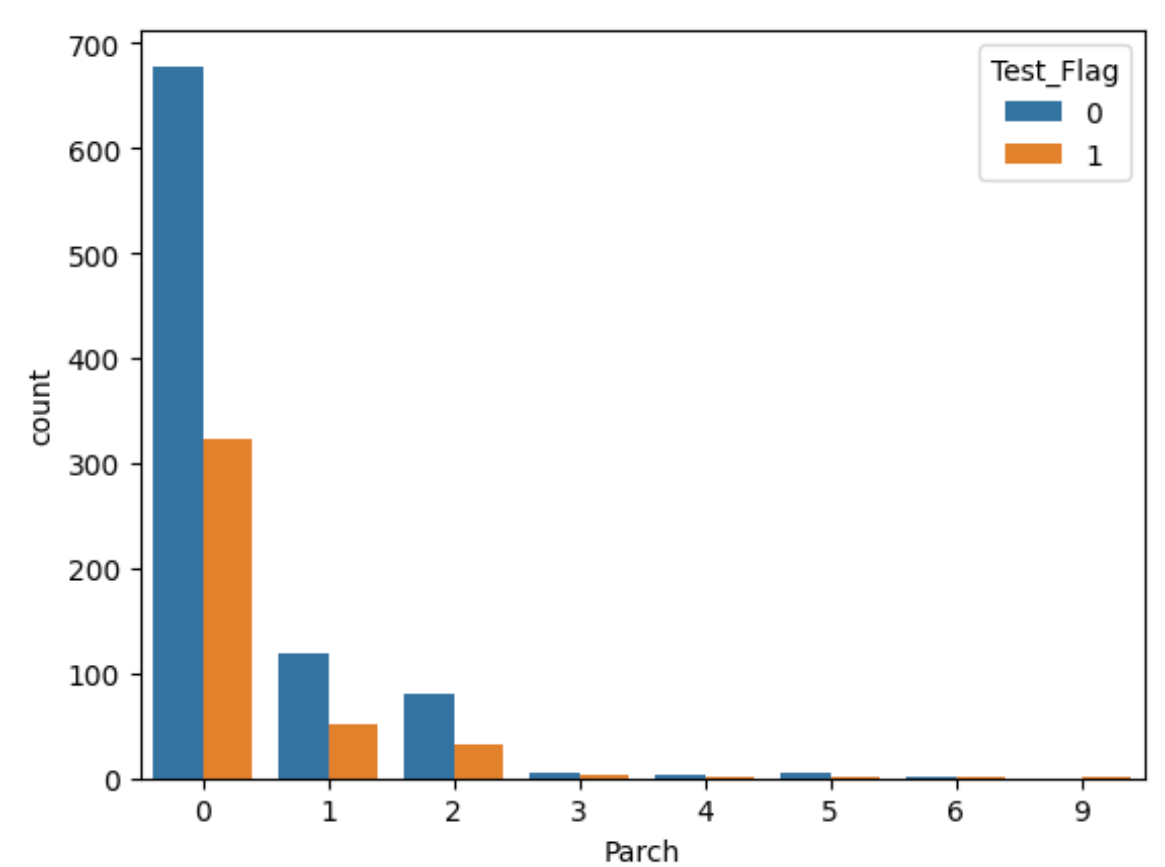
以上からSexについては男性が多く、Fareには外れ値があること、SibSpとParchから0人が多いことを確認。
2-3 データの相関性を可視化
- seaborn.heatmap()とは色の濃淡や色相でデータの密度や値の分布を可視化します。
sns.heatmap(train_df[['Survived','Age','SibSp','Parch','Fare']].corr(),
vmax=1,vmin=-1,annot=True)
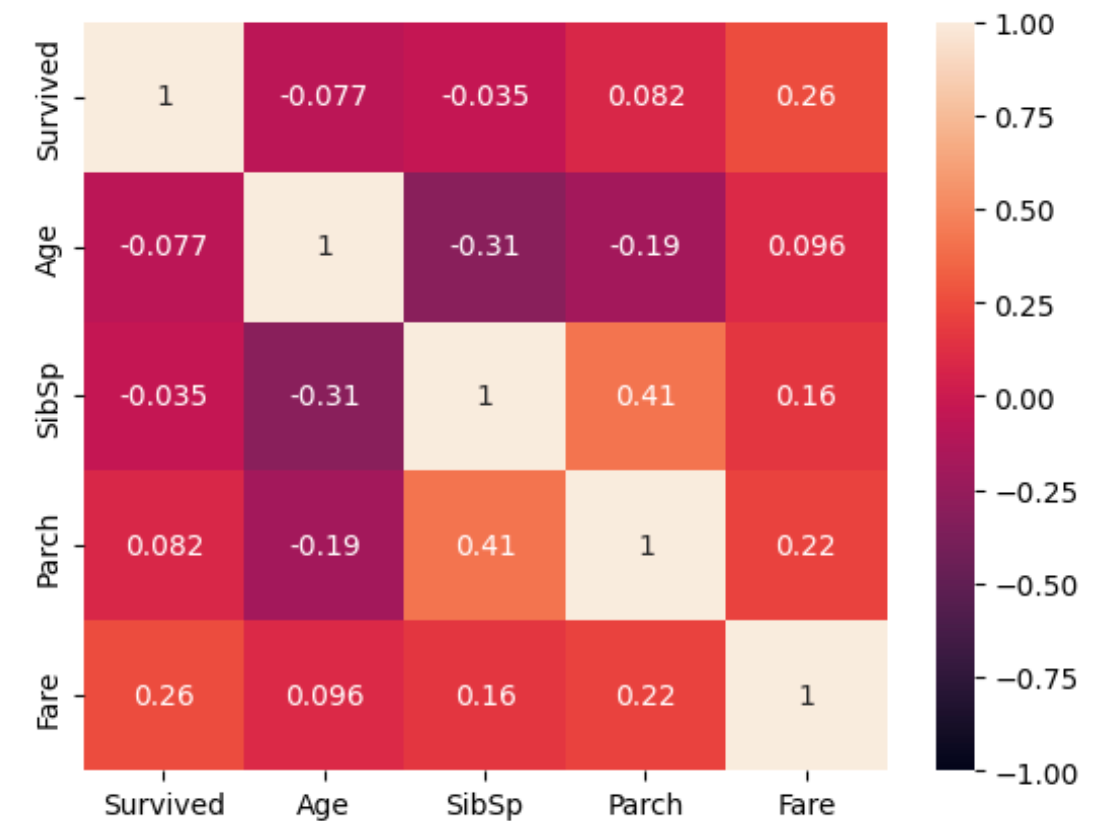
ヒートマップによりSidSpとParchにおいて正の弱い相関、SidSpとAgeにおいて負の弱い相関があることを確認。
2-4 各カラム(Sex,Pclass,Emberked,Age,Fare)と目的変数(Survived)の関係を可視化。
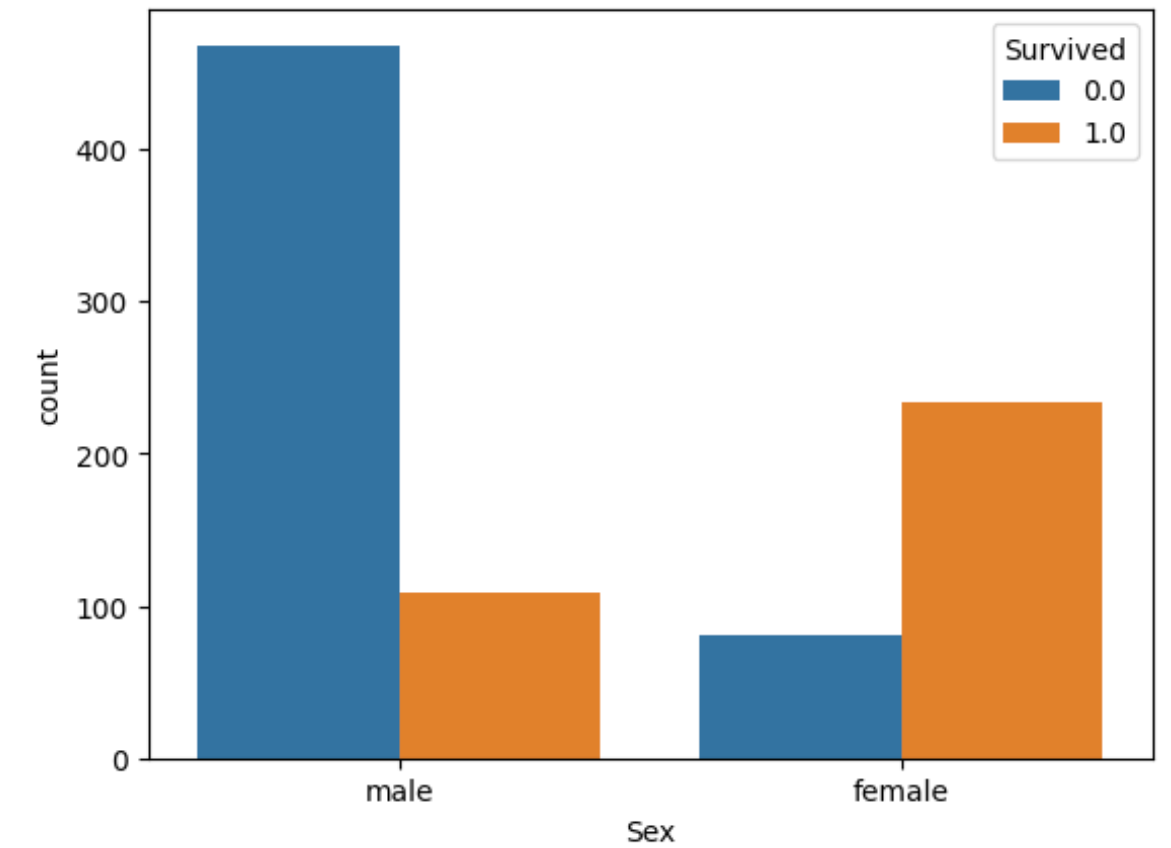
sns.countplot(x='Sex',hue='Survived',data=all_df)
plt.show()
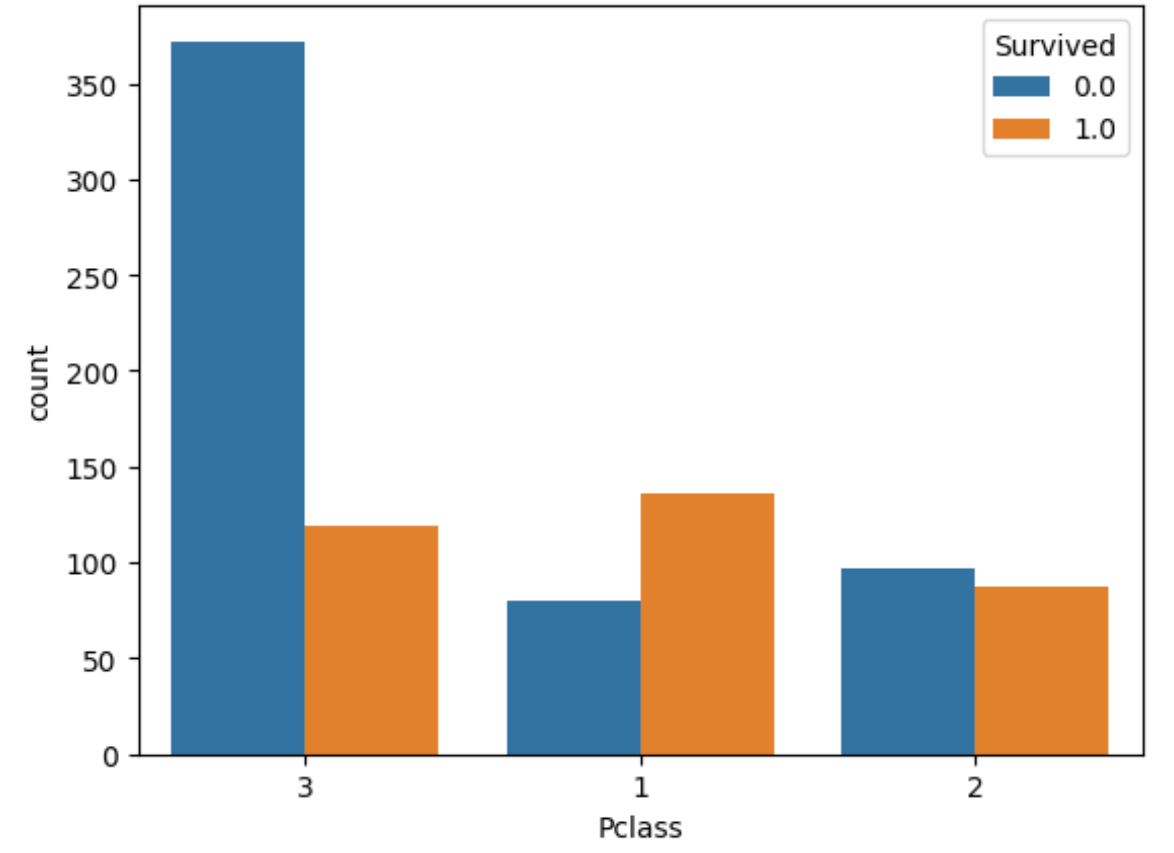
sns.countplot(x='Pclass',hue='Survived',data=all_df)
plt.show()
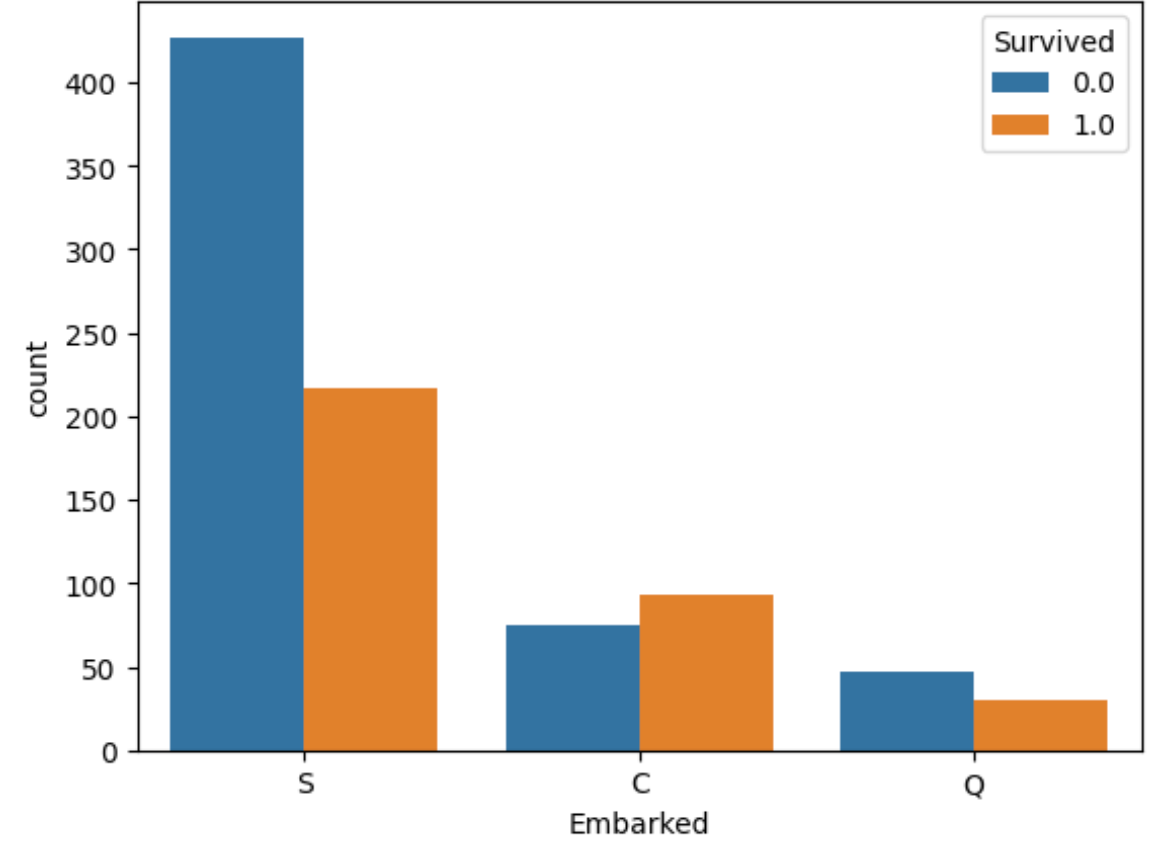
sns.countplot(x='Embarked',hue='Survived',data=all_df)
plt.show()
fig = sns.FacetGrid(train_df,col='Survived',hue='Survived',height=4)
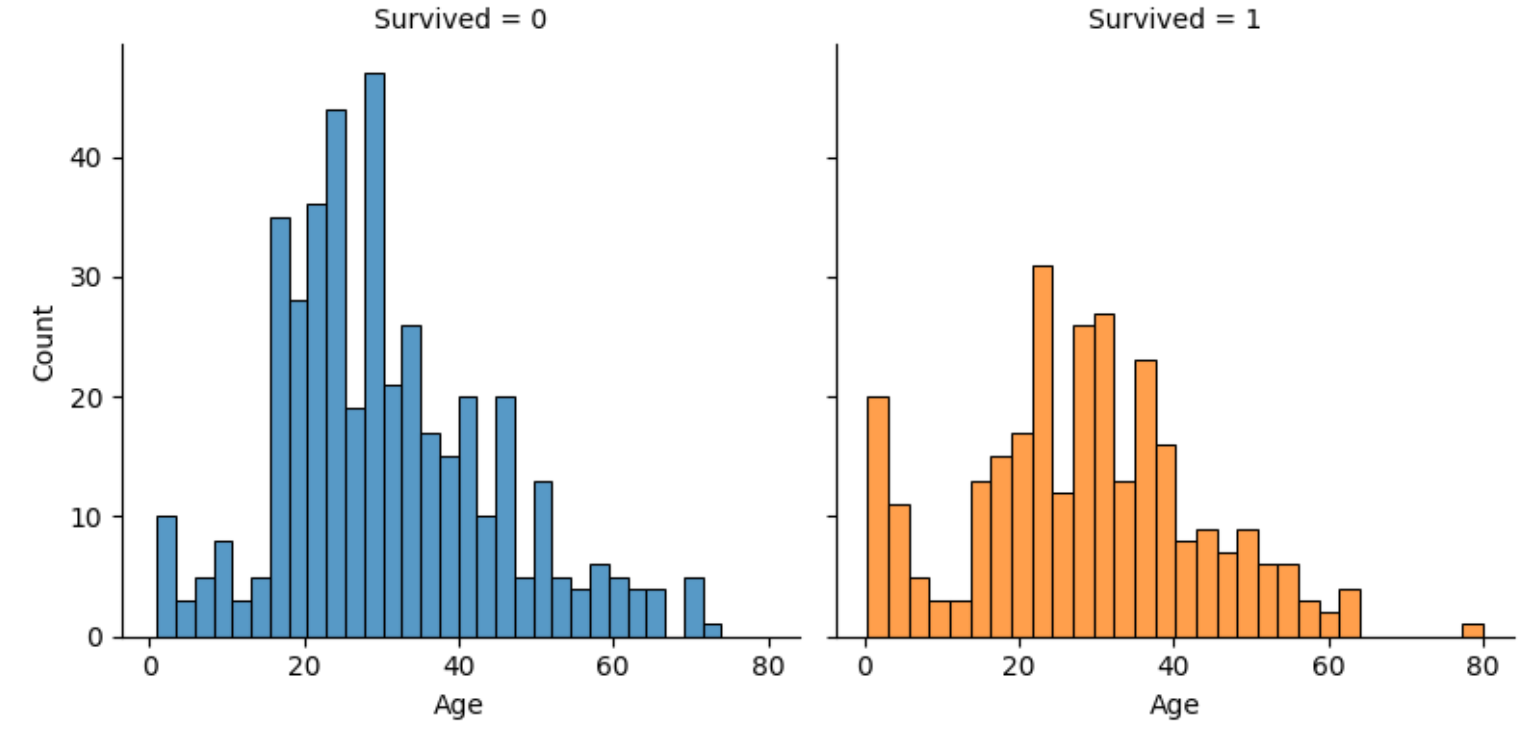
fig.map(sns.histplot,'Age',bins=30,kde=False)
fig = sns.FacetGrid(train_df,col='Survived',hue='Survived',height=4)
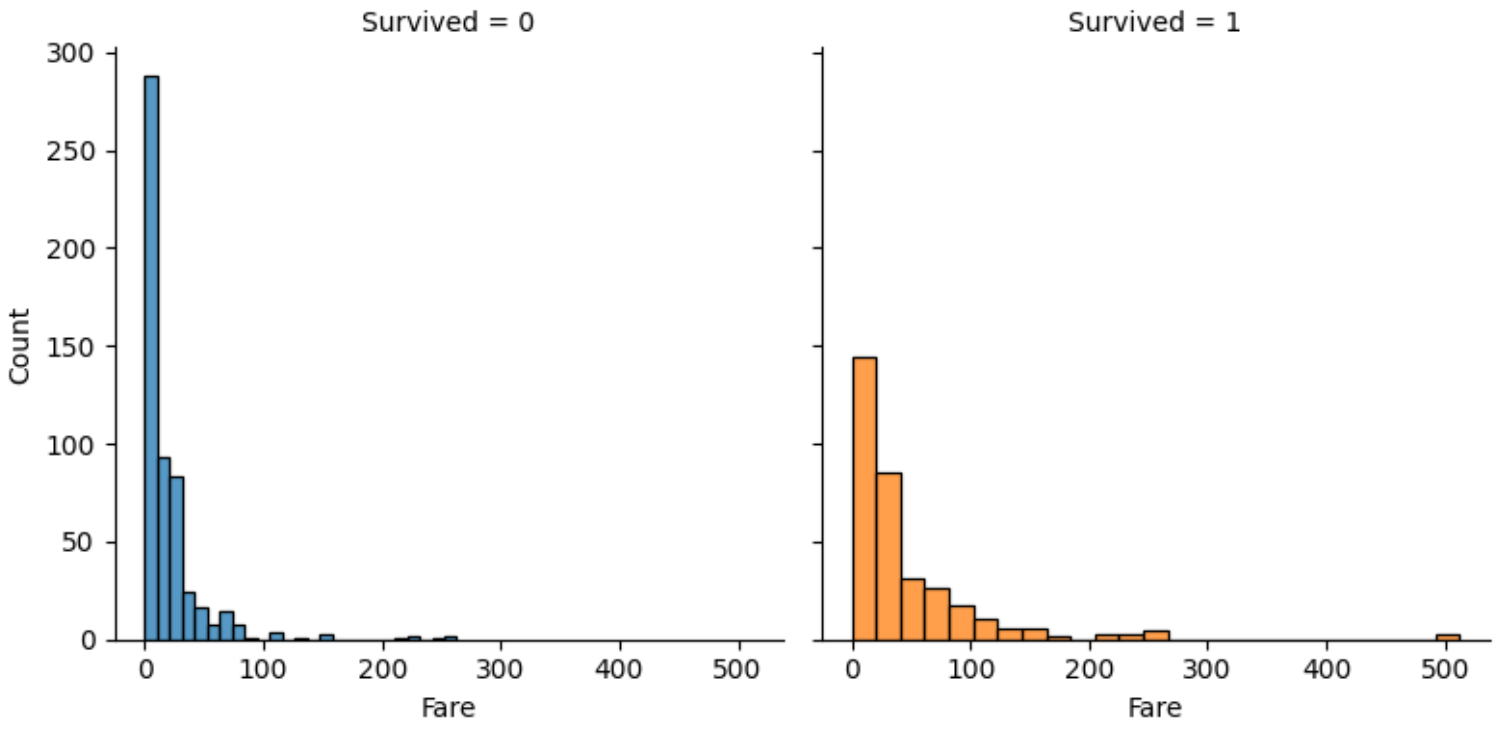
fig.map(sns.histplot,'Fare',bins=25,kde=False)
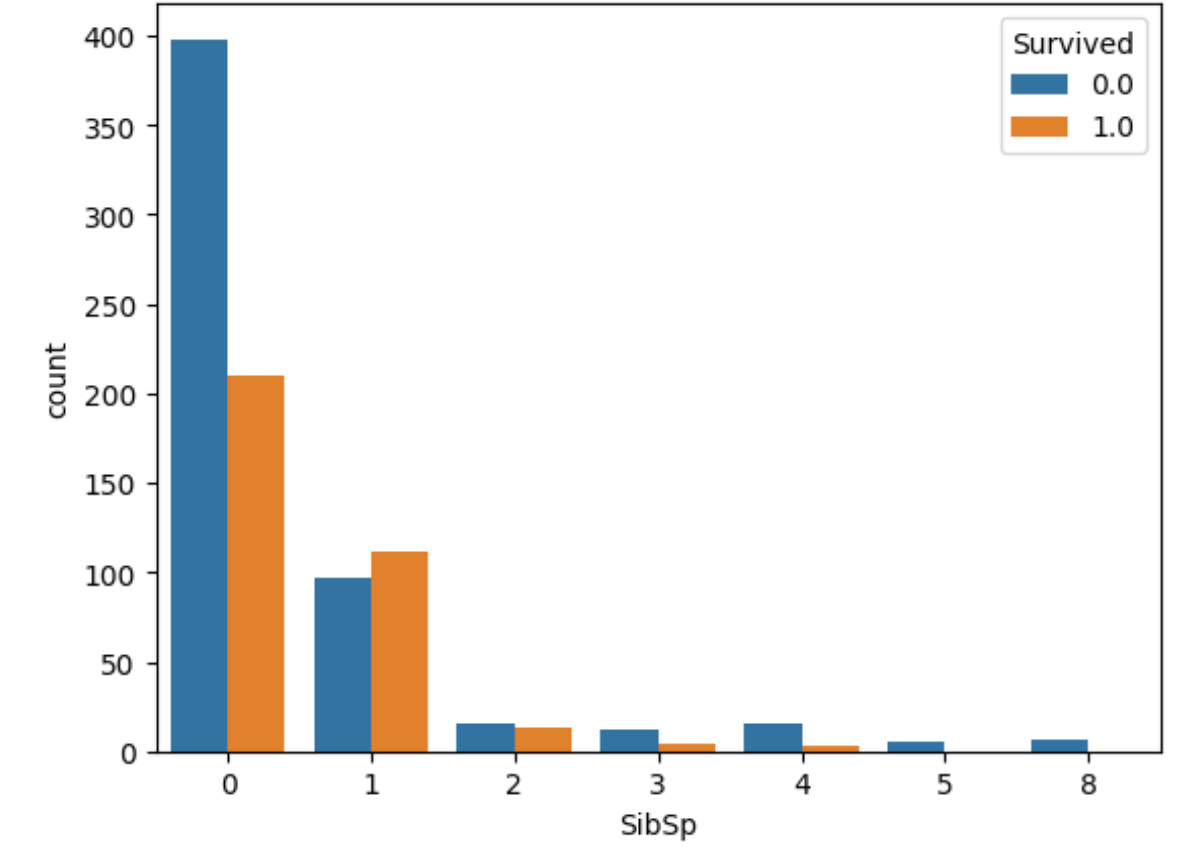
sns.countplot(x='SibSp',hue='Survived',data=all_df)
plt.legend(title='Survived',loc='upper right')
plt.show()
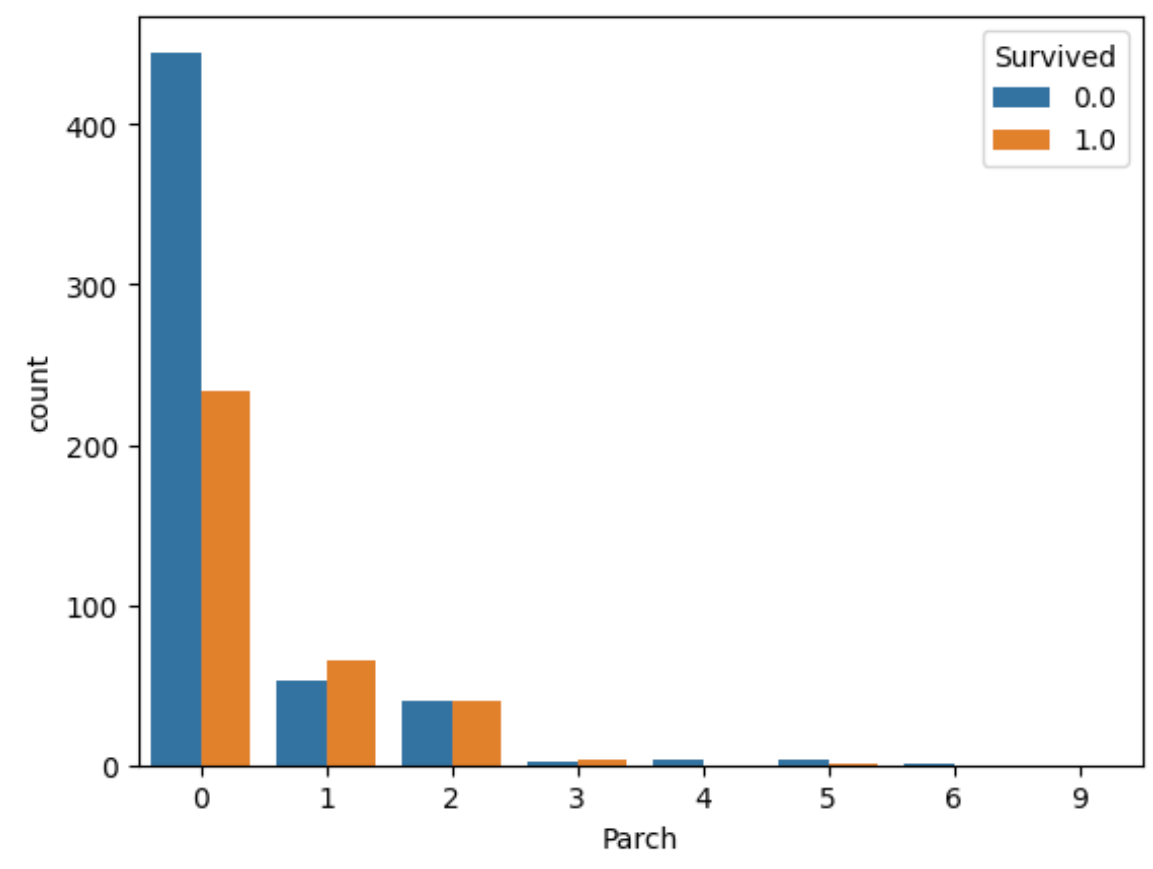
sns.countplot(x='Parch',hue='Survived',data=all_df)
plt.legend(title='Survived',loc='upper right')
plt.show()
- 性別で生存率に差がある。
- Pclass(チケットのクラス)は数字が大きいほど生存率は下がる。
- Embarked(乗船した港)によっても生存率に差がある。
- Ageについては0~10歳程度が生存率が高く20~30代は生存率が低い。
- Fareから運賃が低いほど生存率も低い。
- SisSpと相関で1~2人が生存率が高い。
- Parchから同乗した親子供1~3人の生存率が高い。
3 特徴量作成
3-1 特徴量作成の方針
- SidSip,Parchは前処理なし。
- Age,Emberked,Fareの欠損値を補完。Cabinは欠損が多いので採用しない。
- Fareの外れ値の対策として4つの区間に分類し数値データをカテゴリカル変数に変換する。
- Ageも年代別で生存率が異なるので同様に4つの区分に分類し数値データをカテゴリカル変数に変換する。
- カテゴリカル変数はSex,Emberked,Pclass,Age,Fareを採用しそれぞれにOne-Hot Encodingを行う。
all_df['Embarked'] = all_df['Embarked'].fillna('NaN') #Nanで補完
all_df['Age'] = all_df['Age'].fillna(all_df['Age'].median()) #中央値で補完
all_df['Fare'] = all_df['Fare'].fillna(all_df['Fare'].median()) # 中央値で補完
# Fareを4つの区間に分類し、カテゴリカル変数に変換
all_df['FareBand'] = pd.qcut(all_df['Fare'],4)
# Ageを4つの区間に分類し、カテゴリカル変数に変換
all_df['AgeBand'] = pd.qcut(all_df['Age'],4)
# Sex、PclassをOne-Hot_Encodingで変換
all_df = pd.get_dummies(all_df,columns=['Sex','Pclass'])
all_df = pd.get_dummies(all_df,columns=['Embarked','AgeBand','FareBand'])
注)pd.qcut() 関数はデータを指定された分位数(quantiles)に基づいてバンドに分割するためのPandasの関数でこの場合Fare 列の値を4つの等間隔のバンドに分割。
注)pd.get_dummies() カテゴリカルな特徴量をダミー変数(one-hotエンコーディング)に変換し、内容はall_df データフレーム内の Sex 列と Pclass 列をダミー変数に変換。
3-2 検証データの作成と分析
- 今回はホールドアウト法を使用。
- Survivedの0か1の二値分類なので一般的なロジスティック回帰を使用。
from sklearn.model_selection import train_test_split
# 前処理を施したall_dfを訓練データとテストデータに分割
train = all_df[all_df['Test_Flag']==0]
test = all_df[all_df['Test_Flag']==1].reset_index(drop=True)
# 訓練データのSurvivedをtargetにする
target = train['Survived']
# 今回学習に用いないカラムを削除
drop_col = ['PassengerId','Age','Ticket', 'Fare','Cabin',
'Test_Flag','Name','Survived']
train = train.drop(drop_col,axis=1)
test = test.drop(drop_col,axis=1)
# 訓練データの一部を分割し検証データを作成
X_train,X_val,y_train,y_val = train_test_split(train,target,test_size=0.2,shuffle=True,random_state=0)
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score
model = LogisticRegression()
model.fit(X_train,y_train)
# 訓練データに対しての予測を行い、正答率を算出
y_pred = model.predict(X_train)
print(accuracy_score(y_train,y_pred)) #結果が0.800561797752809
4 特徴量の追加
4-1 新しい特徴量
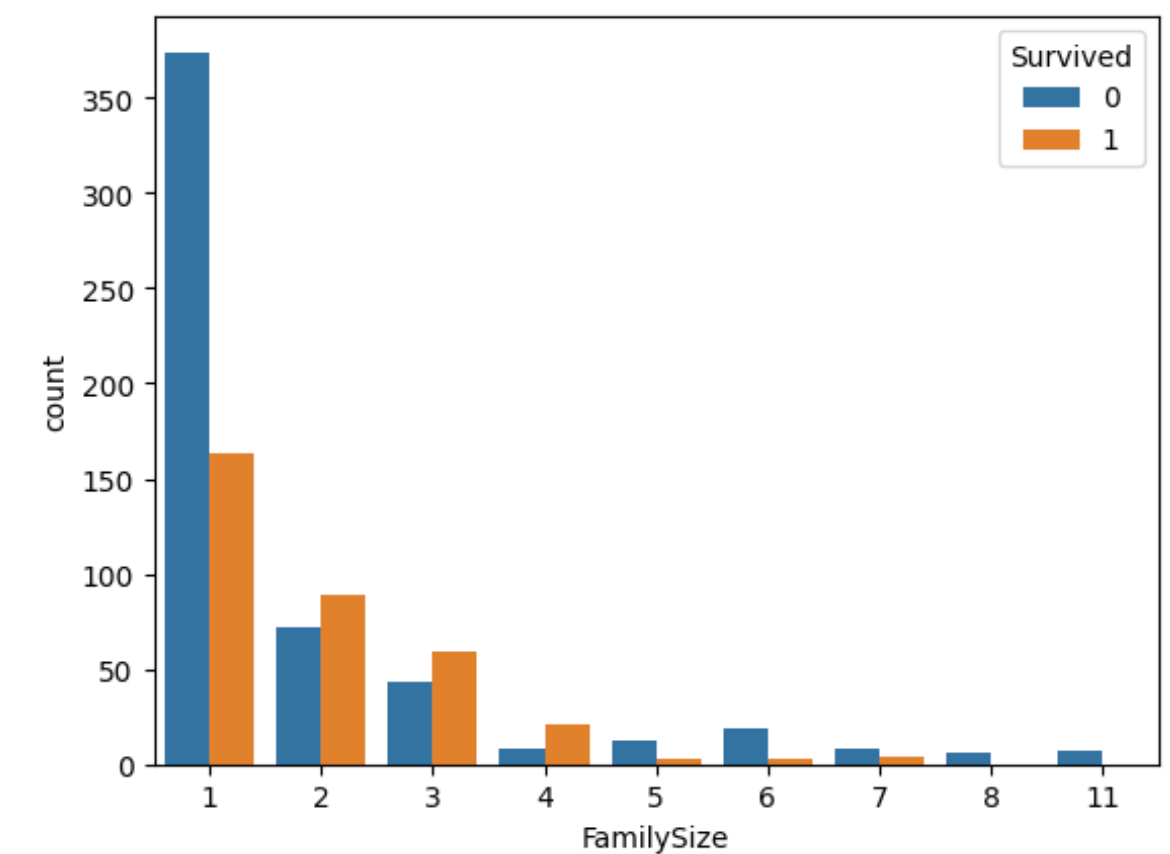
以前の分析から家族(SidSp,Parch)の人数と生存率には関係性があるので利用する。同乗した家族の人数(FamilySize)という新たな特徴量を作成し可視化する。
# 同乗した家族の人数 = 兄弟・配偶者の人数 + 両親・子供の人数 + 本人
train_df['FamilySize'] = train_df['SibSp'] + train_df['Parch'] +1
# FamilySizeと生存率の関係を可視化
sns.countplot(x='FamilySize',hue='Survived',data=train_df)
plt.legend(title='Survived',loc='upper right')
plt.show()
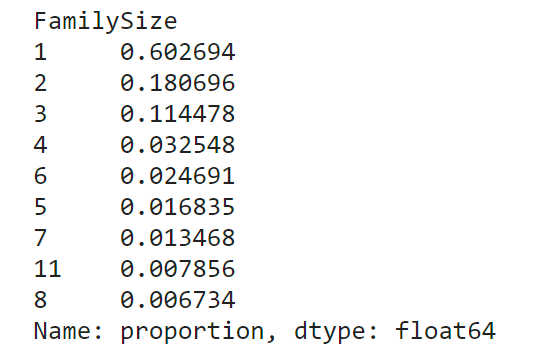
# 家族人数毎のデータに含まれる割合
display(train_df['FamilySize'].value_counts(ascending=False,normalize=True))
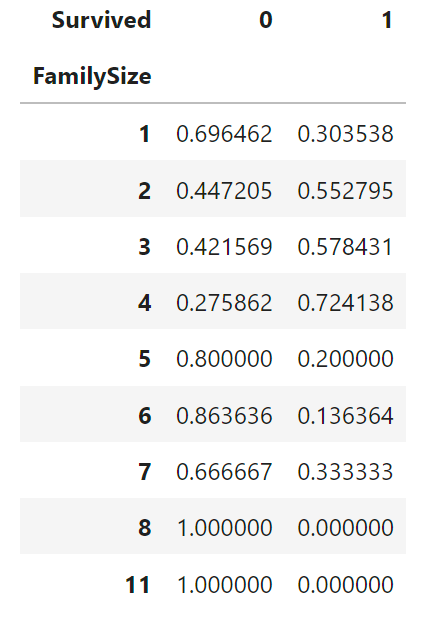
# 家族人数毎の生存率
display(pd.crosstab(train_df['FamilySize'],train_df['Survived'],normalize='index'))
- 全体に占める1人で乗船した人の割合は多いが生存率は低い。2~4人の家族で乗船した場合は生存率が高い。5人以上の家族で乗船した場合は生存率は低い。
- これにより1人で乗船した人が全体の6割、2~4人家族で乗船したのは全体の約3割で生存率は5割を超えている。5人以上で乗船した割合が全体の約1割で生存率は1~3割程度。
4-2 別の角度からデータを見てみる
1人で乗船した人か2人以上で乗船したかに軸を変えてみる。
# 1人で乗船した人のカテゴリーを作成
# 1人で乗船した人を1, 2人以上で乗船した人を0
train_df['Alone'] = train_df['FamilySize'].map(lambda s: 1 if s == 1 else 0)
# 「1人で乗船した人か2人以上で乗船した人か」と生存率の関係を可視化
sns.countplot(x='Alone',hue='Survived',data=train_df)
plt.legend(title='Suvived',loc='upper right')
plt.show()
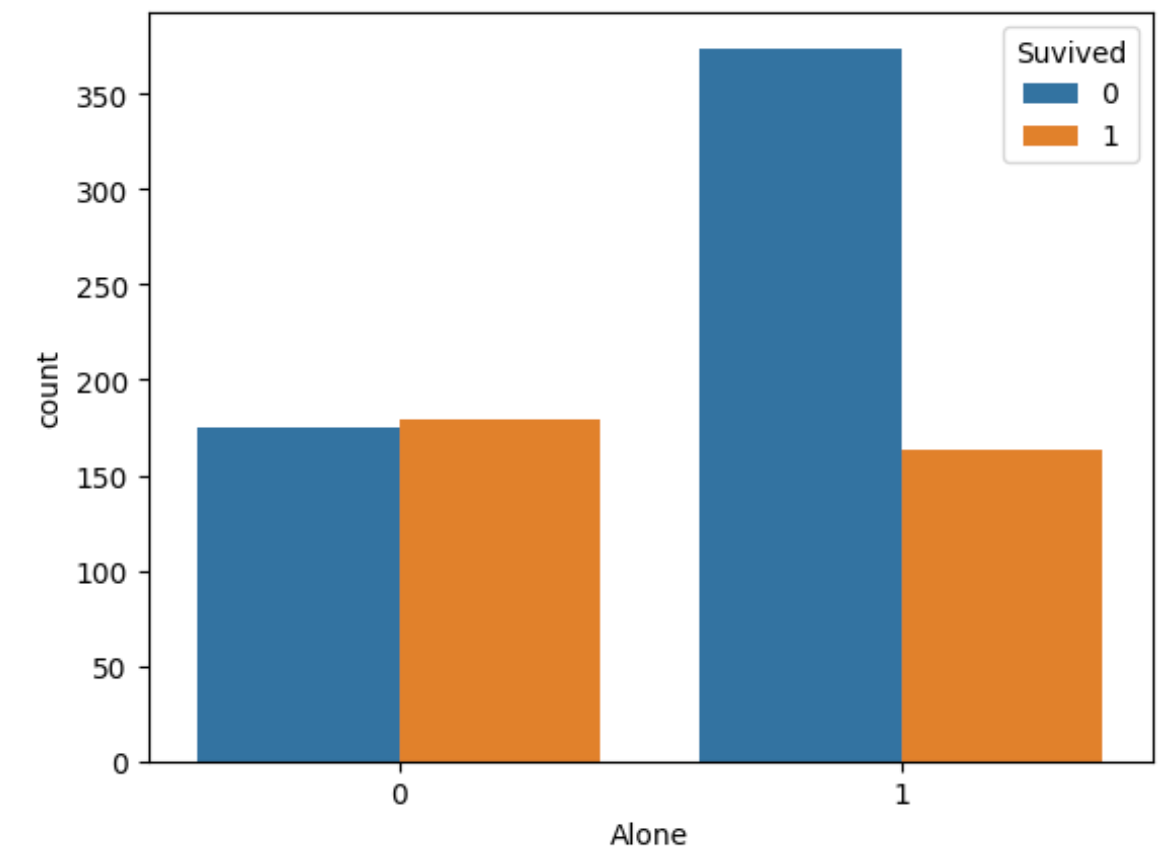
グラフから1人で乗船する場合は生存率は低く、2人以上なら約5割もある。この新しい特徴量の追加したコードが
train_df = pd.read_csv('/kaggle/input/titanic/train.csv')
test_df = pd.read_csv('/kaggle/input/titanic/test.csv')
sample_sub = pd.read_csv('/kaggle/input/titanic/gender_submission.csv')
all_df = pd.concat([train_df,test_df],axis=0).reset_index(drop=True)
all_df['Test_Flag'] = 0
all_df.loc[train_df.shape[0]: , 'Test_Flag'] = 1
#特徴量作成
all_df['Age'] = all_df['Age'].fillna(all_df['Age'].median())
all_df['Fare'] = all_df['Fare'].fillna(all_df['Fare'].median())
all_df['Embarked'] = all_df['Embarked'].fillna('NaN')
all_df['FareBand'] = pd.qcut(all_df['Fare'],4)
all_df['AgeBand'] = pd.qcut(all_df['Age'],4)
# 同乗した家族の人数を追加
all_df['FamilySize'] = all_df['SibSp'] + all_df['Parch'] + 1
# 同乗した家族の人数をもとに分割
all_df['Alone'] = all_df['FamilySize'].map(lambda s : 1 if s == 1 else 0)
all_df['Medf'] = all_df['FamilySize'].map(lambda s : 1 if 2 <= s <= 4 else 0)
all_df['LargeF'] = all_df['FamilySize'].map(lambda s : 1 if s >= 5 else 0)
# カテゴリカル変数をOne-Hot Encodeingで数値値
all_df = pd.get_dummies(all_df,columns=['Sex','Pclass'])
all_df = pd.get_dummies(all_df,columns=['AgeBand','FareBand','Embarked'])
from sklearn.model_selection import train_test_split
# 結合していた訓練データとテストデータを元に戻す
train = all_df[all_df['Test_Flag']==0]
test = all_df[all_df['Test_Flag']==1].reset_index(drop=True)
target = train['Survived']
# 今回、学習に用いないカラムを削除
drop_col = [
'PassengerId','Age',
'Ticket',
'Fare','Cabin',
'Test_Flag','Name','Survived'
]
train = train.drop(drop_col, axis=1)
test = test.drop(drop_col, axis=1)
X_train,X_val,y_train,y_val = train_test_split(train,target,test_size=0.2,shuffle=True,random_state=0)
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score
model = LogisticRegression()
model.fit(X_train,y_train)
y_pred = model.predict(X_train)
print(accuracy_score(y_train,y_pred)) #結果が0.8089887640449438
前回の結果が0.800561797752809 今回が0.8089887640449438とわずかに上昇。他の方法を考えます。
4-3 アルゴリズムの変更
今まではホールドアウト法でしたが今回はKFold使用しロジスティック回帰、SVM,RandomForest、LightGBMの4つのアルゴリズムを採用して検証してみます。
# n_splits で分割数が指定でき今回は3つに分割
cv = KFold(n_splits=3,random_state=0,shuffle=True)
train_acc_list = []
val_acc_list = []
# fold毎に学習データのインデックスと評価データのインデックス取得する
for i, (tun_index,val_index) in enumerate(cv.split(train,target)):
print(f'Fold : {i}')
X_train ,X_val = train.loc[trn_index], train.loc[val_index]
y_train ,y_val = target[trn_index], target[val_index]
model=LogisticRegression()
model.fit(X_train,y_train)
y_pred = model.predict(X_train)
train_acc = accuracy_score(y_train,y_pred)
train_acc_list.append(train_acc)
y_pred_val = model.predict(X_val)
val_acc = accuracy_score(y_val,y_pred_val)
print(val_acc)
val_acc_list.append(val_acc)
print('-'*10 + 'Result' + '-'*10)
print(f'Train_acc : {train_acc_list},Ave : {np.mean(train_acc_list)}')
print(f'Valid_acc : {val_acc_list}, Ave : {np.mean(val_acc_list)}')
ロジスティック回帰の結果が
Fold : 0
0.8148148148148148
Fold : 1
0.8215488215488216
Fold : 2
0.8013468013468014
----------Result----------
Train_acc : [0.8134642356241234, 0.8134642356241234, 0.8134642356241234],Ave : 0.8134642356241234
Valid_acc : [0.8148148148148148, 0.8215488215488216, 0.8013468013468014], Ave : 0.8125701459034792
cv = KFold(n_splits=3,random_state=0,shuffle=True)
train_acc_list = []
val_acc_list = []
for i, (tun_index,val_index) in enumerate(cv.split(train,target)):
print(f'Fold : {i}')
X_train ,X_val = train.loc[trn_index], train.loc[val_index]
y_train ,y_val = target[trn_index], target[val_index]
model=LogisticRegression()
model.fit(X_train,y_train)
y_pred = model.predict(X_train)
train_acc = accuracy_score(y_train,y_pred)
train_acc_list.append(train_acc)
y_pred_val = model.predict(X_val)
val_acc = accuracy_score(y_val,y_pred_val)
print(val_acc)
val_acc_list.append(val_acc)
print('-'*10 + 'Result' + '-'*10)
print(f'Train_acc : {train_acc_list},Ave : {np.mean(train_acc_list)}')
print(f'Valid_acc : {val_acc_list}, Ave : {np.mean(val_acc_list)}')
SVMの結果が
Fold : 0
0.8367003367003367
0.8114478114478114
Fold : 1
0.8148148148148148
0.8080808080808081
Fold : 2
0.8265993265993266
0.8080808080808081
----------Result----------
Train_acc : [0.8367003367003367, 0.8148148148148148, 0.8265993265993266] , Ave : 0.8260381593714926
Valid_acc : [0.8114478114478114, 0.8080808080808081, 0.8080808080808081] , Ave : 0.8092031425364757
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import accuracy_score
# データを読み込む
train_df = pd.read_csv('/kaggle/input/titanic/train.csv')
test_df = pd.read_csv('/kaggle/input/titanic/test.csv')
# 特徴量作成
# 結合していた訓練データとテストデータを元に戻す
train = all_df[all_df['Test_Flag'] == 0]
test = all_df[all_df['Test_Flag'] == 1].reset_index(drop=True)
target = train['Survived']
# 不要なカラムを削除
drop_col = [
'PassengerId', 'Age', 'Ticket', 'Fare', 'Cabin',
'Test_Flag', 'Name', 'Survived'
]
train = train.drop(drop_col, axis=1)
test = test.drop(drop_col, axis=1)
# 訓練データと評価データに分割
X_train, X_val, y_train, y_val = train_test_split(train, target, test_size=0.2, shuffle=True, random_state=0)
# ランダムフォレストモデルの作成と訓練
model = RandomForestClassifier(random_state=0)
model.fit(X_train, y_train)
# トレーニングデータでの予測
y_pred_train = model.predict(X_train)
train_acc = accuracy_score(y_train, y_pred_train)
print(f"Train Accuracy: {train_acc}")
# 評価データでの予測
y_pred_val = model.predict(X_val)
val_acc = accuracy_score(y_val, y_pred_val)
print(f"Validation Accuracy: {val_acc}")
ランダムフォレストの結果が
Train Accuracy: 0.8862359550561798
Validation Accuracy: 0.8324022346368715
import lightgbm as lgb
cv = KFold(n_splits=3, random_state=0, shuffle=True)
train_acc_list = []
val_acc_list = []
lgb_params = {
"objective":"binary",
"metric":"binary_error",
"seed":0,}
# LightGBMがカラム名に[]や{}を含んでいるとエラーが出るので、変更する
rename_dict = {
'AgeBand_(0.169, 22.0]' : 'AgeBand_1',
'AgeBand_(22.0, 28.0]' : 'AgeBand_2',
'AgeBand_(28.0, 35.0]' : 'AgeBand_3',
'AgeBand_(35.0, 80.0]' : 'AgeBand_4',
'FareBand_(-0.001, 7.896]' : 'FareBand_1',
'FareBand_(7.896, 14.454]' : 'FareBand_2',
'FareBand_(14.454, 31.275]' : 'FareBand_3',
'FareBand_(31.275, 512.329]' : 'FareBand_4'
}
for i ,(trn_index, val_index) in enumerate(cv.split(train, target)):
print(f'Fold : {i}')
# データ全体(Xとy)を学習データと評価データに分割
X_train ,X_val = train.loc[trn_index].rename(columns =rename_dict), train.loc[val_index].rename(columns =rename_dict)
y_train ,y_val = target[trn_index], target[val_index]
# LigthGBM用のデータセットを定義
lgb_train = lgb.Dataset(X_train, y_train)
lgb_valid = lgb.Dataset(X_val, y_val)
model = lgb.train(
params = lgb_params,
train_set = lgb_train,
valid_sets = [lgb_train, lgb_valid],
verbose_eval = 100 ,
early_stopping_rounds=10
)
y_pred = model.predict(X_train)
train_acc = accuracy_score(
# y_predは0〜1の確率になっています。
y_train, np.where(y_pred>=0.5, 1, 0)
)
print(train_acc)
train_acc_list.append(train_acc)
y_pred_val = model.predict(X_val)
val_acc = accuracy_score(
y_val, np.where(y_pred_val>=0.5, 1, 0)
)
print(val_acc)
val_acc_list.append(val_acc)
print('-'*10 + 'Result' +'-'*10)
print(f'Train_acc : {train_acc_list} , Ave : {np.mean(train_acc_list)}')
print(f'Valid_acc : {val_acc_list} , Ave : {np.mean(val_acc_list)}')
LightGBMの結果が
Train_acc : [0.8282828282828283, 0.7828282828282829, 0.8451178451178452] , Ave : 0.8187429854096521
Valid_acc : [0.8215488215488216, 0.835016835016835, 0.7912457912457912] , Ave : 0.8159371492704826
| アルゴリズム | TrainのAve結果 |
|---|---|
| ロジスティック回帰 | 0.8134642356241234 |
| SVM | 0.8260381593714926 |
| RandomForestClassifier | 0.8862359550561798 |
| LightGM | 0.8187429854096521 |
以上の結果訓練データの結果ならランダムフォレスト
が圧倒的ですね。
4-4 名前についての特徴量とは
名前自体ではなくMr,Miss等の敬称について調べてみます。敬称は性別や結婚の有無によって異なる。正規表現で該当箇所を抽出し訓練データとテストデータの重複をベン図で可視化します。
col_name = 'Name'
venn2(subsets=(set(train_df[col_name].unique()),set(test_df[col_name].unique())),set_labels=('Train','Test'),)
plt.show()
display(train_df['Name'].head(10))
print('---欠損---')
print(all_df['Name'].isna().sum())
0 Braund, Mr. Owen Harris
1 Cumings, Mrs. John Bradley (Florence Briggs Th...
2 Heikkinen, Miss. Laina
3 Futrelle, Mrs. Jacques Heath (Lily May Peel)
4 Allen, Mr. William Henry
5 Moran, Mr. James
6 McCarthy, Mr. Timothy J
7 Palsson, Master. Gosta Leonard
8 Johnson, Mrs. Oscar W (Elisabeth Vilhelmina Berg)
9 Nasser, Mrs. Nicholas (Adele Achem)
Name: Name, dtype: object
---欠損---
0
# 訓練データとテストデータの重複をベン図で可視化
venn2(subsets=(set(train_df.Name.str.extract(' ([A-Za-z]+)\.', expand=False)),set(test_df.Name.str.extract(' ([A-Za-z]+)\.' ,expand=False))),set_labels=('Train', 'Test'),)
plt.show()
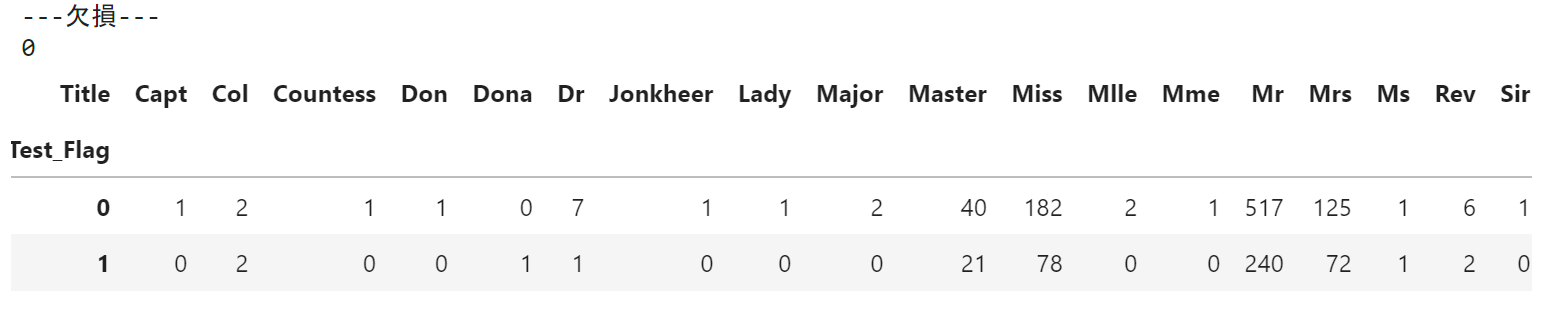
# 敬称を抽出したカラム「Title」を追加しTitleの欠損数を表示、訓練データとテストデータ別に各敬称の登場回数を集計。
all_df['Title'] = all_df.Name.str.extract(' ([A-Za-z]+)\.',expand=False)
# 「Title」の欠損数を表示
print('---欠損---')
print(all_df['Title'].isna().sum())
# 訓練データとテストデータ別に、各敬称の登場回数を集計
# Test_Flag=0 が訓練データ
# Test_Flag=1 がテストデータ
display(pd.crosstab(all_df['Test_Flag'],all_df['Title']))
- 敬称に欠損なし。
- 上記のベン図と集計からテストデータのみ'Dona'という敬称がある。
- 出現頻度はMr,Miss,Mrs,Masterが多くこれら以外の出現は低い。
#更に調べるために敬称毎の平均年齢を算出
all_df.groupby('Title')['Age'].mean()
| Title | 平均年齢 |
|---|---|
| Capt | 70.000000 |
| Col | 54.000000 |
| Countess | 33.000000 |
| Don | 40.000000 |
| Dona | 39.000000 |
| Dr | 41.625000 |
| Jonkheer | 38.000000 |
| Lady | 48.000000 |
| Major | 48.500000 |
| Master | 8.435738 |
| Miss | 22.971500 |
| Mlle | 24.000000 |
| Mme | 24.000000 |
| Mr | 31.263540 |
| Mrs | 35.761421 |
| Ms | 28.000000 |
| Rev | 41.250000 |
| Sir | 49.000000 |
# 敬称毎の生存率の集計
display(all_df[all_df['Test_Flag']==0].groupby('Title')['Survived'].agg(['mean','count']))
| Title | mean | count |
|---|---|---|
| Capt | 0.000000 | 1 |
| Col | 0.500000 | 2 |
| Countess | 1.000000 | 1 |
| Don | 0.000000 | 1 |
| Dr | 0.428571 | 7 |
| Jonkheer | 0.000000 | 1 |
| Lady | 1.000000 | 1 |
| Major | 0.500000 | 2 |
| Master | 0.575000 | 40 |
| Miss | 0.697802 | 182 |
| Mlle | 1.000000 | 2 |
| Mme | 1.000000 | 1 |
| Mr | 0.156673 | 517 |
| Mrs | 0.792000 | 125 |
| Ms | 1.000000 | 1 |
| Rev | 0.000000 | 6 |
| Sir | 1.000000 | 1 |
上記2つの表より、
- 敬称Masterの平均年齢は約8.4歳とおそらく子供を表す敬称かと思われる。
- 敬称別生存率だとMiss,Mrs,Masterの生存率が高くMrの生存率がかなり悪いことが確認できる。
4-5 敬称による新たな特徴量
各データの敬称を抽出しMaster,Miss,Mr,Mrs,それ以外の5つのカテゴリーを持つカテゴリー変数を作成。
# 敬称を抽出
all_df['Title'] = all_df.Name.str.extract(' ([A^Za-z]+)\.',expand=False)
# 敬称を該当するカテゴリに変換する関数を定義
def prepro_name_title(Title):
if Title == 'Master':
return 0
elif Title == 'Miss':
return 1
elif Title == 'Mr':
return 2
elif Title == 'Mrs':
return 3
else:
return 4
# Titelをカテゴリカル変数化
all_df['Title_Encode'] = all_df['Title'].map(prepro_name_title)
今までのワンホットエンコーディングはカテゴリーつ一つに対し特徴量を作成するため含まれるカテゴリが多いと特徴量が膨大になってしまう問題があります。今回はLabel-Encodingを用いてみる。この手法だとその数値の大きさをモデルが考慮してしまう可能性があるのでロジスティック回帰やニューラルネットワークは使用できません。今回は木構造がベースになっているのでLightGBMを用います。
from sklearn.model_selection import KFold
import lightgbm as lgb
from sklearn.metrics import accuracy_score
cv = KFold(n_splits=3, random_state=0, shuffle=True)
train_acc_list = []
val_acc_list = []
models = []
# ハイパーパラメータを定義
lgb_params = {
"objective":"binary",
"metric": "binary_error",
"force_row_wise" : True,
"seed" : 0,
'learning_rate': 0.1,
'min_data_in_leaf': 5
}
# LightGBMがカラム名に[]や{}を含んでいるとエラーが出るので、変更する
rename_dict = {
'AgeBand_(0.169, 22.0]' : 'AgeBand_1',
'AgeBand_(22.0, 28.0]' : 'AgeBand_2',
'AgeBand_(28.0, 35.0]' : 'AgeBand_3',
'AgeBand_(35.0, 80.0]' : 'AgeBand_4',
'FareBand_(-0.001, 7.896]' : 'FareBand_1',
'FareBand_(7.896, 14.454]' : 'FareBand_2',
'FareBand_(14.454, 31.275]' : 'FareBand_3',
'FareBand_(31.275, 512.329]' : 'FareBand_4'
}
for i ,(trn_index, val_index) in enumerate(cv.split(train, target)):
print(f'Fold : {i}')
X_train ,X_val = train.loc[trn_index].rename( columns =rename_dict), train.loc[val_index].rename( columns =rename_dict)
y_train ,y_val = target[trn_index], target[val_index]
# LigthGBM用のデータセットを定義
lgb_train = lgb.Dataset(X_train, y_train)
lgb_valid = lgb.Dataset(X_val, y_val)
model = lgb.train(
params = lgb_params,
train_set = lgb_train,
valid_sets = [lgb_train, lgb_valid],
verbose_eval = 100 ,
early_stopping_rounds=10
)
y_pred = model.predict(X_train)
train_acc = accuracy_score(
y_train, np.where(y_pred>=0.5, 1, 0)
)
print(train_acc)
train_acc_list.append(train_acc)
y_pred_val = model.predict(X_val)
val_acc = accuracy_score(
y_val, np.where(y_pred_val>=0.5, 1, 0)
)
print(val_acc)
val_acc_list.append(val_acc)
models.append(model)
print('-'*10 + 'Result' +'-'*10)
print(f'Train_acc : {train_acc_list} , Ave : {np.mean(train_acc_list)}')
print(f'Valid_acc : {val_acc_list} , Ave : {np.mean(val_acc_list)}')
----------Result----------
Train_acc : [0.835016835016835, 0.797979797979798, 0.8686868686868687] ,
Ave : 0.8338945005611672
Valid_acc : [0.8047138047138047, 0.8619528619528619, 0.7744107744107744] ,
Ave : 0.8136924803591471
5 ハイパーパラメータチューニングとアンサンブル
ハイパーパラメータチューニング
今回はベイズ最適化を使います。探索ライブラリのoptunaを用いてLightGBMのハイパーパラメータを探索します。
import optuna
def objective(trial):
# 今までと同様にクロスバリデーションでモデルを評価します
cv = KFold(n_splits=3, random_state=0, shuffle=True)
val_acc_list = []
lgb_params = {
"objective":"binary",
"metric": "binary_error",
"force_row_wise" : True,
"seed" : 0,
'learning_rate': 0.1,
'min_data_in_leaf': 5,
# optunaでパラメータ探索する区間を指定
"max_depth": trial.suggest_int('max_depth', 3, 20),
"num_leaves":trial.suggest_int('num_leaves',21,41),
"learning_rate":trial.suggest_uniform('learning_rate',0.1,0.2)
}
# LightGBMがカラム名に[]や{}を含んでいるとエラーが出るので、変更する
rename_dict = {
'AgeBand_(0.169, 22.0]' : 'AgeBand_1',
'AgeBand_(22.0, 28.0]' : 'AgeBand_2',
'AgeBand_(28.0, 35.0]' : 'AgeBand_3',
'AgeBand_(35.0, 80.0]' : 'AgeBand_4',
'FareBand_(-0.001, 7.896]' : 'FareBand_1',
'FareBand_(7.896, 14.454]' : 'FareBand_2',
'FareBand_(14.454, 31.275]' : 'FareBand_3',
'FareBand_(31.275, 512.329]' : 'FareBand_4'
}
# KFold で学習させる
for i ,(trn_index, val_index) in enumerate(cv.split(train, target)):
print(f'Fold : {i}')
X_train ,X_val = train.loc[trn_index].rename( columns =rename_dict), train.loc[val_index].rename( columns =rename_dict)
y_train ,y_val = target[trn_index], target[val_index]
lgb_train = lgb.Dataset(X_train, y_train)
lgb_valid = lgb.Dataset(X_val, y_val)
model = lgb.train(
params = lgb_params,
train_set = lgb_train,
valid_sets = [lgb_train, lgb_valid],
verbose_eval = -1 ,
early_stopping_rounds=10
)
y_pred = model.predict(X_train)
y_pred_val = model.predict(X_val)
val_acc = accuracy_score(
y_val, np.where(y_pred_val>=0.5, 1, 0)
)
val_acc_list.append(val_acc)
# 最適化の指標にする値を返り値に設定
return np.mean(val_acc_list)
# 最適化タスクを定義
# 引数のdirectionには最適化する指標を最大化したいのか、最小化したいのかを指定
study = optuna.create_study(direction="maximize")
# 最適化を実行します。この時の探索の試行回数をn_trialsで指定
study.optimize(objective, n_trials=100)
# 最適なパラメータとその時の精度を表示
print("best_value", study.best_value)
print("best_params", study.best_params)
# 最適化の過程を表示 & 可視化
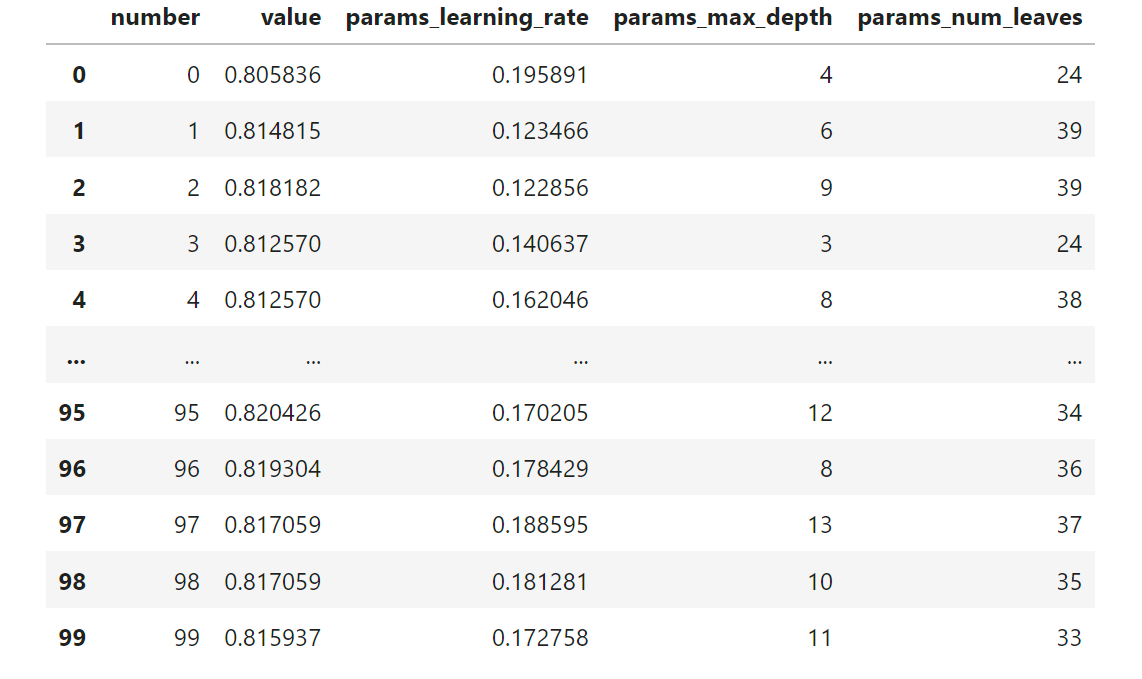
optuna_log_df = study.trials_dataframe(attrs=("number", "value", "params"))
display(optuna_log_df)
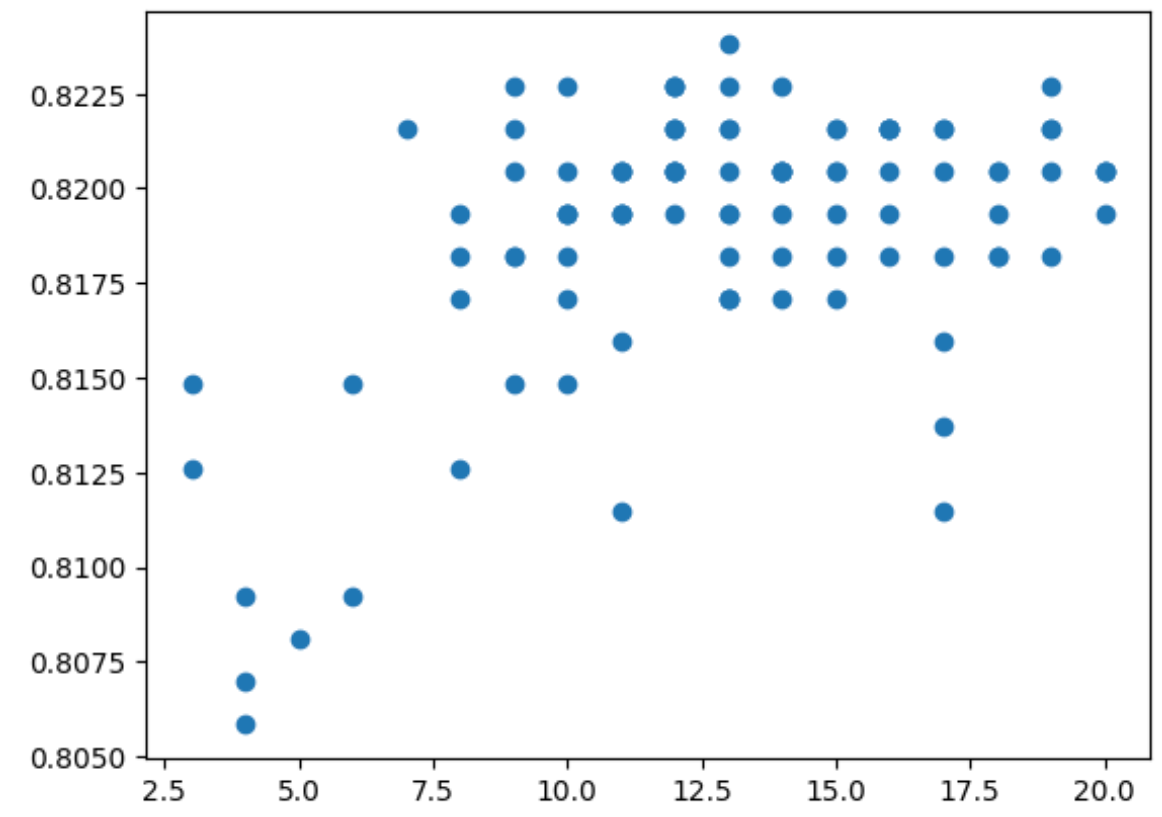
# max_depthと正答率の関係を可視化
plt.scatter(optuna_log_df["params_max_depth"], optuna_log_df["value"])
best_value 0.8237934904601572
best_params {'max_depth': 13, 'num_leaves': 35, 'learning_rate': 0.17722952605537962}
アンサンブル
アンサンブルとは複数のモデルを用いて予測を行うことで、一般的に多様なモデルの結果を組み合わせることで汎化性能が向上する場合があります。
今回はLightGBM、Random Forest、ロジスティック回帰、SVMの4つのモデルで予測を行います。(MLPも使用したいのですがなぜかエラーになりどうしても進めないので今回はMLPは省きます。)
まずはハイパーパラメータチューニングなしで予測すると
import pandas as pd
import numpy as np
from sklearn.ensemble import RandomForestClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from sklearn.model_selection import KFold
from sklearn.metrics import accuracy_score
import lightgbm as lgb
import random
import scipy.stats as stats
import os
# 乱数を固定
np.random.seed(0)
random.seed(0)
os.environ["PYTHONHASHSEED"] = "0"
train_df = pd.read_csv('/kaggle/input/titanic/train.csv')
test_df = pd.read_csv('/kaggle/input/titanic/test.csv')
sample_sub = pd.read_csv('/kaggle/input/titanic/gender_submission.csv')
all_df = pd.concat([train_df, test_df], axis=0).reset_index(drop=True)
all_df['Test_Flag'] = 0
all_df.loc[train_df.shape[0]:, 'Test_Flag'] = 1
all_df['Age'] = all_df['Age'].fillna(all_df['Age'].median())
all_df['Fare'] = all_df['Fare'].fillna(all_df['Fare'].median())
all_df['Embarked'] = all_df['Embarked'].fillna('NaN')
all_df['FareBand'] = pd.qcut(all_df['Fare'], 4)
all_df['AgeBand'] = pd.qcut(all_df['Age'], 4)
all_df['FamilySize'] = all_df['SibSp'] + all_df['Parch'] + 1
all_df['MedF'] = all_df['FamilySize'].map(lambda s: 1 if 2 <= s <= 4 else 0)
all_df['LargeF'] = all_df['FamilySize'].map(lambda s: 1 if s >= 5 else 0)
all_df['Alone'] = all_df['FamilySize'].map(lambda s: 1 if s == 1 else 0)
all_df['Title'] = all_df.Name.str.extract(' ([A-Za-z]+)\.', expand=False)
def prepro_name_title(Title):
if Title == 'Master':
return 0
elif Title == 'Miss':
return 1
elif Title == 'Mr':
return 2
elif Title == 'Mrs':
return 3
else:
return 4
all_df['Title_Encode'] = all_df['Title'].map(prepro_name_title)
all_df = pd.get_dummies(all_df, columns=["Sex", "Pclass"])
all_df = pd.get_dummies(all_df, columns=['AgeBand', 'FareBand', 'Embarked'])
train = all_df[all_df['Test_Flag'] == 0]
test = all_df[all_df['Test_Flag'] == 1].reset_index(drop=True)
target = train['Survived']
drop_col = [
'PassengerId', 'Age', 'Ticket', 'Title', 'Fare', 'Cabin', 'Test_Flag', 'Name', 'Survived'
]
train = train.drop(drop_col, axis=1)
test = test.drop(drop_col, axis=1)
cv = KFold(n_splits=3, random_state=0, shuffle=True)
train_acc_list = []
val_acc_list = []
lgb_params = {
"objective": "binary",
"metric": "binary_error",
"force_row_wise": True,
"seed": 0,
'learning_rate': 0.1,
'min_data_in_leaf': 5,
'max_depth': 16
}
rename_dict = {
'AgeBand_(0.169, 22.0]': 'AgeBand_1',
'AgeBand_(22.0, 28.0]': 'AgeBand_2',
'AgeBand_(28.0, 35.0]': 'AgeBand_3',
'AgeBand_(35.0, 80.0]': 'AgeBand_4',
'FareBand_(-0.001, 7.896]': 'FareBand_1',
'FareBand_(7.896, 14.454]': 'FareBand_2',
'FareBand_(14.454, 31.275]': 'FareBand_3',
'FareBand_(31.275, 512.329]': 'FareBand_4'
}
for i, (trn_index, val_index) in enumerate(cv.split(train, target)):
print(f'Fold : {i}')
X_train, X_val = train.loc[trn_index].rename(columns=rename_dict), train.loc[val_index].rename(columns=rename_dict)
y_train, y_val = target.iloc[trn_index], target.iloc[val_index]
lgb_train = lgb.Dataset(X_train, y_train)
lgb_valid = lgb.Dataset(X_val, y_val)
model_lgb = lgb.train(
params=lgb_params,
train_set=lgb_train,
valid_sets=[lgb_train, lgb_valid],
verbose_eval=0,
early_stopping_rounds=10
)
print('-' * 10 + ' Start_rf ' + '-' * 10)
model_rf = RandomForestClassifier(
random_state=0, max_depth=15,
min_samples_leaf=5, min_samples_split=5
)
model_rf.fit(X_train, y_train)
print('-' * 10 + ' Start_rogi ' + '-' * 10)
model_rogi = LogisticRegression()
model_rogi.fit(X_train, y_train)
print('-' * 10 + ' Start_SVM ' + '-' * 10)
model_svm = SVC(random_state=0)
model_svm.fit(X_train, y_train)
train_pred = np.zeros((len(y_train), 4))
train_pred[:, 0] = np.where(model_lgb.predict(X_train) >= 0.5, 1, 0)
train_pred[:, 1] = model_rf.predict(X_train)
train_pred[:, 2] = model_rogi.predict(X_train)
train_pred[:, 3] = model_svm.predict(X_train)
train_acc = accuracy_score(
y_train, stats.mode(train_pred, axis=1)[0]
)
train_acc_list.append(train_acc)
val_pred = np.zeros((len(y_val), 4))
val_pred[:, 0] = np.where(model_lgb.predict(X_val) >= 0.5, 1, 0)
val_pred[:, 1] = model_rf.predict(X_val)
val_pred[:, 2] = model_rogi.predict(X_val)
val_pred[:, 3] = model_svm.predict(X_val)
val_acc = accuracy_score(
y_val, stats.mode(val_pred, axis=1)[0]
)
val_acc_list.append(val_acc)
print('-' * 10 + 'Result' + '-' * 10)
print(f'Train_acc : {train_acc_list}, Ave: {np.mean(train_acc_list)}')
print(f'Valid_acc : {val_acc_list}, Ave: {np.mean(val_acc_list)}')
Train_acc : [0.8417508417508418, 0.8316498316498316, 0.8585858585858586], Ave: 0.8439955106621774
Valid_acc : [0.8215488215488216, 0.8316498316498316, 0.8114478114478114], Ave: 0.8215488215488215
結果としては一番よくなっています。ここにさきほどLightGBMの最適なハイパーパラメータチューニングしたものを組み込むと
import pandas as pd
import numpy as np
from sklearn.ensemble import RandomForestClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from sklearn.model_selection import KFold
from sklearn.metrics import accuracy_score
import lightgbm as lgb
import random
import scipy.stats as stats
import os
# 乱数を固定
np.random.seed(0)
random.seed(0)
os.environ["PYTHONHASHSEED"] = "0"
train_df = pd.read_csv('/kaggle/input/titanic/train.csv')
test_df = pd.read_csv('/kaggle/input/titanic/test.csv')
sample_sub = pd.read_csv('/kaggle/input/titanic/gender_submission.csv')
all_df = pd.concat([train_df, test_df], axis=0).reset_index(drop=True)
all_df['Test_Flag'] = 0
all_df.loc[train_df.shape[0]:, 'Test_Flag'] = 1
all_df['Age'] = all_df['Age'].fillna(all_df['Age'].median())
all_df['Fare'] = all_df['Fare'].fillna(all_df['Fare'].median())
all_df['Embarked'] = all_df['Embarked'].fillna('NaN')
all_df['FareBand'] = pd.qcut(all_df['Fare'], 4)
all_df['AgeBand'] = pd.qcut(all_df['Age'], 4)
all_df['FamilySize'] = all_df['SibSp'] + all_df['Parch'] + 1
all_df['MedF'] = all_df['FamilySize'].map(lambda s: 1 if 2 <= s <= 4 else 0)
all_df['LargeF'] = all_df['FamilySize'].map(lambda s: 1 if s >= 5 else 0)
all_df['Alone'] = all_df['FamilySize'].map(lambda s: 1 if s == 1 else 0)
all_df['Title'] = all_df.Name.str.extract(' ([A-Za-z]+)\.', expand=False)
def prepro_name_title(Title):
if Title == 'Master':
return 0
elif Title == 'Miss':
return 1
elif Title == 'Mr':
return 2
elif Title == 'Mrs':
return 3
else:
return 4
all_df['Title_Encode'] = all_df['Title'].map(prepro_name_title)
all_df = pd.get_dummies(all_df, columns=["Sex", "Pclass"])
all_df = pd.get_dummies(all_df, columns=['AgeBand', 'FareBand', 'Embarked'])
train = all_df[all_df['Test_Flag'] == 0]
test = all_df[all_df['Test_Flag'] == 1].reset_index(drop=True)
target = train['Survived']
drop_col = [
'PassengerId', 'Age', 'Ticket', 'Title', 'Fare', 'Cabin', 'Test_Flag', 'Name', 'Survived'
]
train = train.drop(drop_col, axis=1)
test = test.drop(drop_col, axis=1)
cv = KFold(n_splits=3, random_state=0, shuffle=True)
train_acc_list = []
val_acc_list = []
lgb_params = {
"objective": "binary",
"metric": "binary_error",
"force_row_wise": True,
"seed": 0,
'learning_rate': 0.17722952605537962,
'num_leaves' : 35,
'min_data_in_leaf': 5,
'max_depth': 13
}
rename_dict = {
'AgeBand_(0.169, 22.0]': 'AgeBand_1',
'AgeBand_(22.0, 28.0]': 'AgeBand_2',
'AgeBand_(28.0, 35.0]': 'AgeBand_3',
'AgeBand_(35.0, 80.0]': 'AgeBand_4',
'FareBand_(-0.001, 7.896]': 'FareBand_1',
'FareBand_(7.896, 14.454]': 'FareBand_2',
'FareBand_(14.454, 31.275]': 'FareBand_3',
'FareBand_(31.275, 512.329]': 'FareBand_4'
}
for i, (trn_index, val_index) in enumerate(cv.split(train, target)):
print(f'Fold : {i}')
X_train, X_val = train.loc[trn_index].rename(columns=rename_dict), train.loc[val_index].rename(columns=rename_dict)
y_train, y_val = target.iloc[trn_index], target.iloc[val_index]
lgb_train = lgb.Dataset(X_train, y_train)
lgb_valid = lgb.Dataset(X_val, y_val)
model_lgb = lgb.train(
params=lgb_params,
train_set=lgb_train,
valid_sets=[lgb_train, lgb_valid],
verbose_eval=0,
early_stopping_rounds=10
)
print('-' * 10 + ' Start_rf ' + '-' * 10)
model_rf = RandomForestClassifier(
random_state=0, max_depth=15,
min_samples_leaf=5, min_samples_split=5
)
model_rf.fit(X_train, y_train)
print('-' * 10 + ' Start_rogi ' + '-' * 10)
model_rogi = LogisticRegression()
model_rogi.fit(X_train, y_train)
print('-' * 10 + ' Start_SVM ' + '-' * 10)
model_svm = SVC(random_state=0)
model_svm.fit(X_train, y_train)
train_pred = np.zeros((len(y_train), 4))
train_pred[:, 0] = np.where(model_lgb.predict(X_train) >= 0.5, 1, 0)
train_pred[:, 1] = model_rf.predict(X_train)
train_pred[:, 2] = model_rogi.predict(X_train)
train_pred[:, 3] = model_svm.predict(X_train)
train_acc = accuracy_score(
y_train, stats.mode(train_pred, axis=1)[0]
)
train_acc_list.append(train_acc)
val_pred = np.zeros((len(y_val), 4))
val_pred[:, 0] = np.where(model_lgb.predict(X_val) >= 0.5, 1, 0)
val_pred[:, 1] = model_rf.predict(X_val)
val_pred[:, 2] = model_rogi.predict(X_val)
val_pred[:, 3] = model_svm.predict(X_val)
val_acc = accuracy_score(
y_val, stats.mode(val_pred, axis=1)[0]
)
val_acc_list.append(val_acc)
print('-' * 10 + 'Result' + '-' * 10)
print(f'Train_acc : {train_acc_list}, Ave: {np.mean(train_acc_list)}')
print(f'Valid_acc : {val_acc_list}, Ave: {np.mean(val_acc_list)}')
Train_acc : [0.8417508417508418, 0.8316498316498316, 0.8636363636363636], Ave: 0.8456790123456791
Valid_acc : [0.8215488215488216, 0.8316498316498316, 0.8215488215488216], Ave: 0.8249158249158249
チューニング前:Train_acc Ave: 0.8439955106621774
チューニング後:Train_acc Ave: 0.8456790123456791
となりわずかですが0.0016835016835017だけ向上という結果でした。4つのモデルのうちの一つをチューニングしても総合の結果にはそれほど影響が出ていないことがわかりました。
今回はこれを提出用とします。
6感想
ほとんど講座の内容に沿うものでしたがかなり難しいですね。わからない部分を自分で調べながらでもかなり時間がかかりました。MLPモデルも使いたかったのですがどうしてもコードがエラーになり今回はMLP抜きでの進行です。
スコアが全然低いみたいなので今後もっと学習をすすめたら再挑戦してみたいなと思います。