コロナの感染具合をseabornのヒートマップで表示してみた
目的
コロナの各都道府県での広がり具合を視覚化する
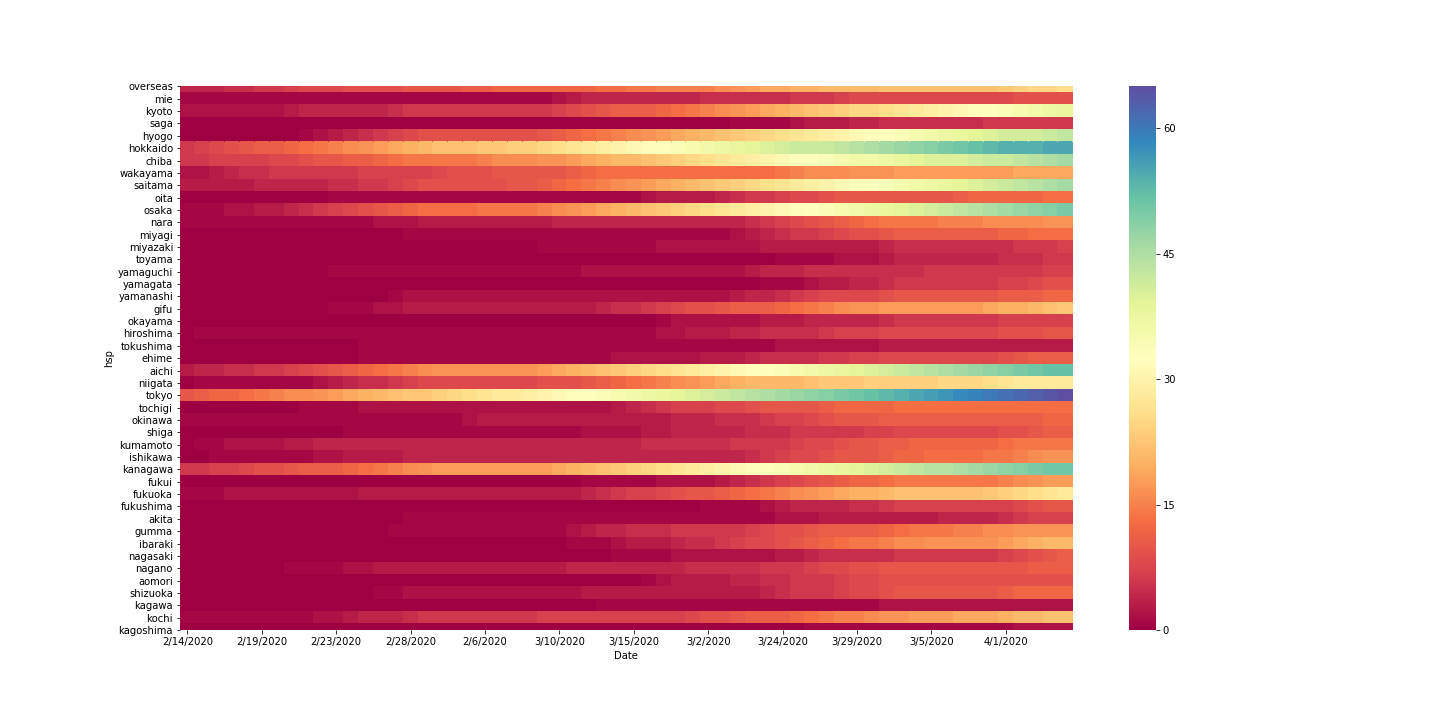
ゴールはこれです。
方法
pythonのseabornを使用する。
感染者数データ(4/5まで)はこちら。https://toyokeizai.net/sp/visual/tko/covid19/
都道府県名データはこちら。https://gist.github.com/mugifly/d6e68a516de4a008687c
いろいろまとめてこちら。https://github.com/kyasby/colona.git
ライブラリをインポート
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
%matplotlib inlineはおまじないです。
numpyはcumsum()のためにインポートします。
感染者数csvをインポート
df = pd.read_csv("COVID-19.csv")
df = df[["受診都道府県", "居住都道府県", "人数", "発症日", "確定日"]]
df = df.rename(columns={"年代":"age", "性別":"sex", "受診都道府県":"hsp", "居住都道府県":"house"})
df
必要なカラムを抜き出し、同時にリネームします。
df[]にリストを渡すことで、そのカラムだけ抜き出すことができます。
df.rename(columns={"old_columns_name":"new_name"},index={"old_index_name":"new_name"})
などと辞書を渡すことで、カラム名やインデックス名を変更できます。
*hspはhospitalです。
日付作り
for i, row in df.iterrows():
if type(row["発症日"])==float:
df.at[i, "発症日"] = row["確定日"]
else:
pass
df = df.rename(columns = {"発症日":"Date"})
ヒートマップの横軸を日付にしたいので、日付を取得します。
しかし、以下のように「発症日」にはNaNが含まれているので、その場合は「確定日」で置き換えます。
| hsp | house | 人数 | 発症日 | 確定日 |
|---|---|---|---|---|
| 神奈川県 | 神奈川県 | 1 | 1/3/2020 | 1/15/2020 |
| 東京都 | 中華人民共和国 | 1 | 1/14/2020 | 1/24/2020 |
| 東京都 | 中華人民共和国 | 1 | 1/21/2020 | 1/25/2020 |
| 大阪府 | 大阪府 | 1 | 1/20/2020 | 1/29/2020 |
| 不明 | 中華人民共和国 | 1 | 1/29/2020 | 1/30/2020 |
| 千葉県 | 中華人民共和国 | 1 | NaN | 1/30/2020 |
最後に、カラム名を「Date」に変更します。
NaNの判定を
type(row["発症日"])==float
このように書きましたが、もっと良い書き方があれば是非教えてください。
都道府県のCSVをインポート
todofuken = pd.read_csv("japan.csv", header=None)[0]
hspの一部を置き換える
df["hsp"].value_counts()
で、「hsp」の中身を確認すると、「羽田空港」や「不明」があることがわかります。
df["hsp"]= df["hsp"].apply(lambda x : "その他" if x not in list(todofuken) else x)
applyとラムダ関数を使い、df["hsp"]の中身を一部書き換えます。
都道府県名リストにない場合は、「その他」、ある場合はそのままの都道府県を入れます
applyとラムダ関数を使うときは、おそらくelseがないと構文エラーになります。(未確認)注意してください。
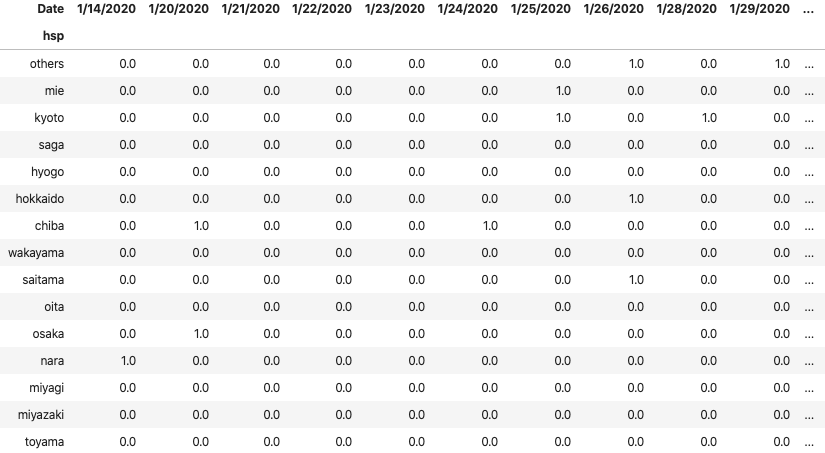
ここまででdfはこのようになっています。
| hsp | house | 人数 | Date | 確定日 |
|---|---|---|---|---|
| 神奈川県 | 神奈川県 | 1 | 1/3/2020 | 1/15/2020 |
| 東京都 | 中華人民共和国 | 1 | 1/14/2020 | 1/24/2020 |
| 東京都 | 中華人民共和国 | 1 | 1/21/2020 | 1/25/2020 |
| 愛知県 | 中華人民共和国 | 1 | 1/23/2020 | 1/26/2020 |
| 愛知県 | 中華人民共和国 | 1 | 1/22/2020 | 1/28/2020 |
| 奈良県 | 奈良県 | 1 | 1/14/2020 | 1/28/2020 |
| 北海道 | 中華人民共和国 | 1 | 1/26/2020 | 1/28/2020 |
| 大阪府 | 大阪府 | 1 | 1/20/2020 | 1/29/2020 |
ピボットにする
pvt = df.pivot_table(index="hsp", columns="Date", values="人数").fillna(0)
pvt = pvt.rename(index = dict(zip(jpn[0], jpn[2]))).rename(index={"その他":"others"})
pandasにはpivot_tableというメソッドが用意されており、文字通りピボットテーブルを作成することができます。(エクセルいらずですね。)また、NaNを0で埋めておきます。
そして、北海道→hokkaidoなどとリネームします。僕の環境では、インデックス名やカラム名に日本語が含まれていると、文字が表示されません。何かインストールすると解決されるらしいですが、リネームで対応します。(より良い方法があれば是非、教えてください。)
jpn[0]の中身は、北海道、青森、など漢字の都道府県名です。
jpn[2]の中身は、hokkaido、aomoriなどローマ字の都道府県名です。
これらをzip関数でペアにし、dict関数で辞書にし、rename関数に渡します。
また、「その他」を「others」に変更します。
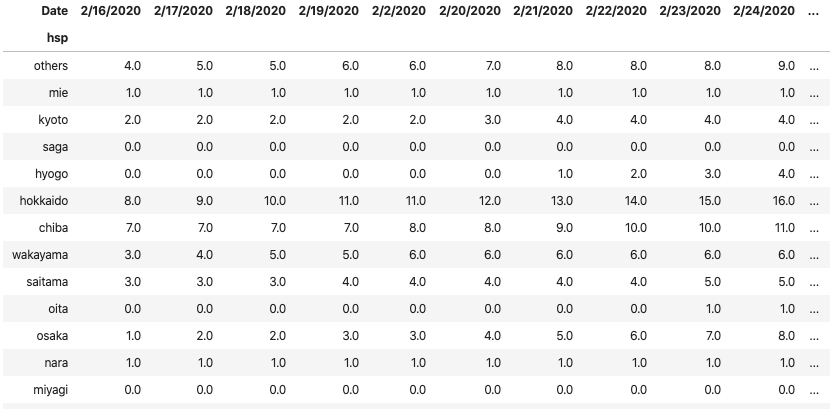
累積人数にする
for i in range(len(pvt)):
pvt.iloc[i]=pvt.iloc[i].cumsum()
pvtから1行ずつ取り出し、numpyのcumsum()関数で累積の人数にします。
表示して、保存する
plt.figure(figsize=(20,10))
sns.heatmap(pvt.iloc[:,-60:] , linewidths=0, cmap='Spectral', cbar=True, xticklabels=5)
plt.savefig("colona.png")
若い日付の日は、感染者が(幸い)ほとんどおらず表示しれても意味がないので60日前から表示することにしました。
:(スライス)を使うことができます。例えば10:20であれば10以上20未満を示します。
こんな感じで、ヒートマップを表示させることができました。