※記事を読んでくださるみなさまへ
・対象はプログラミングもバイオインフォ解析も全く未経験のバイオ研究者です
・筆者は基礎研究を初めて約1年3ヶ月の大学院生です
・いわゆる”DRY”な研究を始めたのは約5ヶ月前からです
・そのため記事の内容にはとんでもない間違いやずっと良い別の方法が存在するかもしれません
・また、筆者と同じレベルの人が0から始めるときの参考になるよう、説明は結構まだるっこいです
・ご指摘の度に記事をブラッシュアップしていきたいです、よろしくお願いします
過去記事一覧
①環境構築まで
②NGSデータのダウンロードとクオリティチェック
③NGSデータのトリミング
④Pipeline (hisat2, samtools, stringtie)
⑤番外編:コードのブラッシュアップ
⑥Rによる発現解析
前回の続きから:trimmomaticのインストール、実行:シングルエンドの場合
シーケンスデータのクオリティチェックが終わったら、次に低品質なリードやアダプター配列と思しき配列を除去、トリミングしなくてはいけません。このような処理ツールの中でもかなり高性能なのが、今回インストールするtrimmomaticです。
conda install trimmomatic
では早速トリミングをしてみましょう。まずは親サイトのクイックスタートどおりコピペ。最初に筆者の実験データ(シングルエンドリード)を使用してやってみました。ペアエンドの方はのちほど。
java -jar trimmomatic-0.35.jar SE -phred33 1_TAAGGCGA-G.fastq.gz(←自分のインプットファイル) 1_TAAGGCGA-G_trimmed.fastq.gz(←ここはアウトプットしたいファイル名) ILLUMINACLIP:TruSeq3-SE:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
すると
Error: Unable to access jarfile trimmomatic-0.35.jar
と怒られる。どこを直していけば良いだろうか。
trimmomatic.jarファイルの場所探し
前回の記事でjava問題は解決していたので、おそらく.jarファイルが見つからず、-jarの実行ができていないのが問題と予想。~/miniconda2/binの中を漁ってみる。
ls -Fl
すると
trimmomatic -> ../share/trimmomatic-0.36-3/trimmomatic*
なる記述を発見。今度は~/miniconda2/share/trimmomatic-0.36-3を同様に漁ると、
trimmomatic.jar
を発見!最初のコードのtrimmomatic-0.35.jar部分を
java -jar ~/miniconda2/share/trimmomatic-0.36-3/trimmomatic.jar SE -phred33 1_TAAGGCGA-G.fastq.gz(←自分のインプットファイル) 1_TAAGGCGA-G_trimmed.fastq.gz(←ここはアウトプットしたいファイル名) ILLUMINACLIP:TruSeq3-SE:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
と書き換えた。するとこんどはTruSeq3-SEなんてファイルはありません、と言われてしまった。どうやら対応するアダプター配列のリストが必要らしい。
アダプター配列のダウンロード
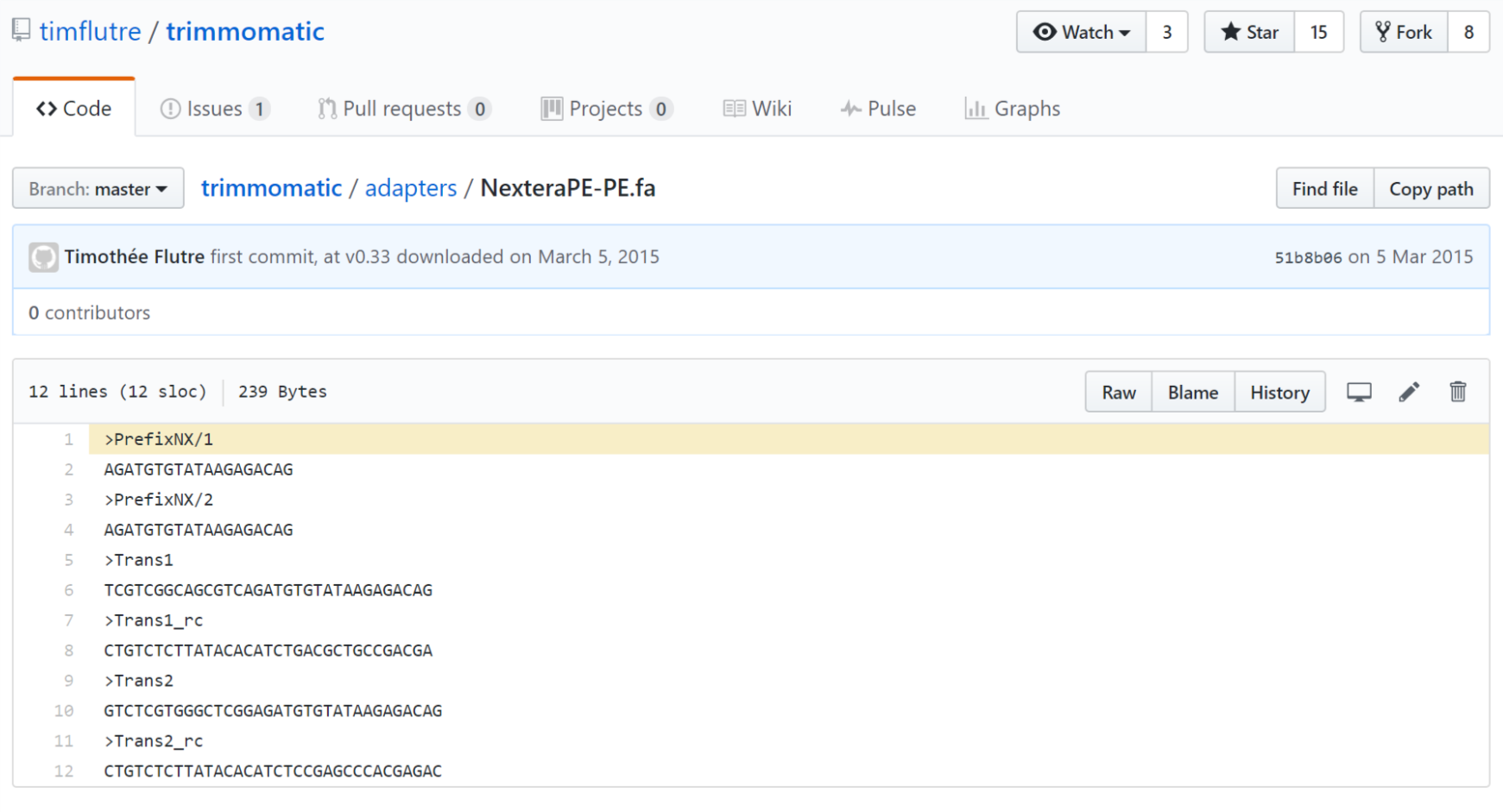
マニュアルを調べてみると、実験ごとに使ったPrep kitが重要で、それに応じたアダプター配列のファイルが必要とのこと。自分の使ったPrep kitがNextraだったので、マニュアルを参考に「NexteraPE-PE.fa」でGoogle検索。
すると、Githubのリンクに辿りついた。
こんな感じのページになるのでメモ帳にコピペ、各行の後ろでEnter押されていることを確認して(.faファイルではこれが大事らしい。特に最後の行を忘れがち)NexteraPE-PE.faと名付け作業ディレクトリに保存。
最終形
java -jar ~/miniconda2/share/trimmomatic-0.36-3/trimmomatic.jar SE 1_TAAGGCGA-G.fastq.gz 1_TAAGGCGA-G_trimmed.fastq.gz ILLUMINACLIP:NexteraPE-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
こうなった。これをコピペすると、特にエラーなく処理が開始される。
Input Reads: 1321477 Surviving: 1301306 (98.47%) Dropped: 20171 (1.53%)
こんな具合に最終レポートで、生き残ったリードの割合と、除去されたリードの割合が出力されます。
ペアエンドの場合
ペアエンドの場合は以下のようなコードになります。
java -jar ~/miniconda2/share/trimmomatic-0.36-3/trimmomatic.jar PE -phred33 ERR188044_chrX_1.fastq.gz ERR188044_chrX_2.fastq.gz ERR188044_chrX_1_paired.fq.gz ERR188044_chrX_1_unpaired.fq.gz ERR188044_chrX_2_paired.fq.gz ERR188044_chrX_2_unpaired.fq.gz ILLUMINACLIP:NexteraPE-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
オプションがSE→PEになり、インプットファイルがForwardとReverseに対応する2つ、アウトプットのファイルがpairedかunpairedにわけられて合計4つ完成します。以降の解析はpairedのファイルを使用してやっていきましょう。
こんな具合に、特にもとのシーケンスの品質が低いデータはトリミングを行ってからマッピングに進みましょう。
次回以降でようやくHISAT2を使ったマッピングの話に進みます。