こんにちは!バイオインフォマティクス系オタク修士学生のroadricefieldです! 今日はpheatmapのお話です.
pheatmapとは
Rのパッケージであるpheatmapはhclustを内包しており, お手軽に階層的クラスタリングを行うことができます.
早速試してみます.
library(pheatmap)
data <- data.frame(A = rnorm(100), B = rnorm(100), C = rnorm(100))
heatmap <- pheatmap(data, color = colorRampPalette(c("blue", "gray88", "red"))(100),
clustering_method = "ward.D2",
clustering_distance_rows = "correlation",
clustering_distance_cols = "correlation",
fontsize = 20, show_rownames = F, cutree_rows = 4)
clustering_methodでhclustがサポートするクラスタリング法を指定できます. clustering_distance_rows, clustering_distance_colsでクラスタリング距離を指定できます. "correlation"(相関係数)とdistがサポートするものが指定できます.
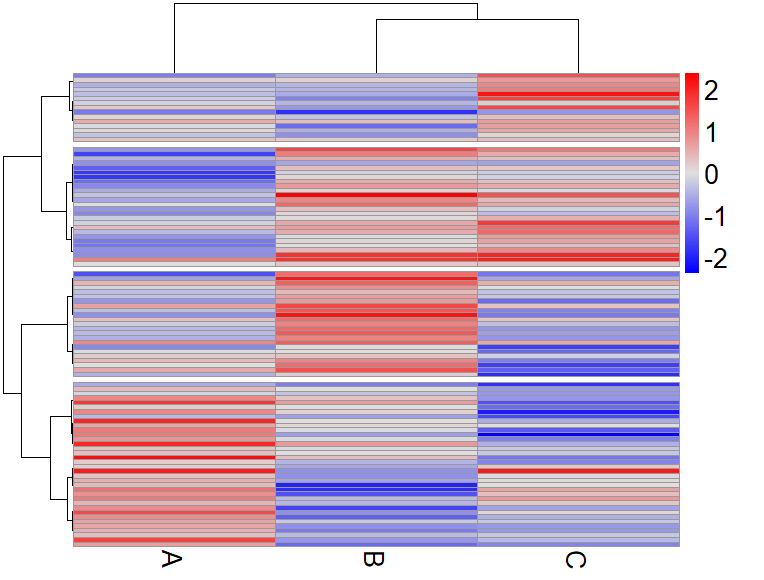
上記のコードを実行すると下のようにプロットされます.
cutree_rows = 4と指定したので4つのクラスターに分けられています.
クラスターを引っこ抜く
クラスタリングしたら各クラスターのデータを取り出したいですよね. pheatmapはクラスタリング結果のプロットはとても簡単な反面, その作業が少しわかりにくくなっています.
まず, クラスタリング後の並びに並び替わったrow namesがheatmap$tree_row$orderに格納されているのでこれを取り出しておきます.
after_clustering_order_IDs <- heatmap$tree_row$order
クラスタリングしたデータフレームのrow namesが普通に通し番号だった場合(上記コードのような場合)は以下のようなクラスタリング後の並びに並び替わったrow namesが得られます.
> after_clustering_order_IDs
[1] 100 44 83 36 52 74 15 89 18 71 34 3 8 25 45 2 58 23 84 24
[21] 61 85 53 96 31 67 93 22 60 88 38 37 87 55 77 99 27 46 76 26
[41] 64 33 90 13 50 14 86 12 41 49 78 6 43 42 39 73 5 91 56 47
[61] 92 48 35 57 29 63 79 17 19 54 1 40 4 95 11 98 32 59 7 81
[81] 70 82 51 69 10 28 21 62 80 66 75 72 16 20 94 97 65 68 9 30
上から100, 44, 83...番目のデータが並んでいるよということですね.
次にどの行がどのクラスターに入ったかを調べましょうcutreeという関数を使って以下のように調べられます.
cluster_attr <- cutree(heatmap$tree_row, k = 4)
> cluster_attr
[1] 1 2 3 1 4 4 1 3 1 1 1 4 4 4 3 1 1 3 1 1 1 2 2 2 3 2 2 1 1 1 2 1 4 3 4 3 2 2 4 1 4
[42] 4 4 3 3 2 4 4 4 4 1 3 2 1 2 4 4 2 1 2 2 1 1 2 1 1 2 1 1 1 3 1 4 3 1 2 2 4 1 1 1 1
[83] 3 2 2 4 2 2 3 4 4 4 2 1 1 2 1 1 2 3
これは並び替わる前の行番号について示されています. つまり元の1行目はクラスター1に, 元の2行目はクラスター2に, 元の3行目はクラスター3に, 元の4行目はクラスター1に, 元の5行目はクラスター4に放り込まれたよということです.
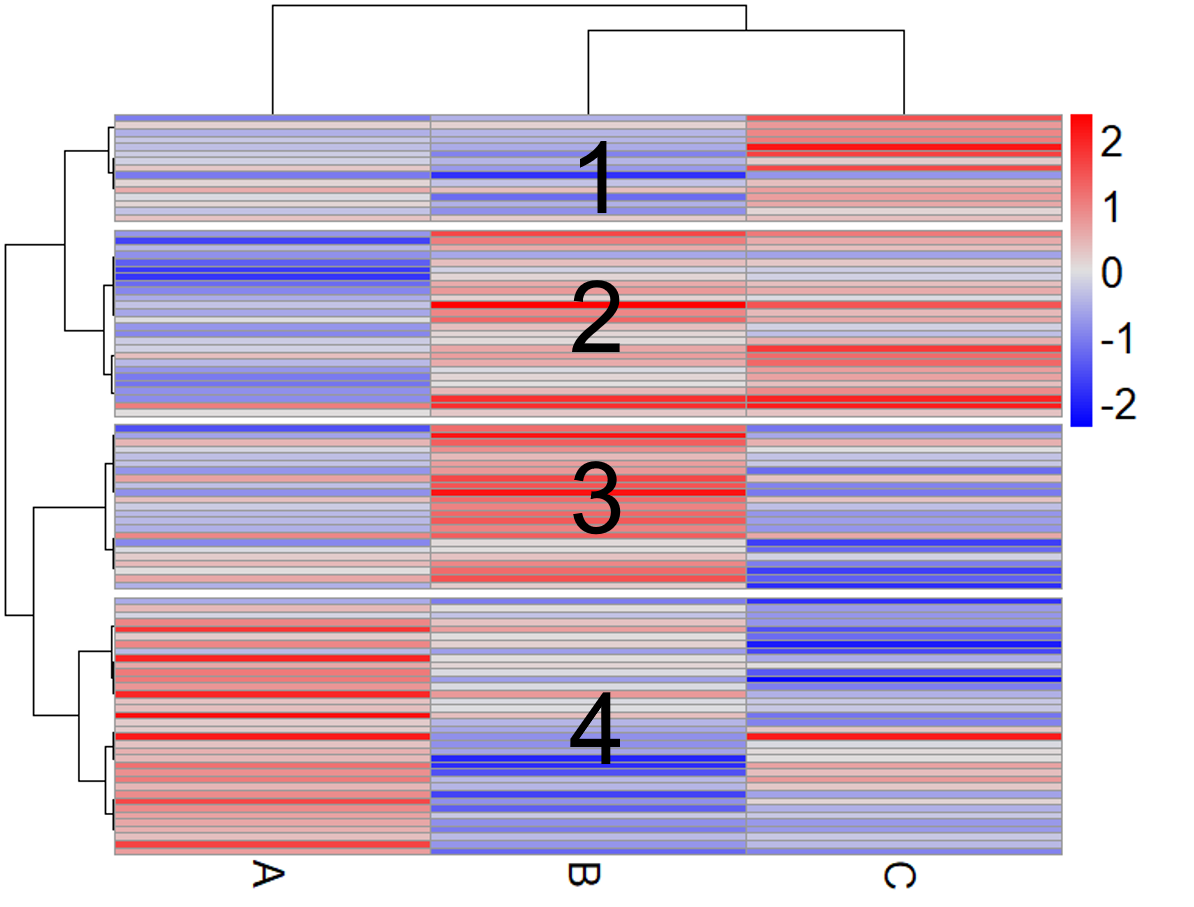
ここで注意です. 僕は最初この1, 2, 3, 4という数字が上のクラスターから順番に割り振られていると思っていたんですよ. こんなふうに
本当でしょうか?一応確認しておきましょうね.
check <- data.frame(clustered_order = after_clustering_order_IDs)
# 並び替える前のデータの順で各行がどのクラスターに放り込まれたかを示すベクトル, cluster_attr
# をクラスタリング後の順番に並び替えてcheckの列に加える.
check$sorted_cluster_attr <- cluster_attr[check$clustered_order]
さて, これでcheck$sorted_cluster_attrに上からクラスター何番なのかが入っています. ややこしいですが. 中を見てみましょう. (途中を省略しています.)
> check
clustered_order sorted_cluster_attr
1 100 3
2 44 3
3 83 3
4 36 3
5 52 3
16 2 2
17 58 2
18 23 2
19 84 2
20 24 2
42 33 4
43 90 4
44 13 4
45 50 4
46 14 4
65 29 1
66 63 1
67 79 1
68 17 1
69 19 1
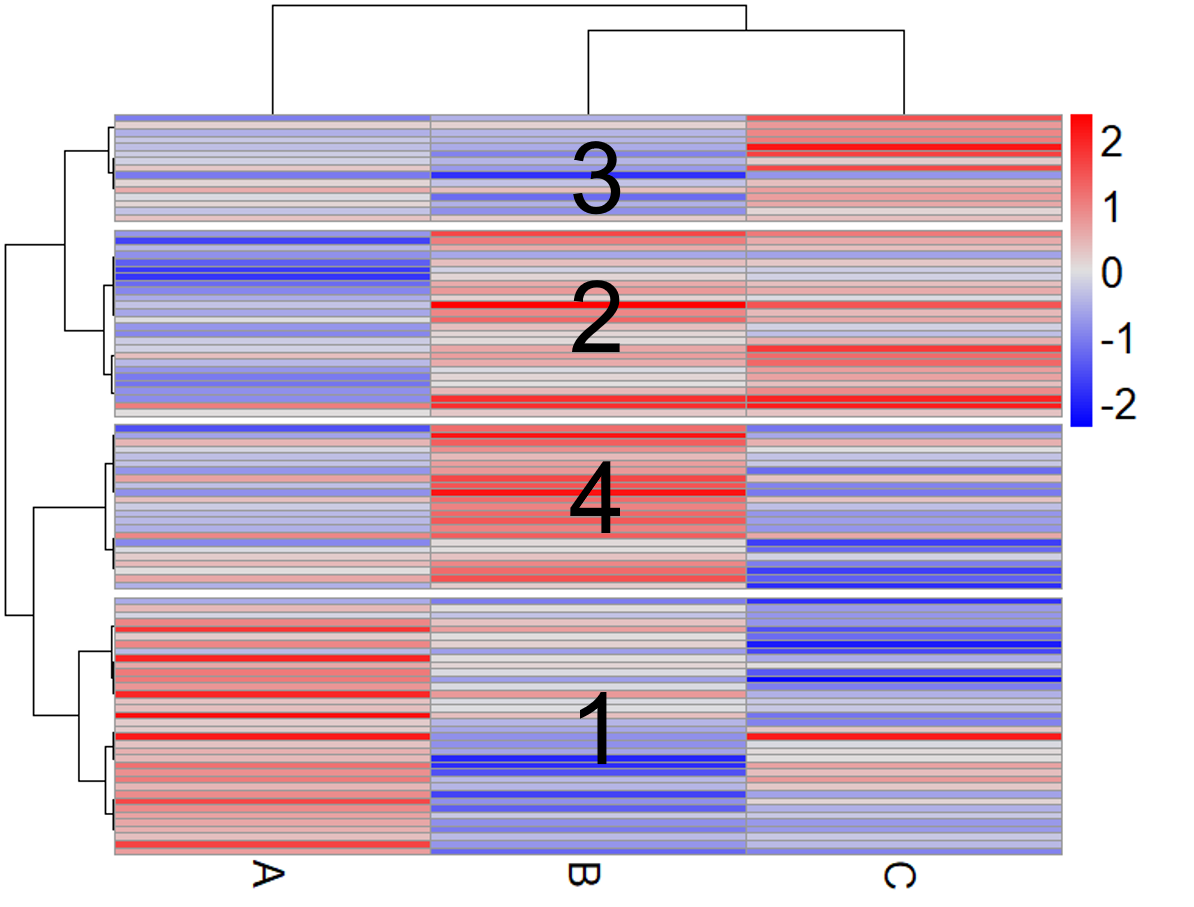
ちゃうやないかい!!!!なんでやねんややこしいなぁ!!! つまり今回の場合こうだったというわけです.
と, いうことは上から3つ目のクラスター(クラスター番号 4)だけ取り出してプロットしたいときは以下のようにすればよいですね.
# dataをクラスタリング後の順に並び替える.
clustered_data <- data[after_clustering_order_IDs,]
# 各行がどのクラスターに放り込まれたかを示す列追加する.
clustered_data$sorted_cluster_attr <- check$sorted_cluster_attr
# クラスター番号4の行を取り出す.
uekaramittsume <- subset(clustered_data, sorted_cluster_attr == 4)
# 4列目のsorted_cluster_attrはいらない.
uekaramittsume <- uekaramittsume[,1:3]
# 列の並びをクラスタリング後に揃えるのを忘れずに. 今回は元の並びから変わってない.
uekaramittsume <- uekaramittsume[,c(1,2,3)]
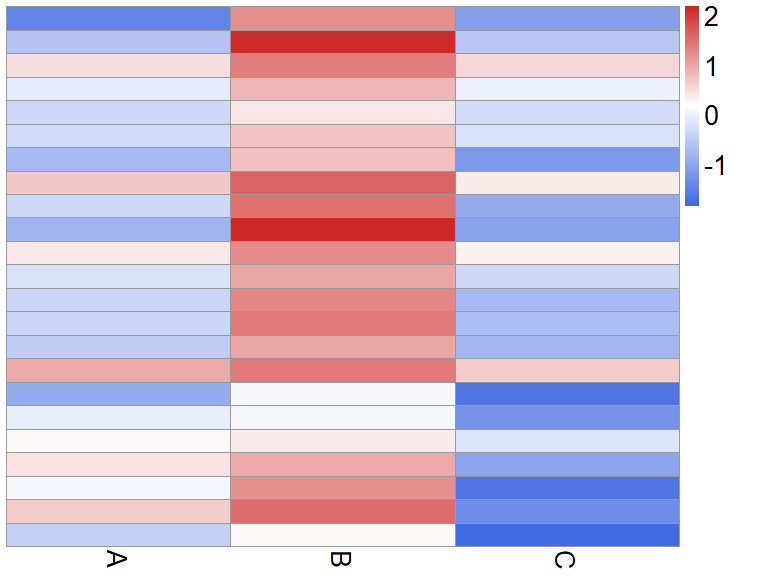
ext_heatmap <- pheatmap(uekaramittsume,cluster_cols = F, cluster_rows = F,
color = colorRampPalette(c("royalblue", "white", "firebrick3"))(100),
fontsize = 20, show_rownames = F)
cluster_cols = Fとcluster_rows = Fを指定するのを忘れないでくださいね. これを忘れると抜き出したデータで新たにクラスタリングを行ってしまうので並びがまた変わってしまいます.
あ!すみません. gray88をwhiteに変えてしまいました. 気にしないでください!
さいごに
今回は手軽に階層的クラスタリングを行うツール, pheatmapを紹介しました. プロットするには超便利ですがクラスターの取り出しが超ややこしいです. 僕は間違いに気づかずに研究を進めてしまうところでした. あぶねぇ~~......
今回のコードを一つにまとめて貼っておきますね.
library(pheatmap)
data <- data.frame(A = rnorm(100), B = rnorm(100), C = rnorm(100))
heatmap <- pheatmap(data, color = colorRampPalette(c("blue", "gray88", "red"))(100),
clustering_method = "ward.D2",
clustering_distance_rows = "correlation",
clustering_distance_cols = "correlation",
fontsize = 20, show_rownames = F, cutree_rows = 4)
after_clustering_order_IDs <- heatmap$tree_row$order
cluster_attr <- cutree(heatmap$tree_row, k = 4)
check <- data.frame(clustered_order = after_clustering_order_IDs)
# 並び替える前のデータの順で各行がどのクラスターに放り込まれたかを示すベクトル, cluster_attr
# をクラスタリング後の順番に並び替えてcheckの列に加える.
check$sorted_cluster_attr <- cluster_attr[check$clustered_order]
# dataをクラスタリング後の順に並び替える.
clustered_data <- data[after_clustering_order_IDs,]
# 各行がどのクラスターに放り込まれたかを示す列追加する.
clustered_data$sorted_cluster_attr <- check$sorted_cluster_attr
# クラスター番号4の行を取り出す.
uekaramittsume <- subset(clustered_data, sorted_cluster_attr == 4)
# 4列目のsorted_cluster_attrはいらない.
uekaramittsume <- uekaramittsume[,1:3]
# 列の並びをクラスタリング後に揃えるのを忘れずに. 今回は元の並びから変わってない.
uekaramittsume <- uekaramittsume[,c(1,2,3)]
ext_heatmap <- pheatmap(uekaramittsume,cluster_cols = F, cluster_rows = F,
color = colorRampPalette(c("royalblue", "white", "firebrick3"))(100),
fontsize = 20, show_rownames = F)