久しぶりの投稿です。私は普段、実験系の研究室(物理系)に所属して日々研究をして過ごしているのですが、私がこれまで使ってきた実験データの解析手法を公開したら誰かの役に立つのではないかとふと思い、記事を書くことにしました。これからPythonを使って実験データの解析をしたいと思っている人の助けになれば幸いです。
この記事に書くこと
タイトルにもあるように、私は実験をしてデータの解析をするときにPython (Jupyter Notebook or Lab.)を使って解析をし、Pythonを使ってグラフを書いて、そのグラフを使ってプレゼンをし、論文を書いています。Pythonがなければ私の研究は全く進まないと言ってもいいでしょう(大げさ)。
実験データの処理には大きく分けて2つの段階があります。解析とグラフ化です。この記事ではこの2つについて私が使っている小技(基礎)をいくつか紹介します。
実験データの解析
データファイルを開く
様々な装置を使って実験をしていると、当然、様々な形式のデータが得られます。本質的には同じテーブルデータでも古い装置では拡張子が.txtだったり、新しめの装置だと.csvだったりします。CSVファイルなら、開く方法はよく知られているようにPandasを使えば普通は一発です。
import pandas as pd
data = pd.read_csv("データファイル名.csv")
しかし、世の中のものにはたいてい例外というものがあります。CSVなのにタブ区切りになっていたり(それCSVじゃなくてTSVだよねっていうやつ)、実験日時やパラメータが長々とヘッダとして入っていたり、色々カオスです。これらの多くはPandasを使って対処することができます。
# タブ(\t)でデータが区切られている時
data = pd.read_csv("データファイル名.csv", sep="\t")
# データの前にヘッダが 10行入っている時
data = pd.read_csv("データファイル名.csv", skiprows=10)
時にはデータファイルにカラム名が書いておらず、数字が羅列されているだけという場合もあります。下のような場合です。
1 1
2 4
3 9
4 16
...
たぶん x = [1, 2, 3, 4,...], y = [1, 4, 9, 16, ...] のつもりなのでしょう。2行くらいならNumpyを使って読み込んでしまってもいいでしょう。
import numpy as np
x, y = np.loadtxt("データファイル.txt").T
Pandasで読み込もうとすると、普通にread_csv()をすると一番最初の行がカラム名として生贄にされてしまいます。これを防ぐためにはひと工夫する必要があります。
data = pd.read_csv(
"データファイル名.txt",
sep=" ",
header=None,
names=["x", "y"]
)
header=Noneを指定することで、このファイルにカラム名の情報がないことを教えて、こちらからnamesでカラム名を指定してやります。
データをこねくり回す
データを分割する
実験装置が有能になればなるほど機械がやってくれる部分が多くなり、結果として様々な条件のデータが1つのファイルにまとまっているということが多くなります。例えば一つのデータファイルに様々な温度での測定データがまとめて入っている状況が良い例でしょう。これは便利ですが、解析の際にはきちんと分割してやらなければなりません。
data = pd.read_csv("データファイル名.csv")
data_300k = data[abs(data.temp - 300) < 0.1] # 300 Kのデータだけ取り出す
上に特定の温度のデータのみを取り出す例を示しましたが、ここで重要なのは、取り出すデータの条件として ==を使わなかった点です。あえて、300 Kからのずれが 0.1 K未満という指定の仕方をしたのは、装置の吐き出すデータには必ずある程度の揺らぎのようなものがあり、300 Kのデータでも記されている温度がぴったり300 Kであることはまずないからです。
ずれの大きさはこの例では 0.1 K 未満としましたが、これは行っている実験によって調整すべき量です。もしも 0.1 K刻みで温度を変えているのならこの条件は緩すぎて使い物になりません。
補間する
解析するときに、あるデータから別のデータを引くということがあると思います。代表的なのは測定データからバックグラウンドを引くというようなときですね。両者のデータの横軸がぴったり合っていれば普通に引き算をすればよいのですが、ずれているときにはまず両方のデータで横軸をそろえる必要があります。
補間の方法はいろいろありますが、線形補間で間に合う場合が多いと思います。
from scipy.interpolate import interp1d
# (x1, y1), (x2, y2) という2組のデータがあり、横軸を x2に合わせたい
f = interp1d(x1, y1)
y = f(x2) # 横軸を x2 に合わせた時の y1
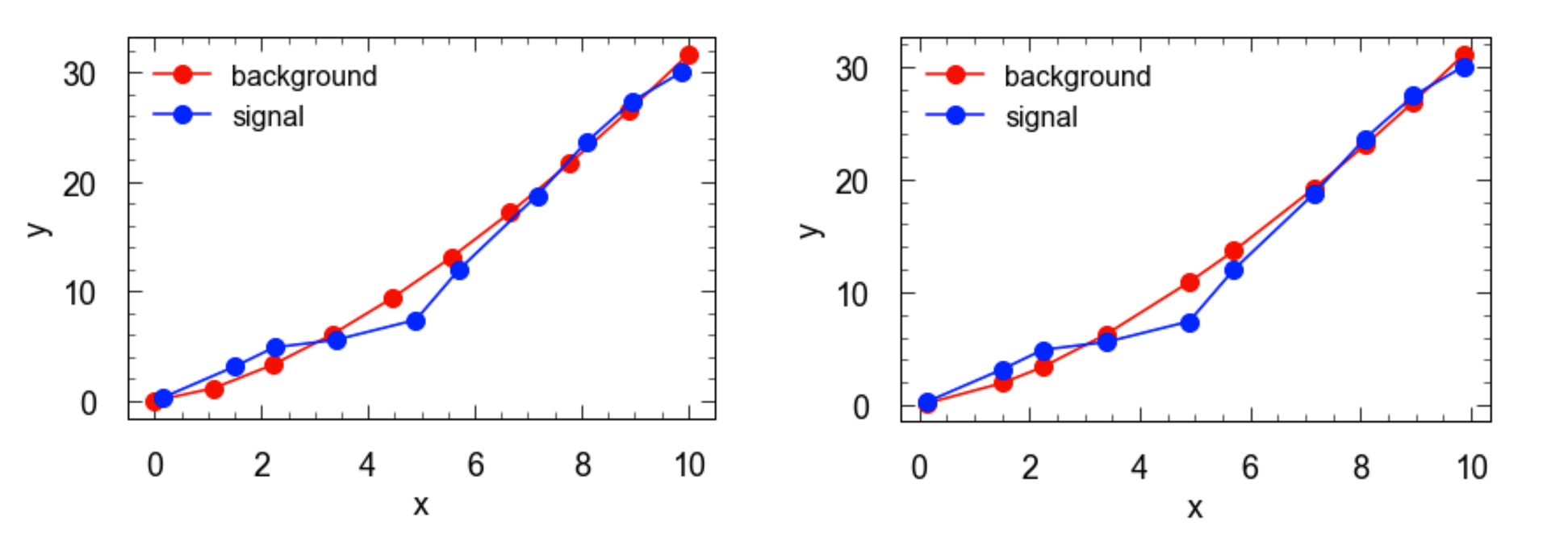
下に例を載せます。左側の図が測定したデータ(signal) とバックグラウンド(background)の生データだとします。これをみると、両者ではx軸の値がずれていてそのまま引くことはできませんが、補間によってx軸がそろえられていることがわかります(右図)。
データの可視化
データを解析して、その次に可視化するという時の小技です。私はグラフの見た目を最低限そろえるために Jupyter の最初のセルで以下のようなコードを実行しています。
import matplotlib.pyplot as plt
plt.rcParams["font.family"] = "Arial"
plt.rcParams["xtick.labelsize"] = 18
plt.rcParams["ytick.labelsize"] = 18
plt.rcParams["xtick.direction"] = "in"
plt.rcParams["ytick.direction"] = "in"
plt.rcParams["xtick.major.size"] = 12
plt.rcParams["ytick.major.size"] = 12
plt.rcParams["xtick.major.pad"] = 18
plt.rcParams["ytick.major.pad"] = 18
plt.rcParams['axes.linewidth'] = 1.0
def mint(ax):
ax.tick_params(which='major', top=True, right=True, length=8)
ax.tick_params(which='minor', top=True, right=True, length=4)
ax.minorticks_on()
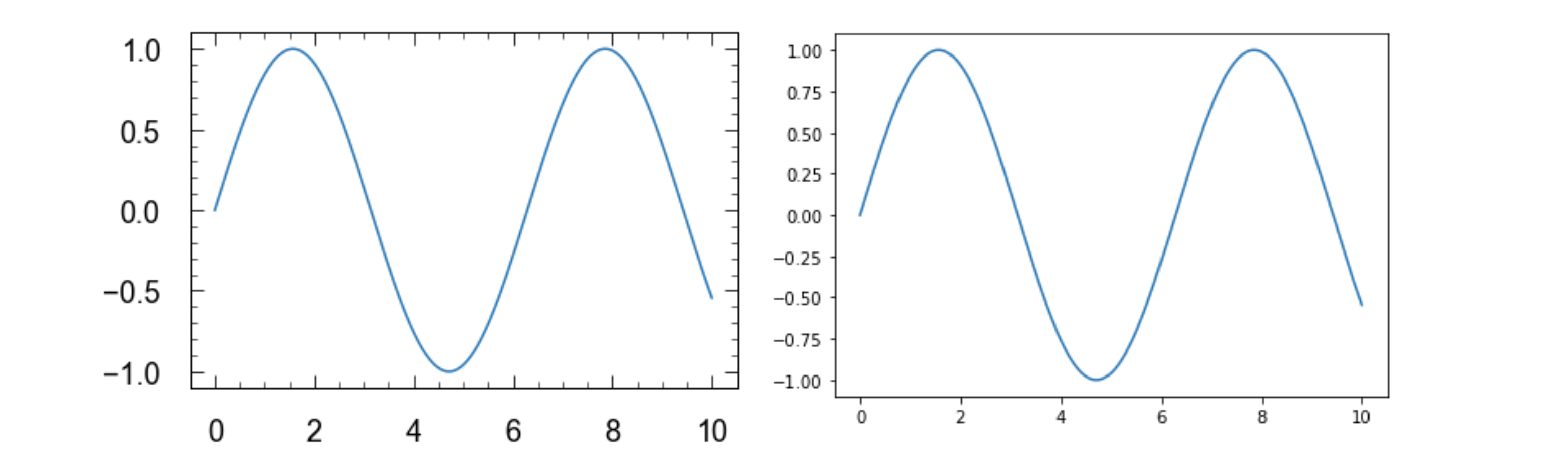
これを最初にしておくと特にただプロットしただけでもそこそこましなグラフができます。
x = np.linspace(0, 10, 500)
y = np.sin(x)
plt.plot(x, y)
mint(plt)
plt.show()
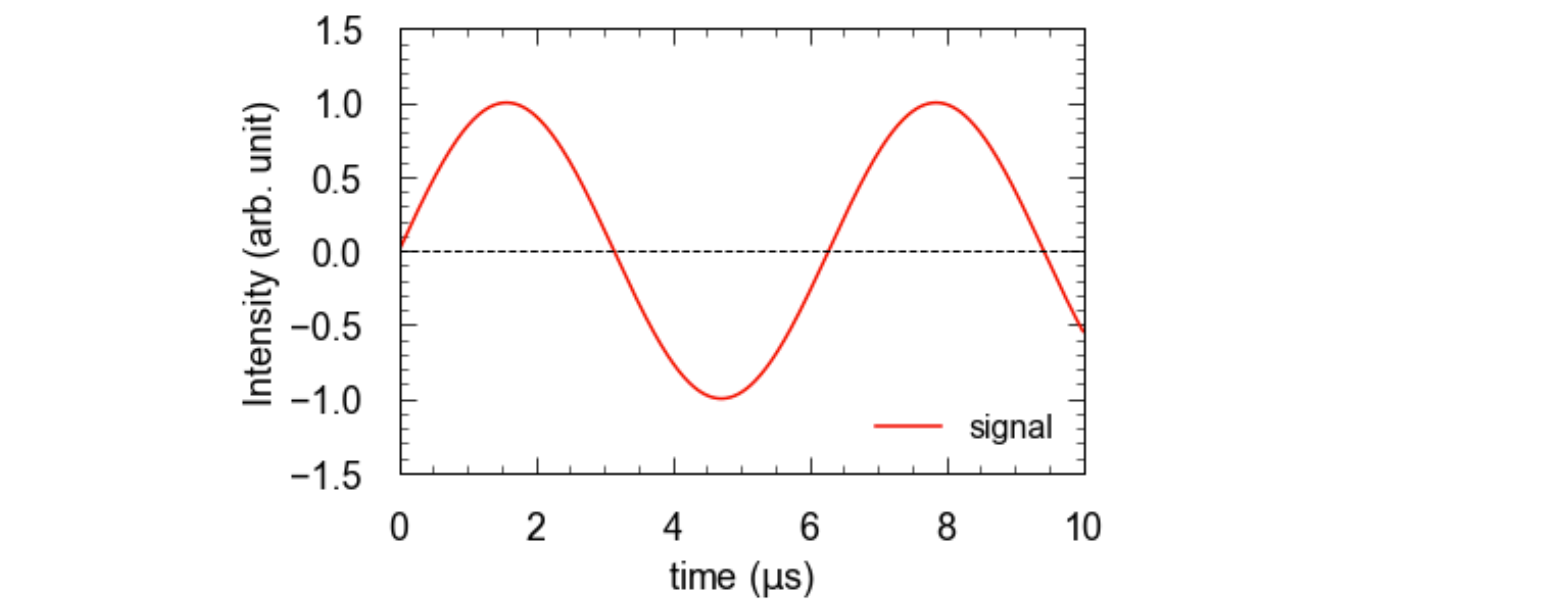
左が上のコードを実行したときの図で、右が何もしていないときです。これに軸ラベルや凡例を付けることで下図まで持っていけます。横軸のラベルを見るとわかるように、LaTeX表記も使えます。
x = np.linspace(0, 10, 500)
y = np.sin(x)
plt.plot(x, y, "r", label="signal")
plt.axhline(0, ls="dashed", lw=1, c="k")
mint(plt)
plt.xlabel(r"time ($\mathrm{\mu}$s)", fontsize=18) # LaTeX 表記も使える
plt.ylabel("Intensity (arb. unit)", fontsize=18)
plt.xlim(0, 10)
plt.ylim(-1.5, 1.5)
plt.legend(frameon=False, fontsize=16, loc="lower right")
plt.show()
このくらいまですればプレゼンには使えます。スクショをとって使ってもいいですが、私は SVG画像として保存して使っています。
plt.savefig("figure.svg", transparent=True)
これを plt.show()の直前に入れとけば保存されます。SVGだと transparent=Trueを指定することで背景が透明になるのでいろいろ使いやすいです。
まとめ
ここまでいろいろ書いてきましたが、紹介できることは思ったほど多くなかったです(泣)(他にもいくつかありましたが私の分野に特化しすぎていて紹介するほどではなかった。。。)他にもなにか思いついたら追加しようと思います。