前回、Apache SparkのMLlibを使って、RandomForestをローカル環境で試してみました。今回はそれをjarにして、AWS EMRのクラスタ上でバッチとして動かしてみます。
当方開発環境がWindowsでして、AWS Cliは入れておりません。ということで、出来るだけWebのAWS Management Consoleからやっていく方針で。
Steps
RandomForestを呼び出すコードをScalaで作る
前回と異なるのは、以下3点:
- SparkContextでMasterを設定しない => spark-submitでの実行時に投げるので、ここではいらない。
- 入力ファイルの場所をs3n://として、S3のロケーションを指定する。
- printlnでの出力は止めて、SparkContext.saveAsTextFileでS3に結果を出力する => 引き続きリダイレクトで標準出力を取ろうと思ってたら、標準出力がYarnのログとして残る形になってクラスタ外から確認するのが面倒だったので。
HelloRfOnCluster.scalaという名前で適当なフォルダにおいてください。そのファイル置いた場所を[your project folder]とします。
import org.apache.spark._
import org.apache.spark.SparkContext
import org.apache.spark.SparkContext._
import org.apache.spark.mllib.tree.RandomForest
import org.apache.spark.mllib.util.MLUtils
import java.util.Date
import scala.collection.mutable.ListBuffer
object HelloRfOnCluster
{
val dateTimeKey = "%tF-%<tT" format new Date
val outputLocation = "s3n://[my bucket]/out/" + dateTimeKey
val conf = new SparkConf().setAppName("HelloRfOnCluster")
val sc = new SparkContext(conf)
// Load and parse the data file.
// libsvm style iris Data - http://www.csie.ntu.edu.tw/~cjlin/libsvmtools/datasets/multiclass/iris.scale
val data = MLUtils.loadLibSVMFile(sc, "s3n://[my bucket]/iris.scale").cache()
// Split the data into training and test sets (30% held out for testing)
val splits = data.randomSplit(Array(0.7, 0.3))
val (trainingData, testData) = (splits(0), splits(1))
val buf = new ListBuffer[String]
def main(args: Array[String]): Unit =
{
buf.append("Start")
trainClassifier()
buf.append("End")
val summaryRDD =sc.makeRDD[String](buf,1)
summaryRDD.saveAsTextFile(outputLocation + "/summary")
}
def trainClassifier() =
{
val startTime = System.currentTimeMillis
// Train a RandomForest model.
// Empty categoricalFeaturesInfo indicates all features are continuous.
val numClasses = 4 // Iris data: 3 labels, (label + 1) value seems to be needed.
val categoricalFeaturesInfo = Map[Int, Int]()
val numTrees = 3 // Use more in practice.
val featureSubsetStrategy = "auto" // Let the algorithm choose.
val impurity = "gini"
val maxDepth = 4 // <= 30
val maxBins = 32
val model = RandomForest.trainClassifier(
trainingData, numClasses, categoricalFeaturesInfo,
numTrees, featureSubsetStrategy, impurity, maxDepth, maxBins)
// Evaluate model on test instances and compute test error
val labelAndPredsRDD = testData.zipWithIndex.map
{
case(current, index) =>
val predictionResult = model.predict(current.features)
(index, current.label, predictionResult, current.label == predictionResult) // Tuple4
}
val execTime = System.currentTimeMillis - startTime
val testDataCount = testData.count()
val testErrCount = labelAndPredsRDD.filter(r => !r._4).count // r._4 = 4th element of tuple (current.label == predictionResult)
val testSuccessRate = 100 - (testErrCount.toDouble / testDataCount * 100)
buf.append("RfClassifier Results: " + testSuccessRate + "% numTrees: " + numTrees + " maxDepth: " + maxDepth + " execTime(msec): " + execTime)
buf.append("Test Data Count = " + testDataCount)
buf.append("Test Error Count = " + testErrCount)
buf.append("Test Success Rate (%) = " + testSuccessRate)
buf.append("Learned classification forest model:\n" + model.toDebugString)
labelAndPredsRDD.map(x => x.toString()).saveAsTextFile(outputLocation + "/details")
}
}
JARを作る (sbt assembly)
ローカルマシンでjarを作って、それをEMRクラスタ上で動かすという、バッチとしての実行を目指していきます。
sbtの設定
まずはsbtの設定。[your project folder]に、build.sbtというファイルを作って、以下の内容を保存。
ポイントはsparkのdependencyをprovidedにすること。Mavenのprovidedと同じで、Sparkの依存部分をjarには含まず、実行時にはEMRのクラスターの方のsparkを使う形にします。
name := "Hello-Rf"
version := "0.0.1"
scalaVersion := "2.10.4"
scalacOptions ++= Seq("-Xlint", "-deprecation", "-unchecked", "-feature", "-Xelide-below", "ALL")
libraryDependencies ++= Seq(
"org.apache.spark" %% "spark-core" % "1.2.1" % "provided",
"org.apache.spark" %% "spark-mllib" % "1.2.1" % "provided"
)
次に、[your project folder]の下に[project]というフォルダを作ります。んで、そこにassembly.sbtというファイルを作りまして、以下の内容を保存。
addSbtPlugin("com.eed3si9n" % "sbt-assembly" % "0.13.0")
sbt assemblyの詳細については、以下を参考にしてください。
sbt/sbt-assembly - GitHub
sbt assemblyの実行
コマントプロンプトにて、以下を実行。
> cd [your project folder]
> sbt assembly
[target]フォルダがビルド中に追加されて、その下の[scala-2.10]フォルダにHello-Rf-assembly-0.0.1.jarが出来上がっているかと思います。
Sparkにjarをsubmitするためのシェルスクリプトを作る
EMRの「Step」としてsparkにjarを投げるとき、s3://elasticmapreduce/libs/script-runner/script-runner.jarを使うのですが、こいつに喰わせるシェルスクリプトを用意しておきます。
spark-submit.shという名前で適当な場所に以下の内容を保存。シェルスクリプトなんで、改行コードは「LF」で。
# !/bin/bash
echo "Start"
date
rm ./Hello-Rf-assembly-0.0.1.jar
echo "Download jar to master-node..."
hadoop fs -get "s3://[my bucket]/Hello-Rf-assembly-0.0.1.jar" "./Hello-Rf-assembly-0.0.1.jar"
date
echo "Spark-Submit"
/home/hadoop/spark/bin/spark-submit --deploy-mode cluster --master yarn-cluster --class HelloRfOnCluster ./Hello-Rf-assembly-0.0.1.jar
if [ $? -ne 0 ]; then
echo "Error";
date
exit 1
else
echo "Done"
date
exit 0
fi
ちなみに、awslabsのExample「Spark ApplicationをEMR StepとしてSubmitする」では、script-runner.jarに直でコマンドを喰わせてます。そのやり方ならこのシェルスクリプトは要らないのですが、引数の扱いが面倒でメンテしずらい感じがしてるので、まあ作っときましょう。
S3にResourceを置く
S3に適当なbucketを作って、ファイルを配置します。私は以下のようにしてます。
[my bucket]
| Hello-Rf-assembly-0.0.1.jar
| spark-submit.sh
| iris.scale
|
└─out
└─log
iris.scaleの詳細については、前回をご参照ください。
AWS Manegement ConsoleからEMRを起動して、Sparkを実行する
引き続き、ブラウザで「AWS Manegement Console」からEMRのクラスタを作って、実際にsparkを実行してみます。
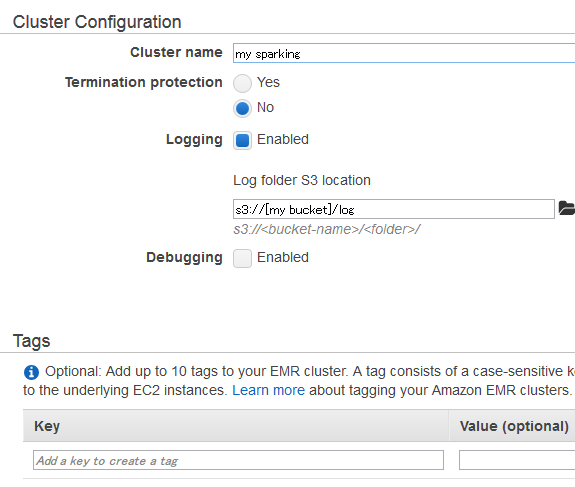
1. Cluster Configuration & Tag
- Cluster name: 適当
- Termination protection: No
- Logging: Enabledにして、s3://[my bucket]/logを指定
- Debugging: チェック外してDisable
- Tag: 空白
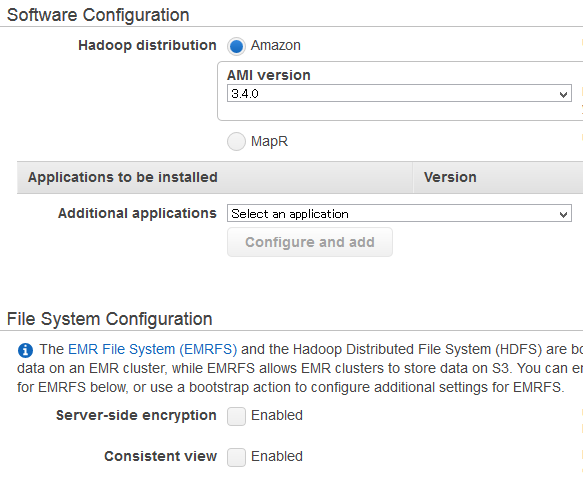
2, Software / File System Configuration
- AMIの指定: デフォルト (3.4.0)
- Applications to be installed: 「Hive」「pig」「Hue」が指定されてると思いますが、今回は使わないので、「×」ボタンを押して全部削除してます。
- File System Configuration: 特に指定しない
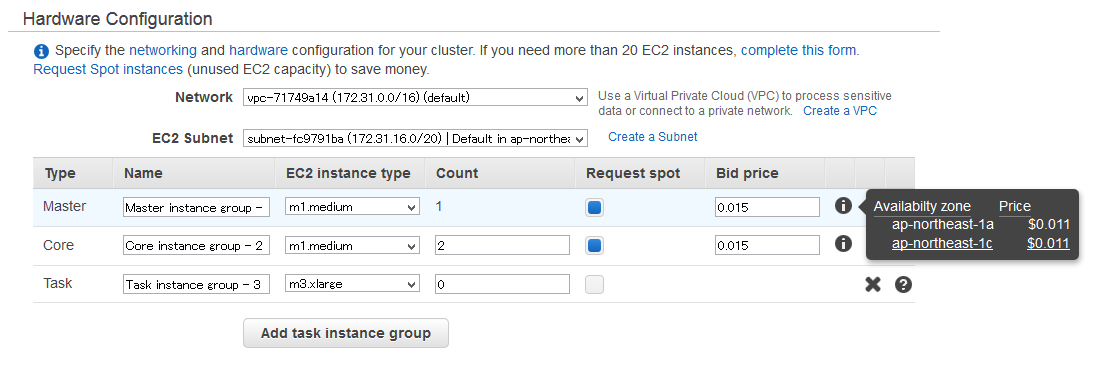
3. Hardware Configuration
ここは「スポットインスタンス」を使って安く上げていきましょう(スポットインスタンスって何、って方は是非グーグル先生に聞いてみて下さい。とても面白い仕組みです)。
「Request spot」にチェックを入れて、「i」アイコンにカーソルを充てると、現在の相場が出ます。
- Network: デフォルト
- EC2 Subnet: これは「Availability Zone」の選択になります。スクリーンショットでは差が無いのでアレですが、spotの値段に差があれば[1c]を選ぶのも手。
- Master: m1.medium, Request Spot = True, Bid price = 0.015
- Core: m1.medium * 2, Request Spot = True, Bid price = 0.015
Bid Priceは、適宜空気を読んだ値付けで。
m1.mediumの相場が0.010だったとして、どうせすぐTerminateするってことなら0.012 or 0.013などとギリギリを狙うもよし。長めのjobを安定運用したいなら0.02にしてみるとか。この辺は状況&気分しだいですね。
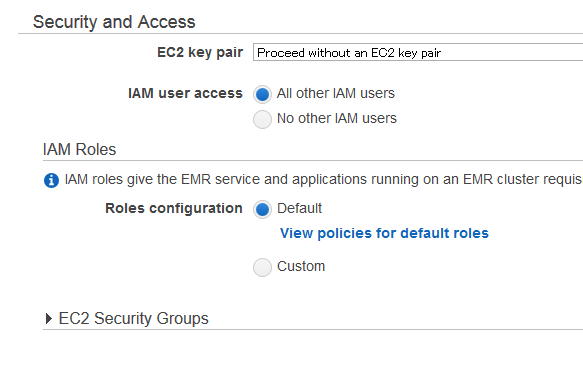
4. Security and Access
EC2 key pairのところだけは、EC2でいつも使っているkey pairを選んでおいてください。Master-nodeへSSHでつなぐときに必要です。
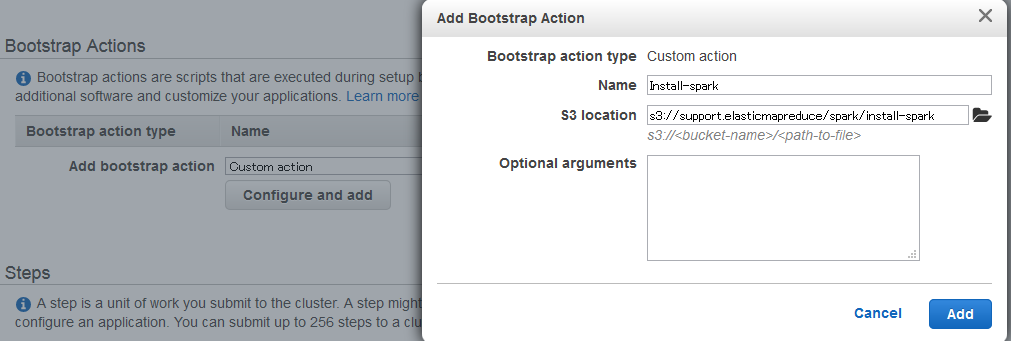
5. Bootstrap Action
EMRクラスタ新規作成時、Sparkがインストールされるようにするための設定をします。
- Add bootstrap actionでプルダウンから「Custom action」を選ぶ
- 「Configure and add」ボタンを押すと設定ダイアログが出る
- Nameを適当に変更(しなくても良いですけど)
- S3 locationに
s3://support.elasticmapreduce/spark/install-sparkを指定 - 「Add」ボタンを押して戻る
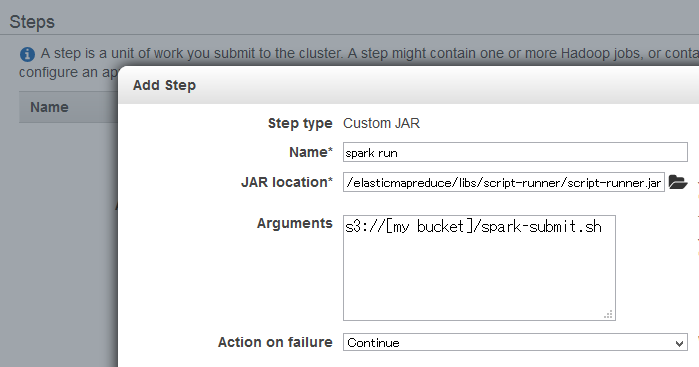
6. Steps
EMRに何かしら一仕事させることを「Step」と呼びますが、それを追加する設定。ここでは、先ほど作ったspark-submit.shを、s3://elasticmapreduce/libs/script-runner/script-runner.jar経由で呼び出して、sparkに仕事させます。
- 「Add Step」のプルダウンで、「Custom JAR」を選択
- 「Configure and add」ボタンを押すと設定ダイアログが出る
- Nameを適当に変更(しなくても良いですけど)
- JAR locationに
s3://elasticmapreduce/libs/script-runner/script-runner.jarを指定 - Argumentsに
s3://[my bucket]/spark-submit.shを指定 - 「Add」ボタンを押して戻る
Auto-terminateはNOのままで。
Create Cluster -> 実行待ち
これで設定は完了なので、「Create Cluster」ボタンを押してしばらく待ちます。
ここ数日試した感じだと、Starting 10分、Bootstrapに数分、Step実行にまた数分ってところですね。
結果
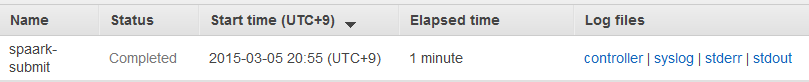
Management Console上
上手く行ってれば、Cluster Detailで、Stepsの項にこんな感じで状況が出てくるはず。尚、ログが見れるようになるまでには少し時間がかかります。
(Nameがtypoってるのスクリーンショット取るときに気づいた。。。)
Failしてたら、logを見つつ、SSHでMaster-nodeに入って探りを入れていく方向になります。
SSHでつなぐときは、User名: hadoop、Masterのホスト名はManegement ConsoleのCluster Detailで探してください。
Sparkが実行中にfailした場合、logに「Tracking URL: http://[ip addr]:9046/proxy/application_xxx」というようなのが出てくる時があります。ポートを開けるの面倒なので、インストールされてるはずの「lynx(テキストブラウザ)」でみると良いかもです。
追記: debug等等については別枠で書いたので、こちらもどうぞ
「Amazon EMR クラスターでホストされているウェブサイトの表示」(とSparkのデバッグ)
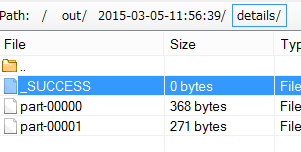
S3に出力されたもの
RandomForestの結果は、S3の[my bucket]/outに出てくるはずです。
こちらはDetailの出力。分割して出てきてるのを見るに、分散されて実行された感はありますね。
Terminate
クラスタの終了はCluster Detailから「Terminate」ボタンを押すだけ。EC2のインスタンスも連動して消えてくれます。
Logging: enabledにしてる場合、S3に保存されたログは自動では消えないので、要らなければ消してください。
その他Tips
SubmitしたJob(Step)を止める
投げてみたら思いのほかjobが重くて、途中で止めてEC2のinstanceを追加したくなる、というようなことがあるかと思います。
残念ながらWeb ConsoleにはStepを止める設定は見当たらず、SSHでmaster-nodeに入って止める形になります。今回の設定ではyarnを使ってsparkを動かしているので、yarnのjobを確認 -> killの流れで止めます。
master-nodeにSSHで入って、
> yarn application -list
とすると、実行中のjobの状況が取れるので、そこからApplication IDを拾って、
> yarn application -kill [your runnning application id]
とすれば、止められます。
EMRのManagement ConsoleにTerminateしたクラスタの情報が残りっぱなし
実験でEMRクラスタを起動 -> Terminateしたのち、そのクラスタの情報はしばらくManagement Consoleのクラスタリストに残り続けます。2週間単位で残るとか何とか言ってますね以下。
How to clean up the list of Terminated AWS EMR clusters?
S3にログが残ってたらそれは削除するとして、後は気にスンナってことみたいですね。
ただ、一つ罠がありまして。
クラスタ設定時に「Debugging: Enabled」にした場合、SimpleDBにデータが自動で作られて、それはEMRクラスタ終了させても消えません。これが残っているとクラスタリストに終了させたクラスタが1ヶ月以上残り続けるということになるようなので、別途手動で消す必要があります。
SimpleDB、Management Consoleからは扱えないっぽいので、AWS Cliで削除するなりしないといけません。私は、EclipseのAWS Toolkit経由で消しました。
Next Step
Kindle版の「Learning Spark: Lightning-fast Big Data Analysis」を眺めてたのですが、「ML Pipelines」を使うと、パラメータのチューニングが出来そうなんですよね。
ML Pipelines: A New High-Level API for MLlib - databricks
気が向いたら試しましょー(といいつつ、Adhocな分析ならもしかしてMicrosoft Azure MLの方がやりやすい?などと目移り中でもあります。。。