最近、PythonやRではどんどん便利なパッケージが開発されている。例えば、Pandasなんかではテーブルデータを整形をするためのメソッドだけでなく、可視化のためのメソッドまで用意されている。なので、Pandasを使えばデータの整形から可視化まで1つのスクリプトで行える!。。。。お願いだからやめてくれ。いや、書き捨てのスクリプトだったり、Jupyter notebook上でそういうことするのは分かる。でもお願いだから再利用する可能性のあるスクリプトで、そういうことするのはやめてくれ。便利だからってこういうスタイルを続けていると後で大変なことになる。そして、自分と他人を大変苦しめる羽目になるのである。なぜ、駄目なのか。ここでは一つ例をあげて説明したい。
Seabornのclustermapとか使うのやめようよ。
個人的に、データの整形から可視化までをやってくれる最もありがた迷惑な関数として、seabornのclustermapがある(Rのheatmapやheatmap.2も同様)。
これはその名の通りclusteringされたheatmapを作成してくれる関数である。複数サンプルの遺伝子発現データなんかを派手に見せるためにデンドログラム付きで、カラーラベル付きのデデーンとしたheatmapを作成してくれる。今や、生命科学界隈の論文では当たり前といっていいほど一般的な図である(参考図)。
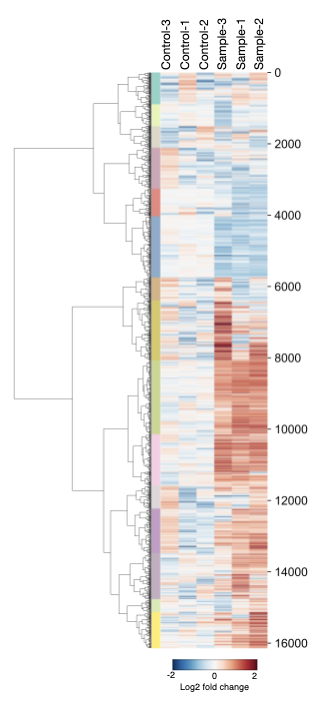
Seabornのclustermap等を使えばインフォマティクスに明るくない人もたった一行のコードでなんかクラスタリングされた図を生成できる。うん素晴らしいね。しかし、最近ある人にこんな感じの質問をされた。
質問者 「Pythonを使って、clustering heatmapを作ったんですがちょっと分からないことがあっていいですか?」
筆者 「うん?いいよー。」
質問者 「heatmapの行や列の順序が分からないんですが、どうすればわかりますか?」
最初、私は意味がわからなかった。なのでこう聞き返した。
筆者 「え、クラスタリングされた後のテーブルをcsvとかの形式で書き出せばいいんじゃない?」
質問者 「??クラスタリングされた後のテーブル?は手元にありません。」
筆者 「え、じゃあどうやってheatmap作ったのさ?。クラスタリングした後のテーブルを入力にしたんじゃないの?」
質問者 「???、ちょっと何言ってるか分からないですが、入力は持っているテーブルデータです。」
何を言ってるのか分からないのはこっちである。目の前の人間は、クラスタリング後のテーブルデータを持っていないのにクラスタリングされたheatmapのPDFファイルはあるという謎のことを言っているのである。このことについて「え、そういうもんじゃないの?」と思う人はマジで反省してほしい。話をつづけると、どうやらseabornのclustermapを使うと、クラスタリングから可視化までを全てやってくれるということらしかった。そして、heatmapの図しか出力されないから、クラスタリング後のテーブルデータなんて無いということらしい。
私は、もう何がなんだかわからなかった。そして心のなかで呟く。
「クラスタリングされた後のテーブルデータを得られなくて、heatmapだけ出力されて何が嬉しいの??。まさか、なんか綺麗な図を作ることを目的にクラスタリングしたってこと?。クラスタリングは別にかっこいい図をつくるための前処理じゃないぞ?。」
しかし、こういう言い方は最近では許されていない、そこでこう聞き返す。
筆者 「そういう関数を使って作ったのはわかった。でもさ、じゃあヒートマップの色だけ変えたい時はどうしてるの?」
質問者 「???質問の意図がわからないですが、cmapのパラメータを変えます」
筆者 「それ、データが大きいと毎回時間かからない?」
質問者 「!!そうなんですよ!。今回もこの図をつくるのに10分ぐらいかかるんですよ!」
その返答を聞いて、私は心がおれかけた。そして、こんな調子でデータ解析をしている生命科学の研究者が多くいるであるという事実に気づき絶望した。。。。。
なぜ、データの加工と可視化のスクリプトは分けるべきなのか。
ここまでの話で、すでにデータの加工と可視化のスクリプトを分けた方がいい理由に気づいた人も多いだろう。スクリプトを分けたほうが理由は大きく2つある。
- 加工過程で作成されるデータを他人も編集可能な形式で残すため。
- 毎回データを加工するという処理を繰り返さないため。
1については、さっきの例でいうとクラスタリング後のテーブルデータを残しておこうということだ。これが残ってないと結局どの要素同士がクラスタリングされたのかよく分からないし(まさかラベルも画像に出力して確認するわけにもいかないし、まじでやめてくれ。)、詳細な数値情報も分からない。他人にデータを渡す時共有するときだって不便だ。
研究や受注解析とかしてると頻繁に解析結果を他者に共有することがある。人によっては綺麗な図だけ渡せば喜んでくれる人もいるが、多くの場合、データを貰った相手もデータの信頼性を確かめるために色々と聞いてくるし、図の見た目や、ちょっとした追加解析は自分の手でしたいという人もいる。なので、基本的に他人にデータを渡す時は生データだけでなく、エクセルでも論文の図と同じ図がつくれるようにまで加工したデータも共有するべきだ。今回の例であれば、クラスタリング後のテーブルデータをラベル付きで共有してあげれば、受け取り手はエクセルでもエクセルのヒートマップ機能を使って、seabornで出力するheatmapと同じ図を作ろうと思えば作れるし、フィルタリングの機能を使えば特定の要素だけでheatmapを作り直すのだっておちゃのこさいさいだ。一方で、画像データだけもらってもできることはない。「うん、綺麗な図だねー。」程度の感想しかでてこないだろう。だって画像データだけもらっても追加の解析等はできないのだから、、、。まぁベクター図ならな。数値データ取り出すこともできるけどな。うん。
2については、入力データのサイズが大きくなるほど問題になる。基本的にはクラスタリングはその手法にもよるが計算オーダーが大きい。なのでデータサイズが数倍になるだけで計算時間は何十倍にもなっていく。最近の生命科学では普通に、1000x1000とか10000x10000とかのサイズのクラスタリングを扱ったりするわけだが、世の中にはヒートマップの色調をかえるためだけにクラスタリングからやり直している人がたくさんいるというのがどうやら現実らしい。1. で書いたようにクラスタリング後のテーブル残しておけば色調だけ変えるなんて一瞬なんだぜ。
うん、まぁな。最近のCPU速いもんな。でもさ、1CPUの速度は結構頭打ちしてるんだぜ。「俺のPCはラップトップだけど8コア16スレッドおおおおおお。だから計算負荷とか関係ないからぁああ!」みたいに思ってるかもしれないけど、clustermapとかみたいなメソッドは並列化できるように実装されてないからさ。Pythonは基本シングルスレッドでしか走らないしさ。うん。毎回クラスタリングするのマジ時間の無駄だからさ。やめようぜ。
ということで、結局質問者には上記の話を伝え、クラスタリングをするための関数を伝授し、クラスタリングされた後のデータをheatmapとして可視化するための方法を伝授したのであった。そこらへんの話は、次の記事で。しかし、あんまり重要性が伝わってなかった気がするんだよなぁ。。。。泣