1.はじめに
ひょんなことからRNA-seqを修行しております。
・RNA-seqとは
細胞の中のmRNAやmiRNAの配列を解読して、発現量の定量、新規転写配列の発見ができる手法です。
参考URL:RNA-Seqとは?
2.最初にちょっと勉強(参考URL)
きっと初心者に優しい参考URLをご紹介
・遺伝子発現量解析 RNA-Seq
非常にわかりやすく、コンパクトにまとめられていると感じます。
・次世代シーケンサー(NGS)を用いた解析と関連データベース・ツール
動画(統合TV)です。次世代シーケンサーについて基礎を学べます。
・ (Rで)マイクロアレイデータ解析
Rのスクリプトが細かく書かれております。
3.ゲノムのインデックスファイルの作成方法
1.簡単なやつ
マッピングする際に、
対象のデータがヒトであればヒト(ホモ・サピエンス)のゲノムデータを用います。
で、どこにあんの???
→Homo sapiens や Mouse などメジャーどころは簡単です。
illuminaのigenomeよりダウンロード出来ます。
参考URL:各種既知遺伝子アノテーション用GTFファイル
2.ちょいめんどいやつ
人生簡単なことばかりではありません。
マイナーなデータにぶち当たることもあります。
では、今回はフェレットのゲノムインデックスとアノテーションファイルを作成します。
・フェレットとは
ご存知、愛くるしいあいつ
(これは個人的見解)
・・・・・・・・・・・・・・・・・・・・・・・・
フェレットは、イタチ科に属する肉食性の哺乳小動物である。
狩猟、実験、毛皮採取、愛玩用に用いられる。
参考URL:wiki
2−1.まず、リファレンスゲノムデータのダウンロード
公共データよりダウンロードします。
ensemble, NCBI, UCSC の3つ団体がありますが、今回はensembleよりダウンロードします。
→NCBIは後日記事upします。
→UCSCも作成し、動作確認をしましたが失敗に終わりました。(フェレットの遺伝子情報が少ない模様)
ensembleのURL:http://asia.ensembl.org/info/data/ftp/index.html
をクリック
いろんな種類のデータがあります。
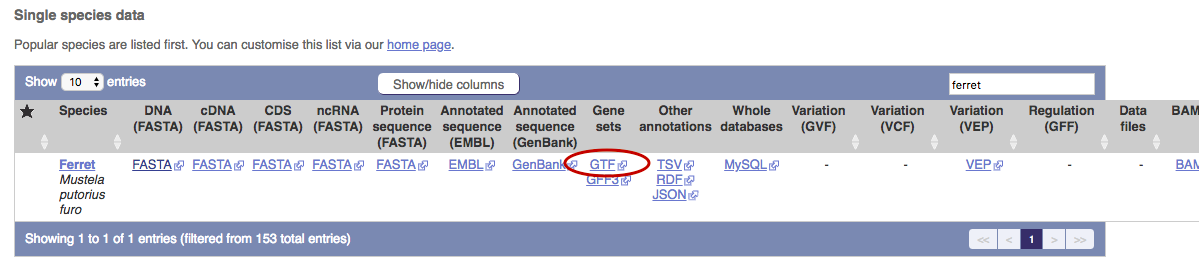
そこで、「Filter」にferret(学名=Mustela putorius furoでもOK)を入力し検索!
ゲノムインデックスの元(リファレンスゲノム)→ "DNA(FASTA)" の [FASTA] をクリック(※一番左の)

xxxxxx.dna.toplevel.fa.gz をダウンロード。
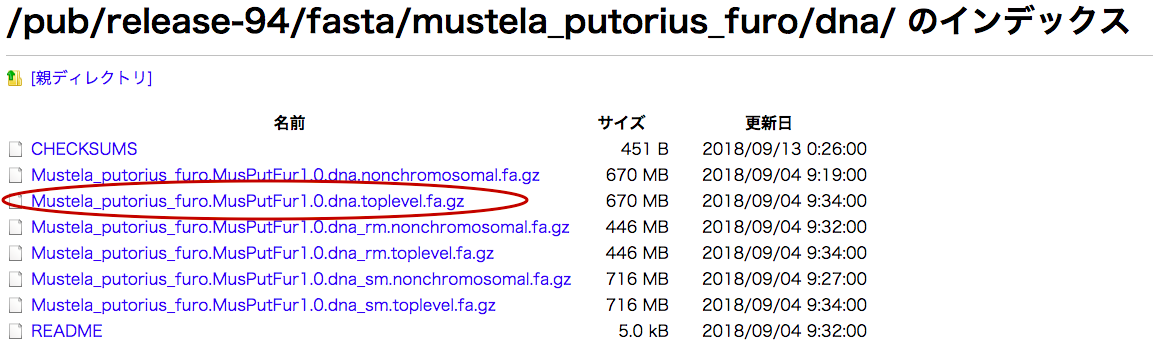
ターミナルで下記コマンドにてダウンロードしたファイルを解凍します。
→ xxxxxx.dna.toplevel.fa というファイルが出来ます。
$ gzip -d Mustela_putorius_furo.MusPutFur1.0.dna.toplevel.fa.gz
2−2.インデックス作成・・・の前にBowtie2をインストール
Bowtie2ってなんぞや。
・Bowtie2とは
次世代シークエンス で得られるリードを、リファレンスに対してマッピングするためのソフト。
参考:bowtie2: Mac へのインストール、使い方、オプションなど
・・・・・・・・・・・・・・・・・・・・・・・・
リファレンスファイルからインデックスを作成するときにも使用します。
Bowtie2のインストール方法は下記コマンドにて(Mac)
※Homebrew がインストールされている場合。インストール方法は下記サイトにて
参考:bowtie2: Mac へのインストール、使い方、オプションなど
$ brew install bowtie2
2−3.いよいよインデックス作成
インストールしたBowtie2を利用して下記コマンドでインデックスを作成しましょう。
・bowtie2-build・・・リファレンスファイルからインデックスを作成
・-f・・・リファレンスファイル指定オプション
※リファレンスファイル xxxxxx.dna.toplevel.fa の後ろに任意のインデックス名を指定
$ bowtie2-build -f Mustela_putorius_furo.MusPutFur1.0.dna.toplevel.fa MUSPF1
→ xxx.1.bt・xxx.2.bt・xxx.3.bt・ xxx.4.bt・xxx.rev.1.bt・xxx.rev.2.btの6つのインデックスファイルが完成!!
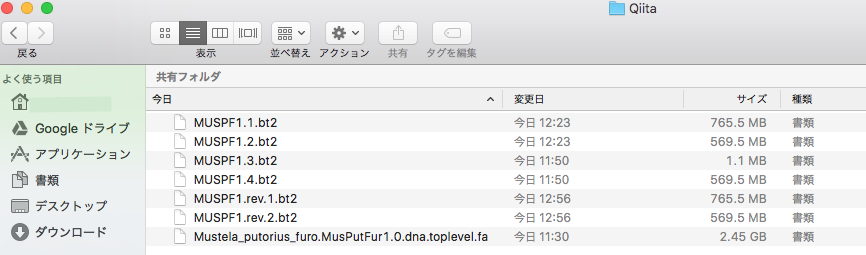
真っ白・・・なので、ファイルのアクセス権限を与えてあげましょう。
参考:Macターミナルコマンド「chmod」の使い方
参考:Linuxコマンド道場 ワイルドカード
$ chmod +x MUSPF1.1.bt2 MUSPF1.2.bt2 ・・・(略)・・・ MUSPF1.rev.2.bt2
または
$ chmod +x *.bt2 # * で一括処理できます。
2-4.ついでに、アノテーションファイルをダウンロード
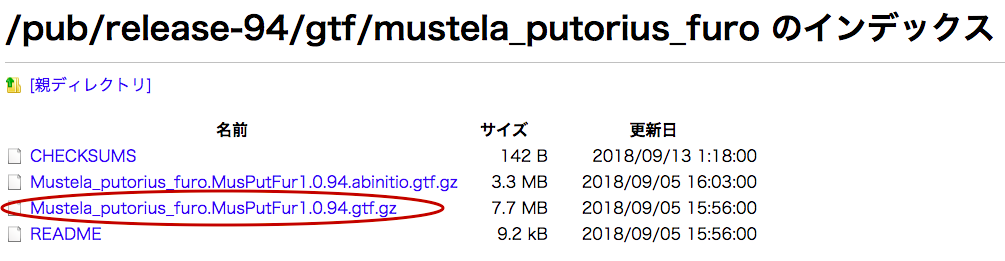
アノテーションファイル→"Gene sets" の [GTF] をクリック。
解凍して、、、
→ xxxxxx.gtf というファイルが出来ます。
$ $ gzip -d Mustela_putorius_furo.MusPutFur1.0.94.gtf.gz
アクセス権限を与えて完成!
$ chmod +x Mustela_putorius_furo.MusPutFur1.0.94.gtf
4.参考URL
・RNA-Seq マッピング TopHat(ショウジョウバエ paired-end)
こちらを参考に作成しました!
・Bowtie2
公式HPです。オプションの詳細がわかります。
以上です!
(更新:10190207)アクセス権限の箇所に、ワイルドカードを記入。便利ですね〜