公共Galaxy サーバーを利用してみよう
GalaxyにログインしなくてもGalaxyの機能を利用できる
公共のGalaxyサーバーは複数存在するけれど、ここでは https://usegalaxy.org/
Galaxyサーバーを利用する事にする
ペイン(機能枠)について
左側のToolsペインには各種のツールが、右側のHistoryペインにはこれまでの解析やデータ等の履歴が表示される
上部のメニューには新規ユーザー登録やログインするための"Login or Register"等が表示されているが、気にせずにまずはこのまま利用してみよう
塩基データをfasta形式でNCBIから取得する
ToolsペインのGet Data をクリックしすると、Get Dataカテゴリーに含まれるツールの一覧が表示される。更に Upload File from your computerをクリックする
以下、同様の手順を次の様にして示すことにする
Get Data => Upload File from your computer
Upload File from your computerを利用してデータを登録する
適当なFasta配列をGalaxyに登録するために、Entrezのfetch api を利用してみる
GenbankのIDを指定してFasta配列を取得してみよう
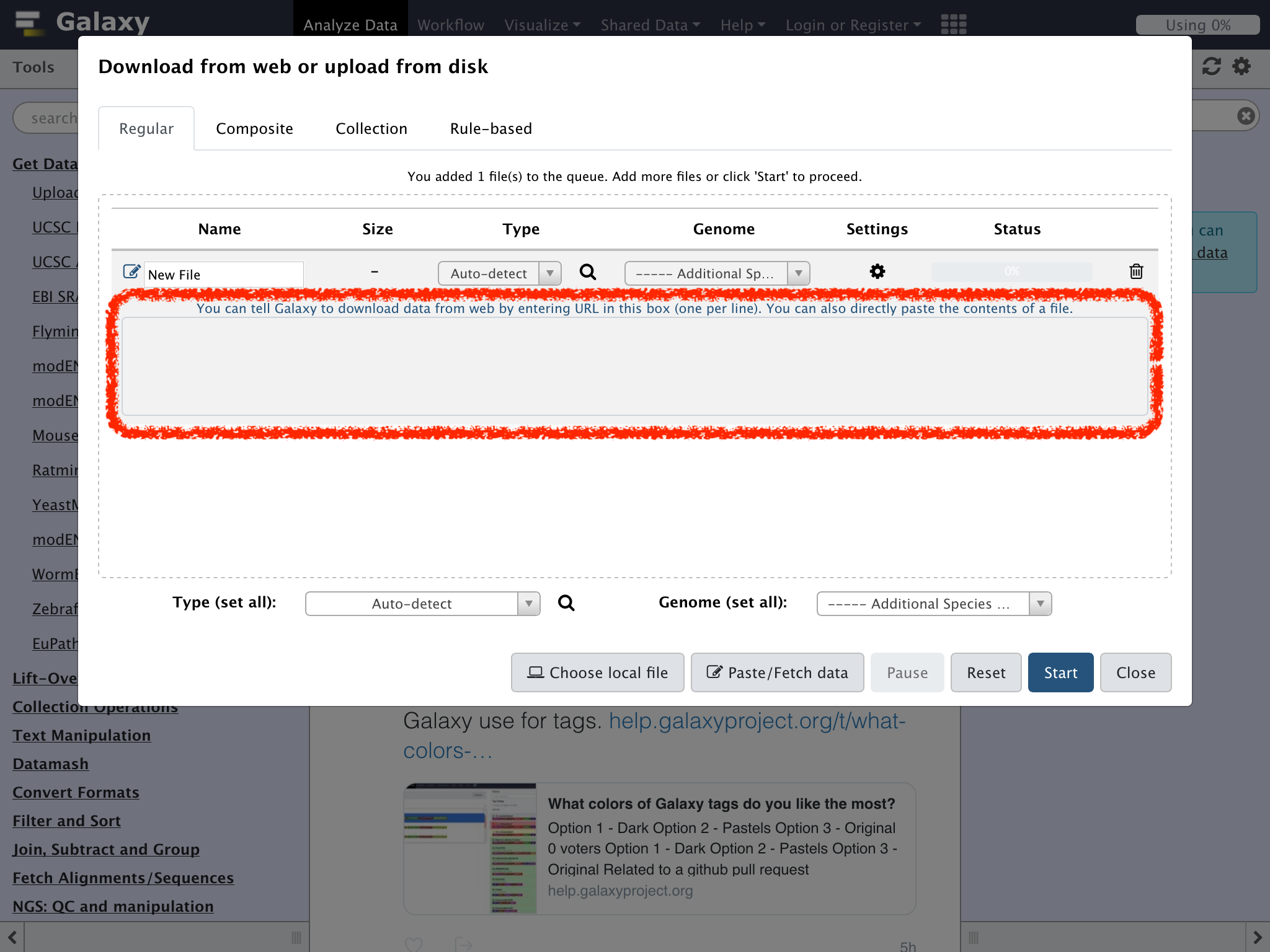
Upload Fileをクリックして開いたウィンドウの画面右下部の"Paste/Fetch data"ボタンをクリック
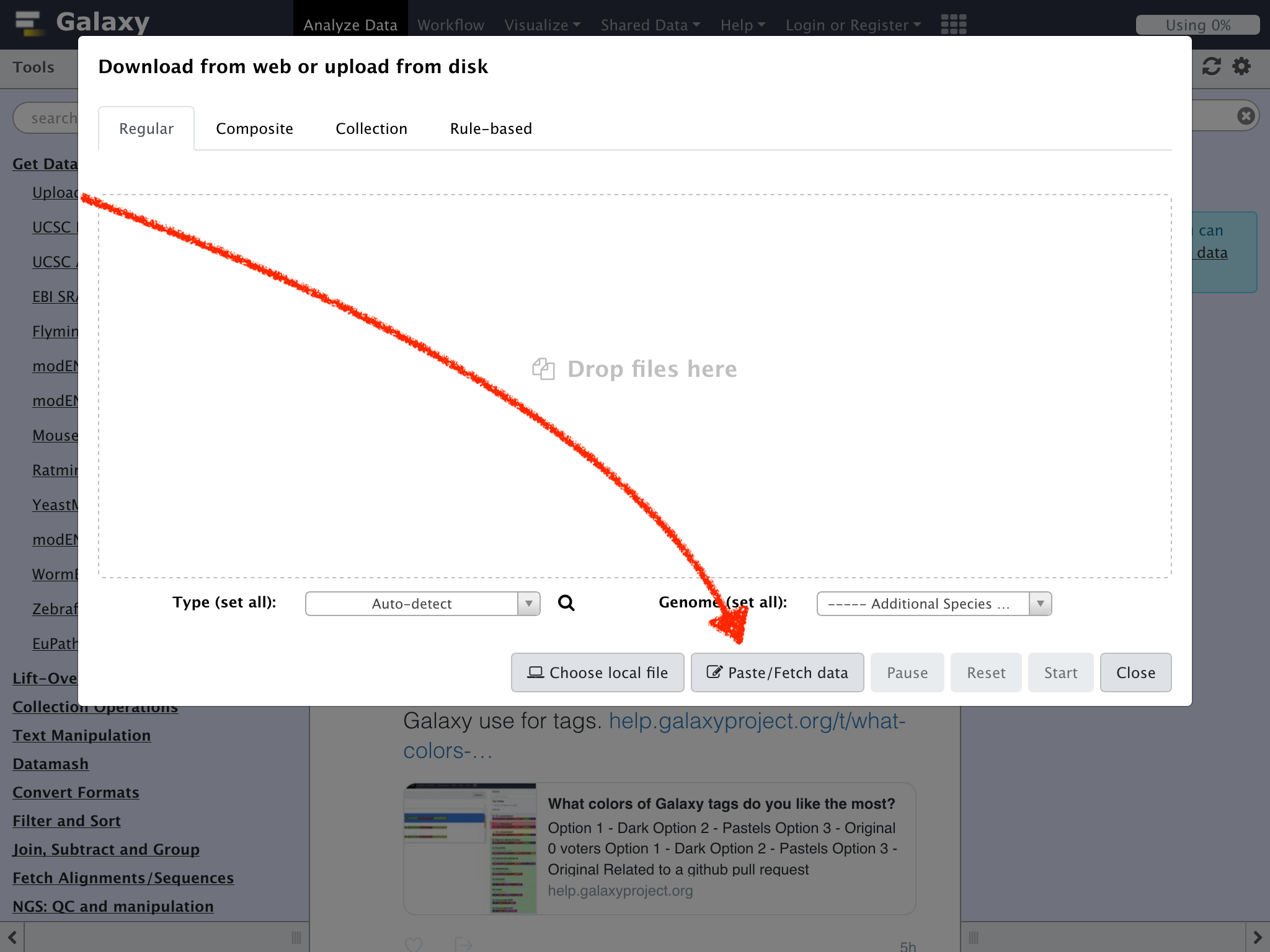
上の画面上の領域に次の内容をコピーアンドペーストする
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=nuccore&id=M24811&rettype=fasta&retmode=text
Typeのドロップダウンリストを"fasta"に変更し、ウィンドウの右下の"Start"をクリック
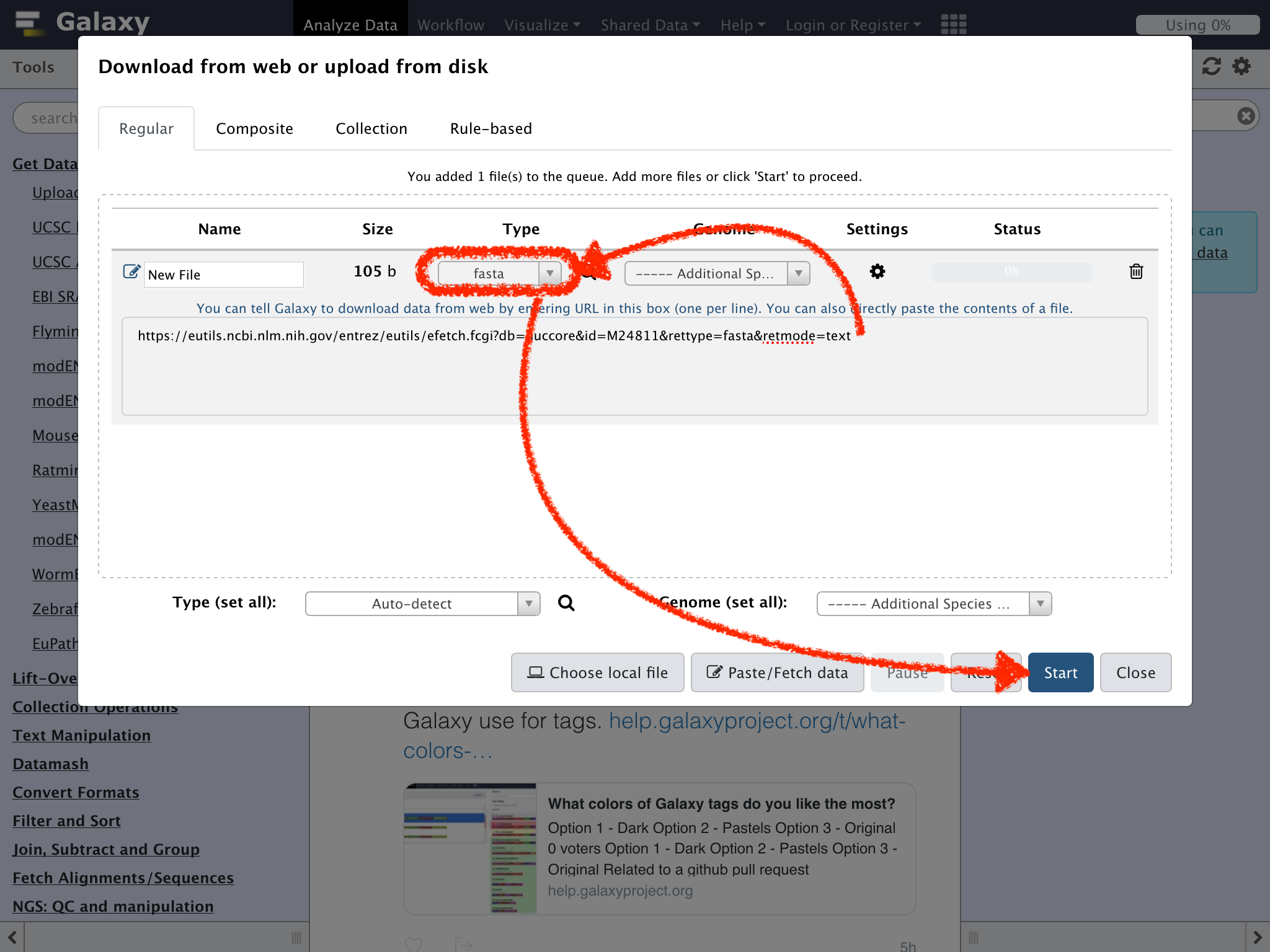
Statusが100%になったら"Close"をクリックする
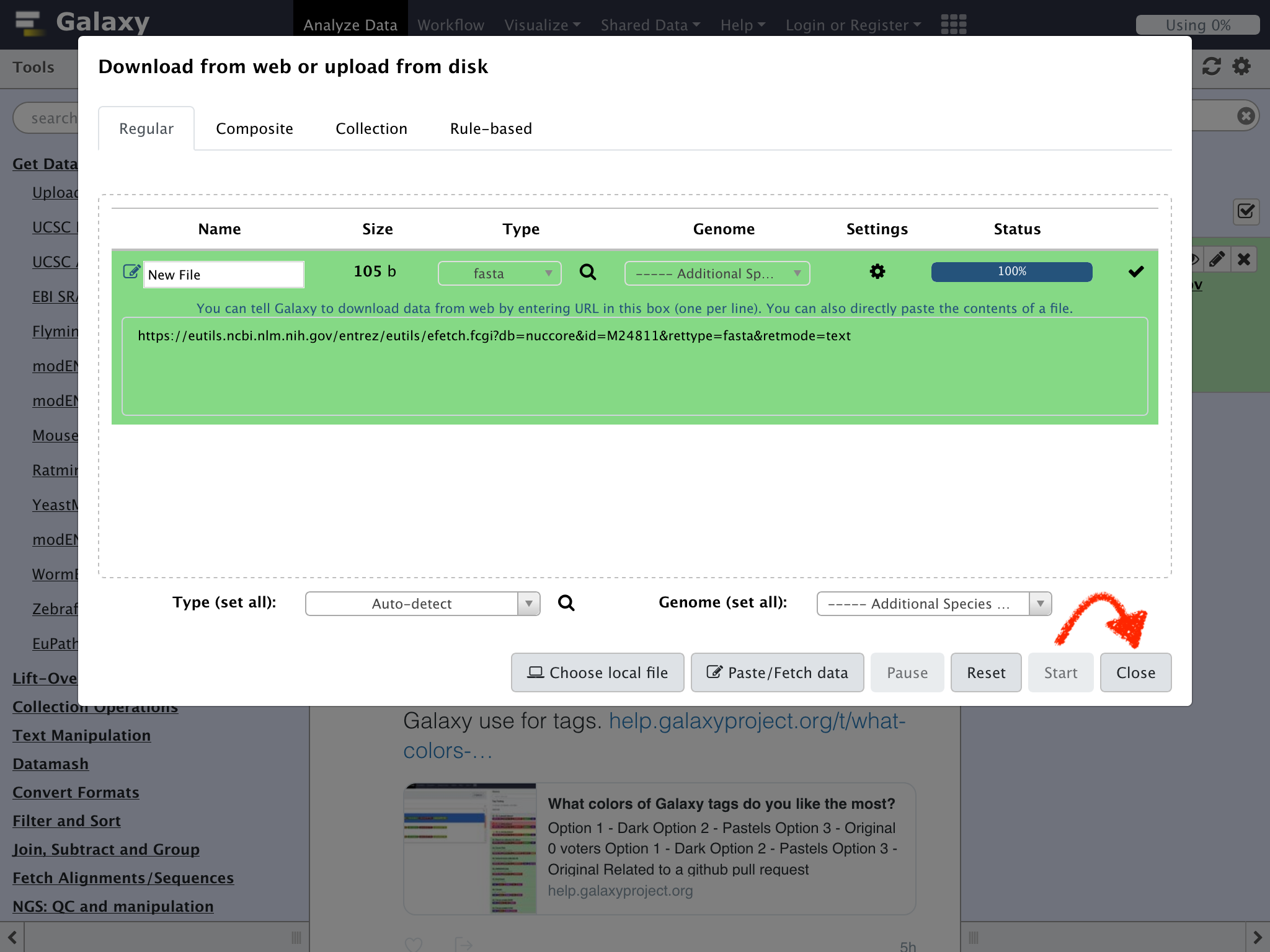
Historyペインにダウンロードの履歴が表示される
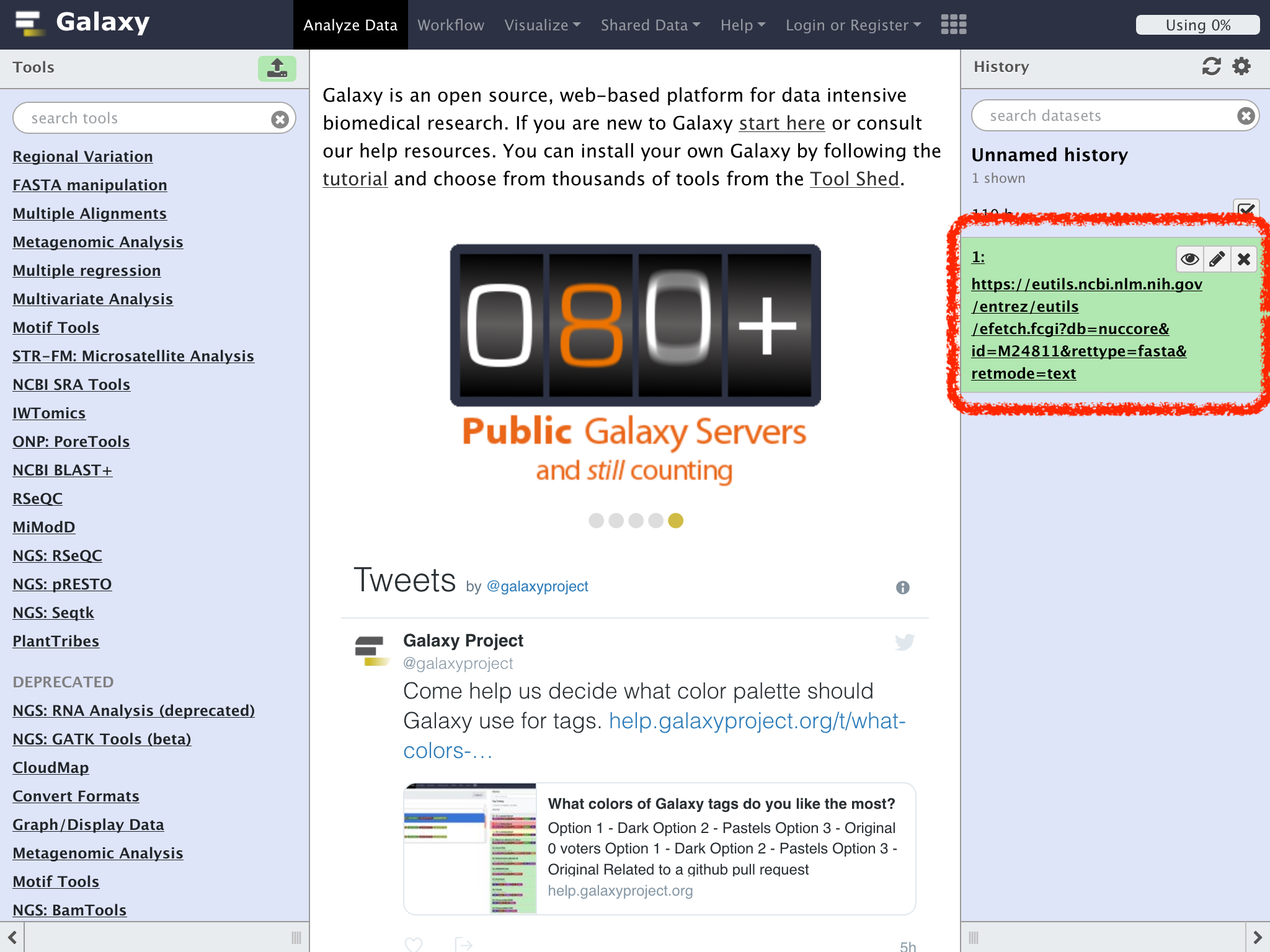
場合によっては、この部分の背景が黄色で実行中を示している事があるかもしれない
その時は実行が終了するまで少し待ってみよう
Blastの実行
先程登録したFastaの配列にBLASTツールを利用してホモロジーサーチを行ってみよう
NCBI BLAST+ => NCBI BLAST + blastn Search nucleotide database with nucleotide query sequence(s)
ちょっと、見つけ難いかもしれないので、念のために手順を説明する
Toolsペインを下方向にスクロールして、**NCBI BLAST+**を探してクリックする
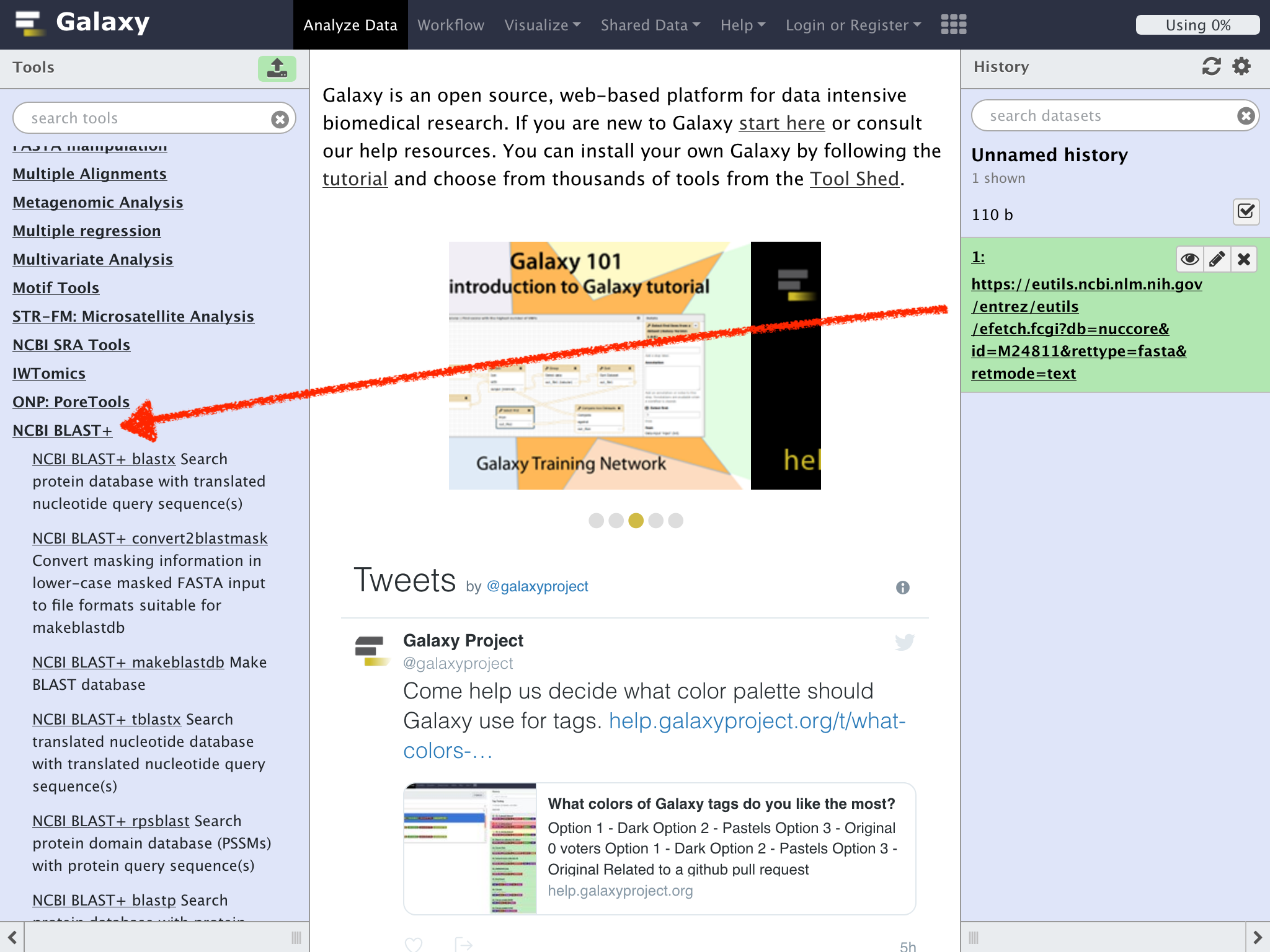
さらに、Toolsペインを下方向にスクロールしてNCBI BLAST + blastn Search nucleotide database with nucleotide query sequence(s)をクリックする
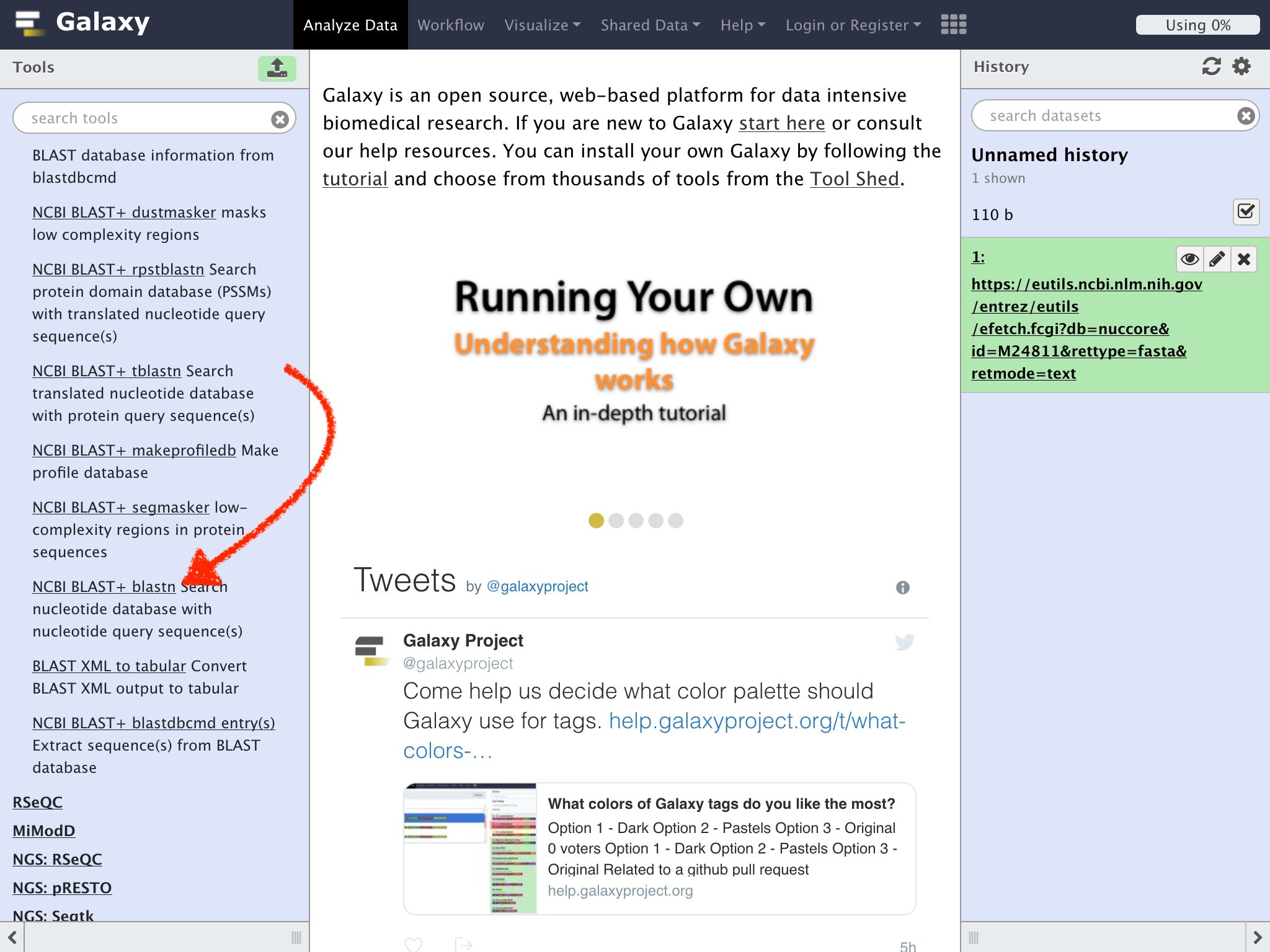
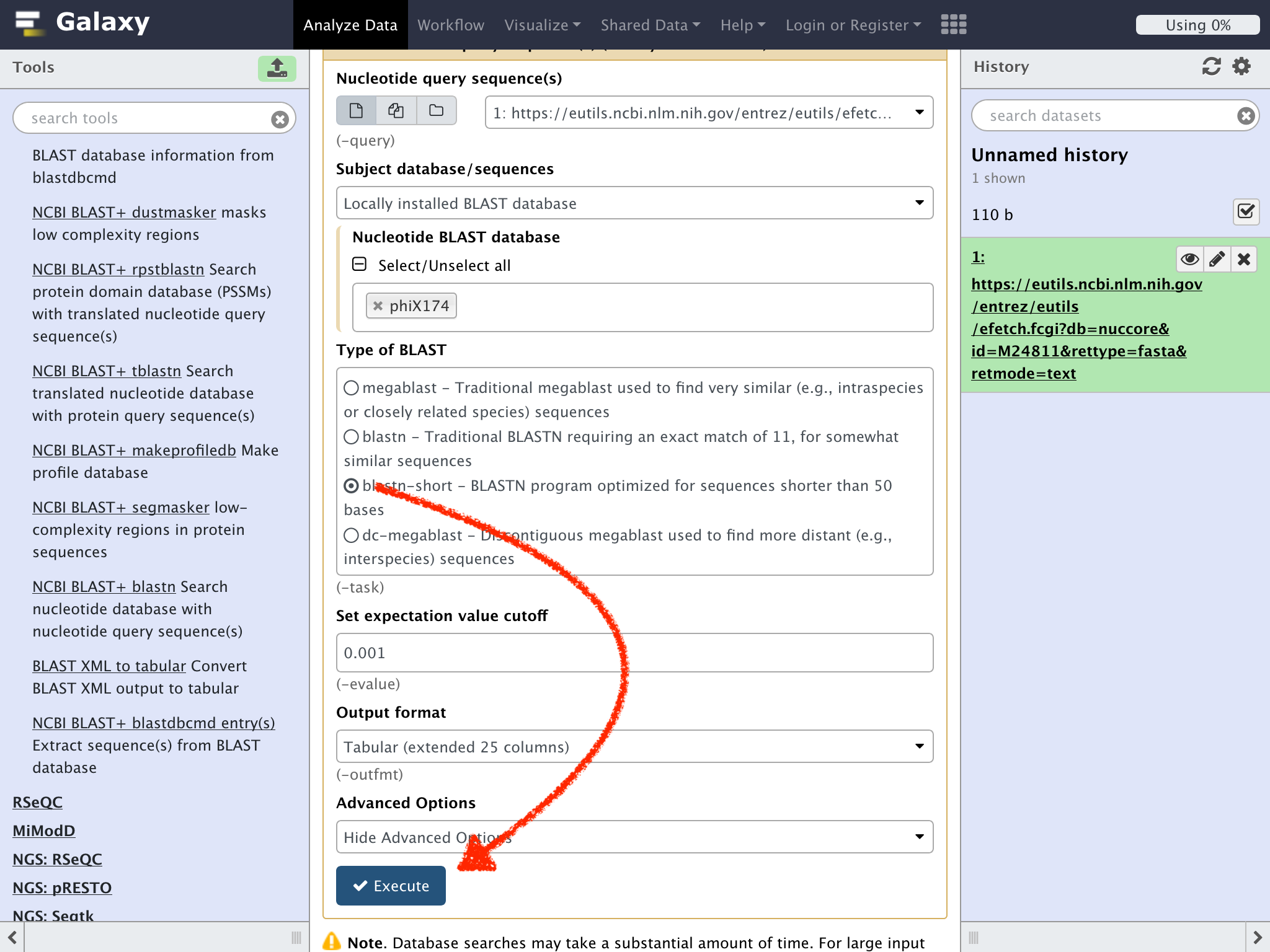
Nucleotide query sequence(s)のドロップダウンリストに先程の配列が指定されていることを確認して、Nucleotide BLAST databaseの "Select/Unselect all"のチェックボックスにチェックを入れる
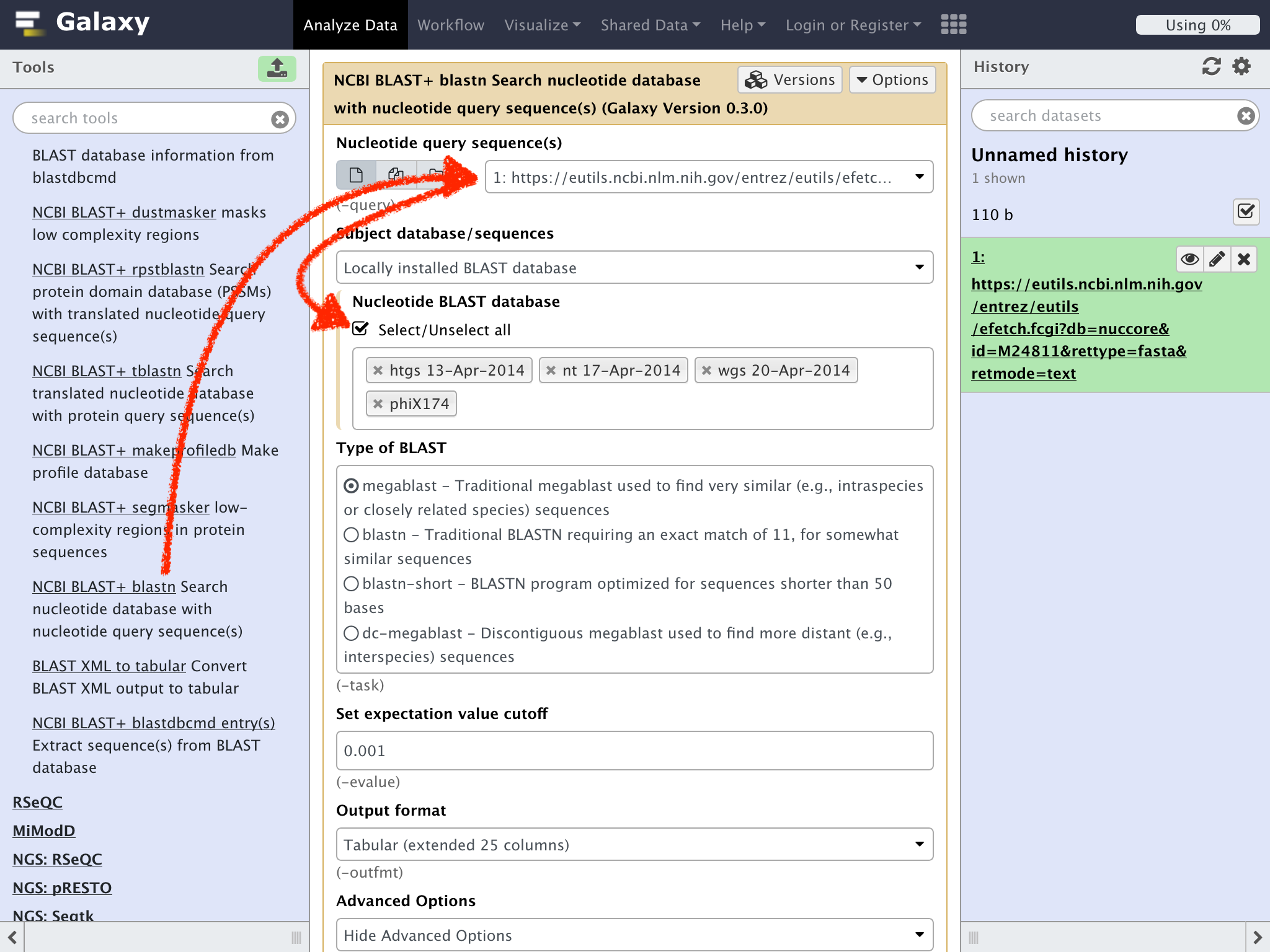
参照可能な配列がボックス内に表示されるので"phiX174"以外の"x"部分をクリックして他の配列を対象から外す
更に、Type of BLASTのラジオボタンで "blastn-short - BLASTN program optimized for sequences shorter than 50 bases"をクリックする
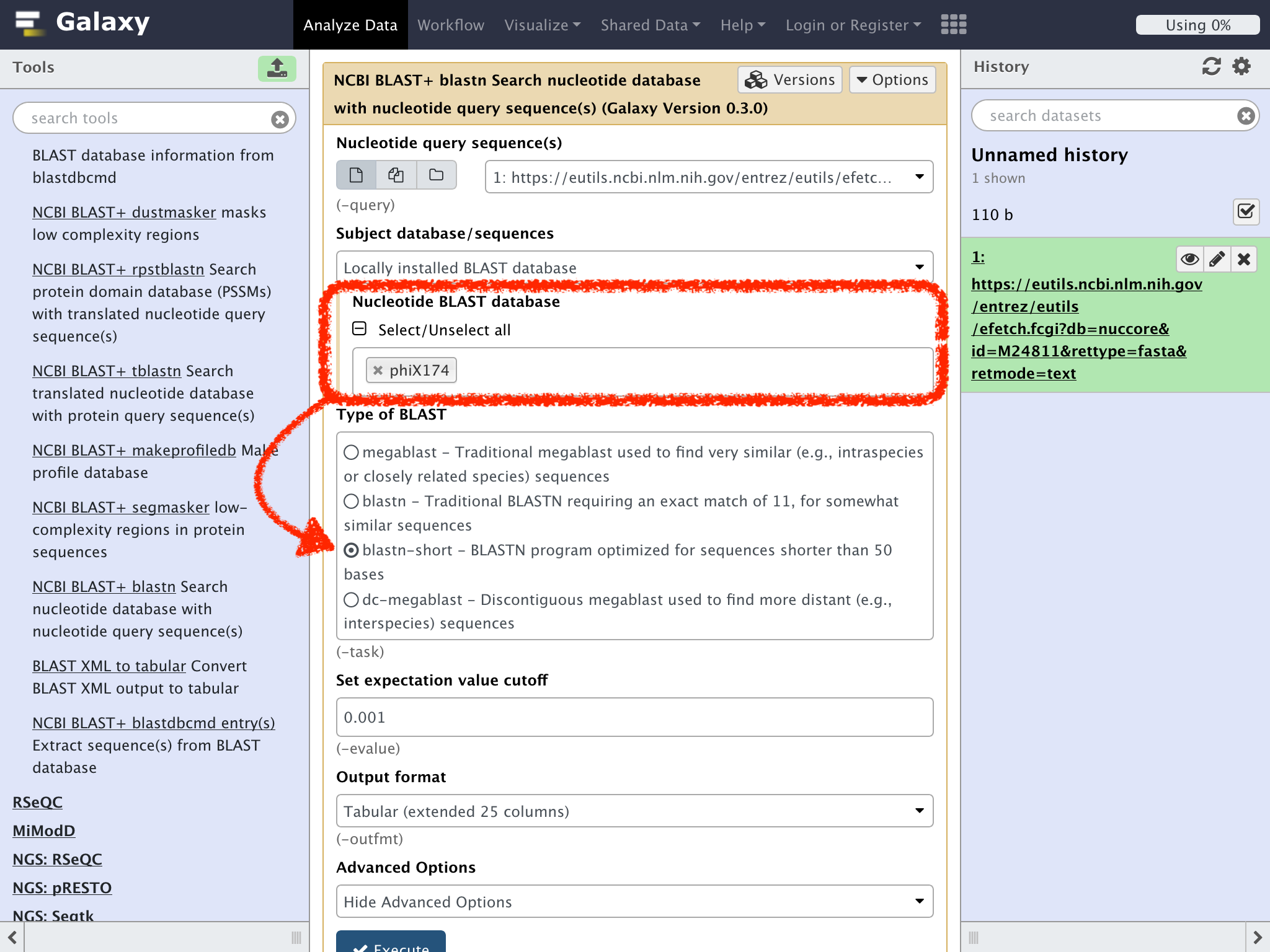
(間違えて、"phiX174"を消してしまった時は、"Select/Unselect all"のチェックボックスを2回クリックしてやり直そう)
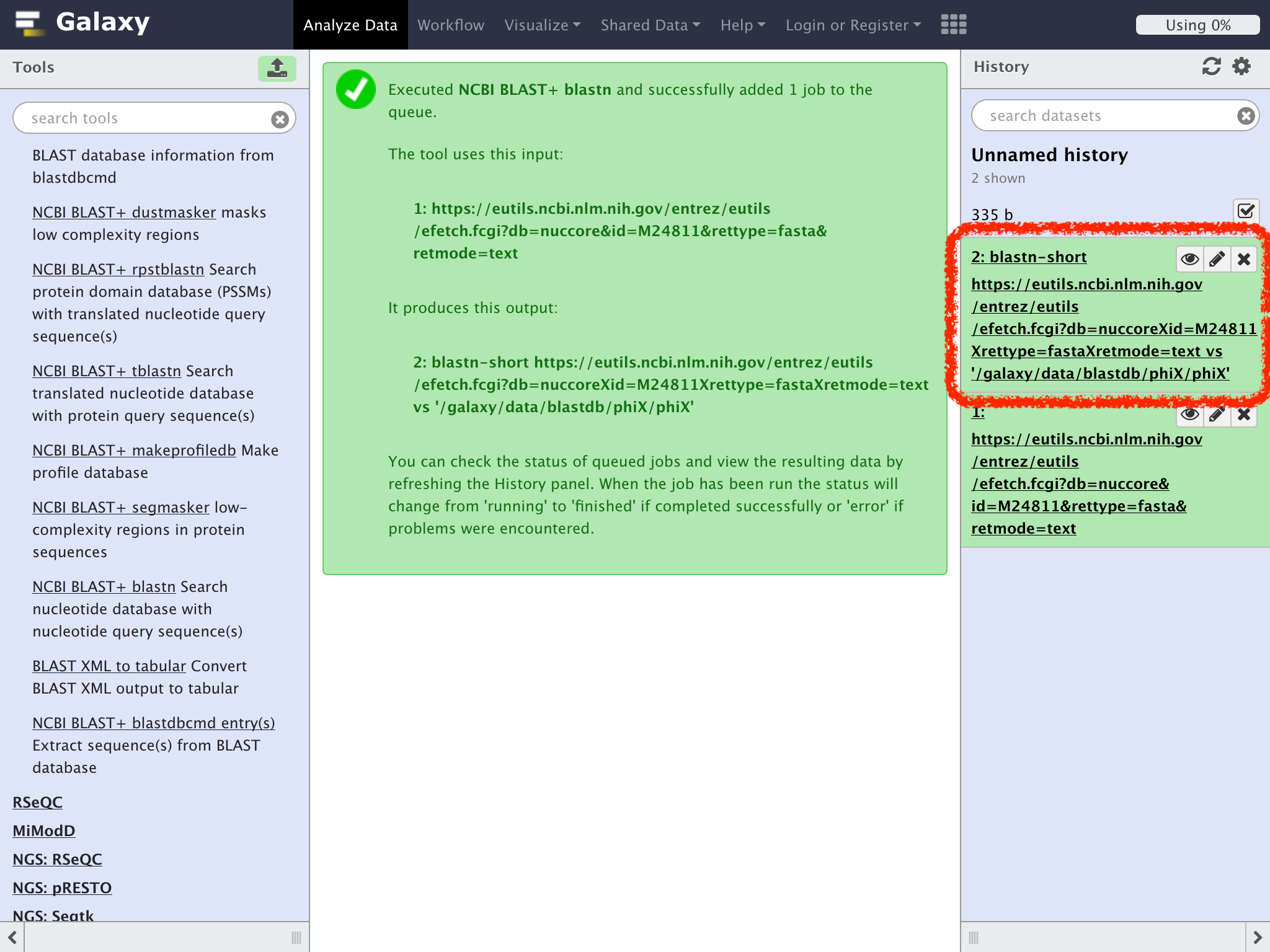
次のような画面でBLASTが実行待ちになる事が確認できる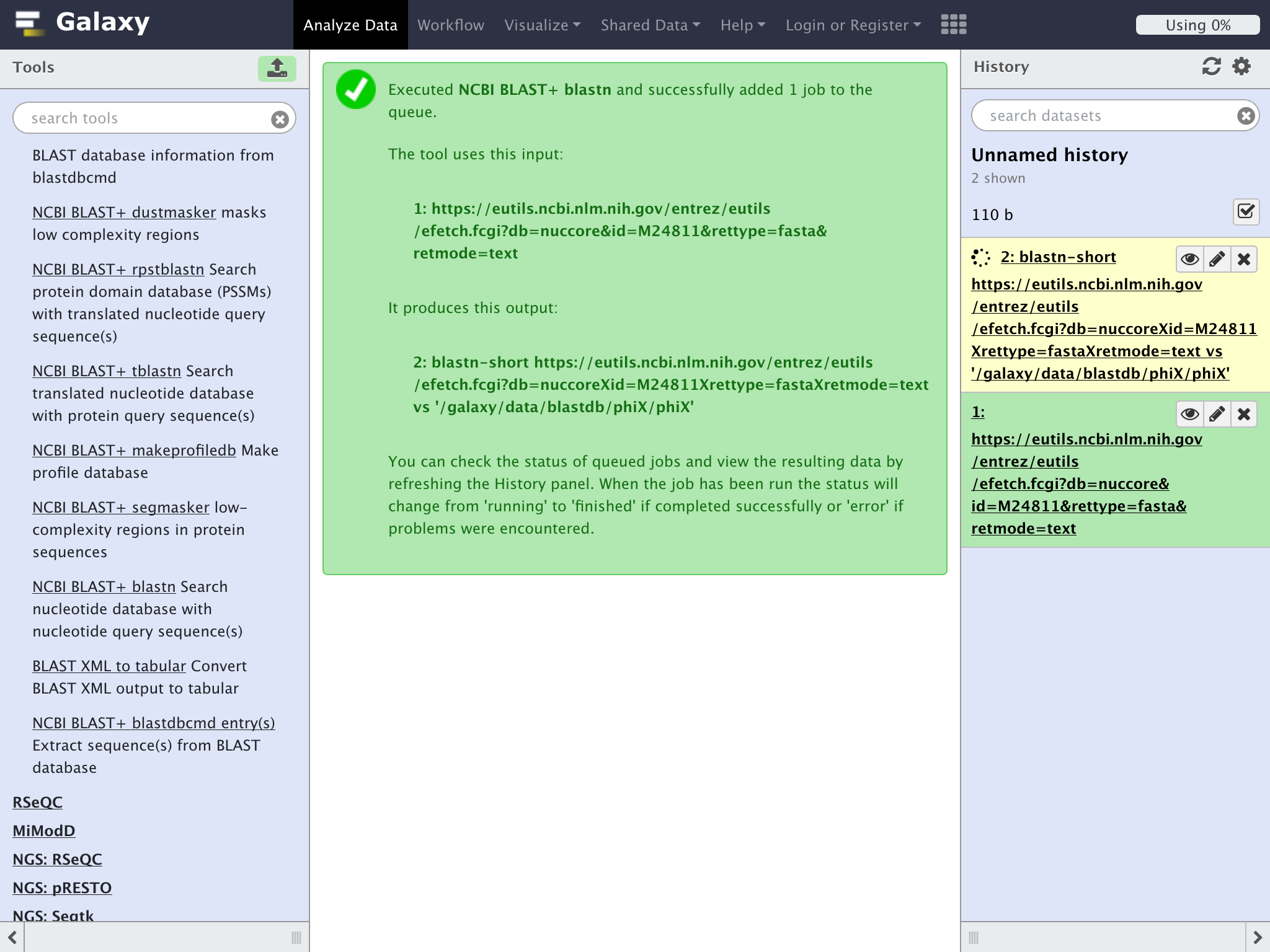
終了までにかかる時間はGalaxyサーバーの混雑具合によって異なるので、慌てずしばらく待ってみよう
目玉の形のアイコン(View data)をクリックして結果を確認しよう
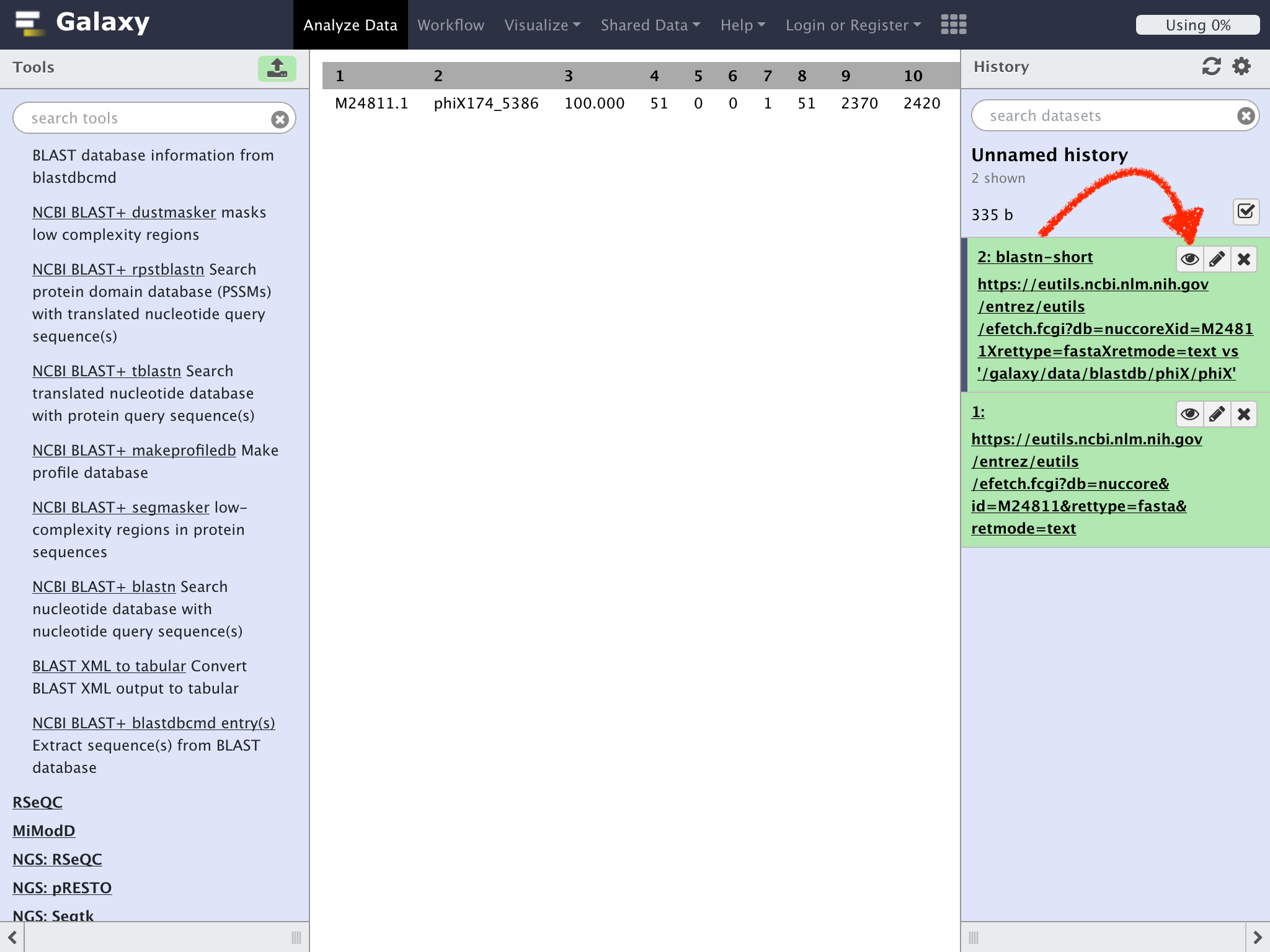
ここでの出力結果は重要では無いので、処理時間の節約のためにPhiXの配列の一部をPhiXと比較している
このようにして、未登録のユーザーでもGalaxyのツールを利用する事ができる
Entrez Programming Utilitiesについて
ここで利用しているEFetch
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgiに関する詳細は次のリンクを参照してほしい
https://www.ncbi.nlm.nih.gov/books/NBK25499/#_chapter4_EFetch_
今回は下記の記述で、配列データをGenBankからfasta形式で取得している
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=nuccore&id=M24811&rettype=fasta&retmode=text
| パラメータ | 指定内容 | 今回の値 |
|---|---|---|
| db | データベース名 | nuccore |
| id | 取得するデータベース上のID | M24811 |
| rettype | データの出力フォーマット | fasta |
| retmode | サーバーから転送時のデータ形式 | text |
今回はここまで![]()