テーブルの高さを設定する
heightの設定とVirtual DOMの利用
3. タブ区切りのファイルをtabulator で表示するを変更してテーブルの高さを指定しよう
これまでは、tabulatorでテーブルを表示する時にテーブル全体の高さを指定していなかった
この場合には、テーブルの高さ(縦方向の大きさ)はデータの行数によって変化し、大きなデータを読み込めばそれだけの大きなサイズのテーブルがブラウザに表示され、ブラウザ全体のスクロールで表示する行を変更していた
ここでは、テーブルの高さをheightパラメーターで明示的に指定する
tabulatorの設定部分を書き換えるために変更を加える
// tabulatorの設定
$("#gene-table").tabulator({
groupBy: "Symbol",
height: "200px",
columns: gene_columns
});
(画面イメージの都合から200pxと少し小さめのサイズとしている)
画面イメージからはわかりにくいけれど、テーブルの表示サイズがheightで指定したサイズとなっている
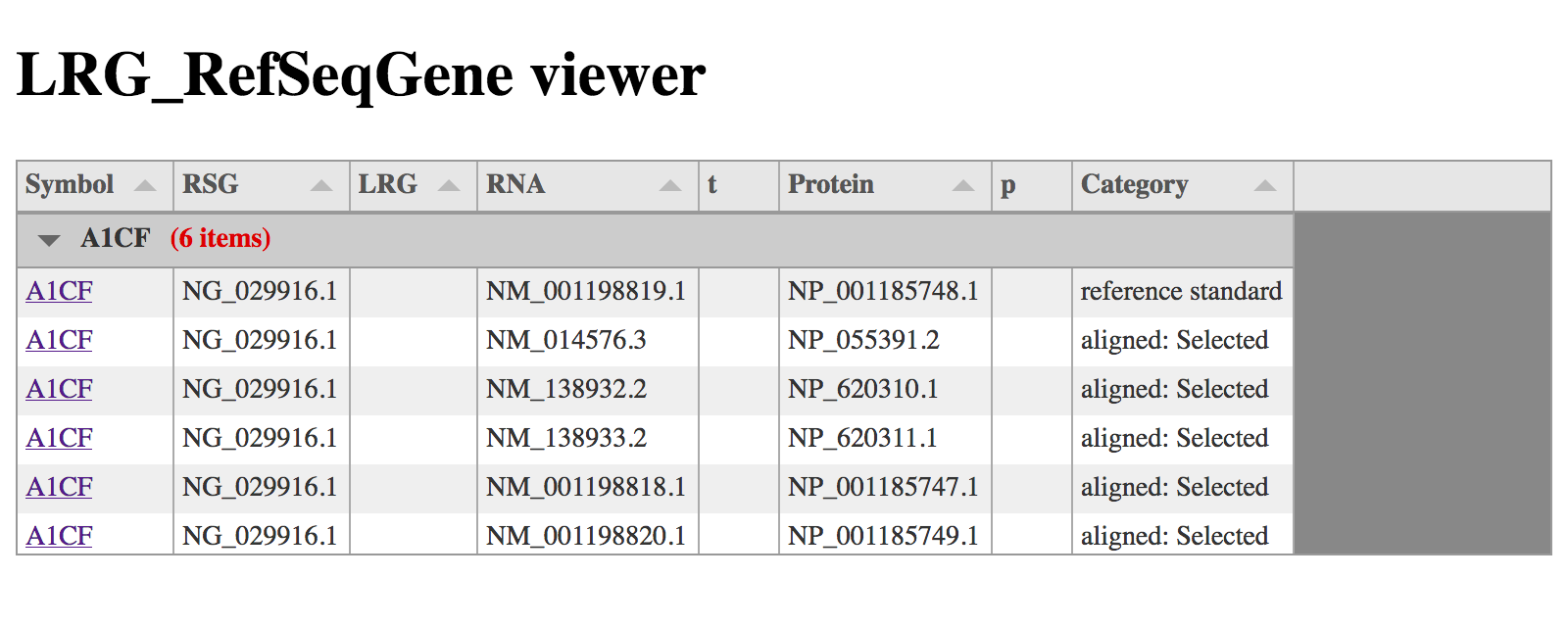
高さを明示的に指定する事の最大のメリットはtabulatorのVirtual DOM(Document Object Model)を利用できるということ
表示可能な領域(とその前後)の行のDOMだけ出力するという処理を行うことで不要な行を書き出さない
数百行のデータを出力する事は大きな問題では無いが、これが数万行となると、ブラウザーによるDOMの生成も描画も過大な負荷となってしまう
数万行のデータをjavascriptのデータ構造として持つ事は比較的容易でも、これを全てDOM要素として出力するのは大変
0.タブ区切りのファイルをtabulator で表示するでJSONのデータセットを作成した際に先頭の100行のデータに限定した理由はテーブル表示の負荷に配慮したもの
heightパラメーターを指定して表示行数を制限できるようにしたので、ここではLRG_RefSeqGeneのデータを全てJSONに変換
create_json.pyを下記の通りに変更して
# !/usr/bin/env python
# -*- coding: utf-8 -*-
from __future__ import print_function
import json
import csv
import argparse
__author__ = 'percipere'
__date__ = '2017/12/13'
__date_of_last_modification__ = '2017/12/25'
# Read data file and create JSON object
def convert_csv_to_json(filename='./LRG_RefSeqGene', delimiter='\t', maximum_lines=100, out_file="./output.json"):
"""
Read csv file and convert it to json file.
The first line of the csv should be delimiter separated column names.
:param filename: file name of the CSV
:param delimiter:
:param maximum_lines: 0 means all lines
"""
data_list = []
with open(filename, 'r') as file_handle:
reader = csv.DictReader(file_handle, delimiter=delimiter)
reader.fieldnames[0] = reader.fieldnames[0][1:] if reader.fieldnames[0].startswith('#') else reader.fieldnames[
0]
for row_id, row in enumerate(reader, start=1):
row.update({"id": row_id})
data_list.append(row)
if maximum_lines and row_id >= maximum_lines:
break
with open(out_file, 'w') as write_handle:
json.dump(data_list, write_handle)
if __name__ == "__main__":
parser = argparse.ArgumentParser(description="LRG_RefSeqGene to JSON file converter. output: output.json")
parser.add_argument('--rows', dest='maximum_lines', required=False, type=int, default=0,
help="Number of rows to convert.")
args = parser.parse_args()
convert_csv_to_json(maximum_lines=args.maximum_lines)
実行
$ python3 ./create_json.py
ブラウザのキャッシュのクリア
新しく作成したjsonの全データの読み込み時に、念のためブラウザに応じて下記の手順でブラウザのキャッシュをクリア
(キャッシュにデータが残っているとテーブルに表示される行数が以前のままとなってしまう)
- IEの場合
- Windows: "Ctrl" + "F5"
- Safariの場合
- Mac: "Option" + "Command" + "E"
- Firefoxの場合
- Windowsの場合 "Ctrl" + "F5"
- Macの場合 "Shft" + "Command" + "R"
- Google Chromeの場合
- Windowsの場合 "Ctrl" + "F5"
- Macの場合 "Shft" + "Command" + "R"
23170行のLRF_RefSeqGeneのデータ全てが表示できるようになる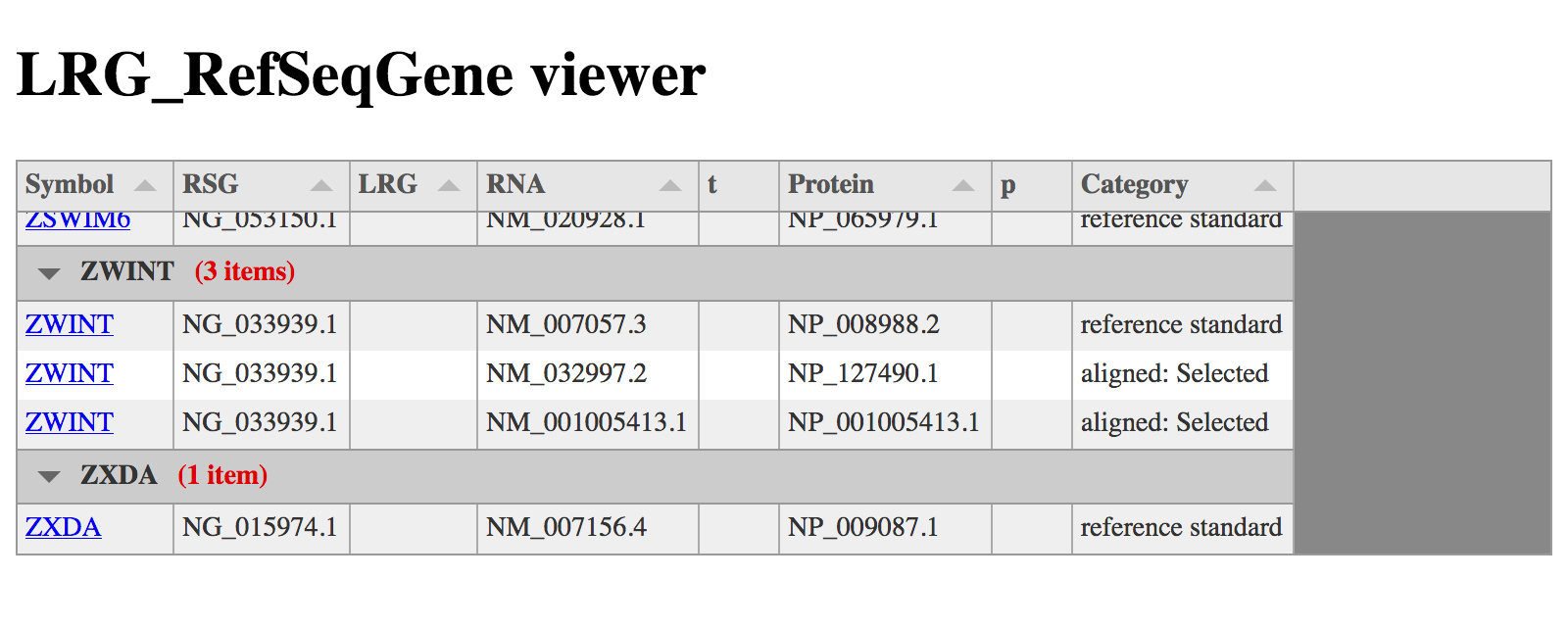
おまけ:
create_json.py は --rows で変換する行数を指定できる
例えば、最初の1000行を取り出すには次の通り
$ python3 ./create_json.py --rows 1000
3. タブ区切りのファイルをtabulator で表示するのように、height パラメータ無しで様々な行数のデータを表示してみるとVirtual DOMの良さを実感できる
今回はここまで![]()