はじめに
逆解析(分子探索)を行う際の、分子の生成に使用する部分構造(フラグメント)を準備する。
今回は、水溶解度データ(金子研究室HPのデータセット)に含まれる1290分子の構造をフラグメント化する。
検討するフラグメント手法は以下の2つ
- RECAP:分子を合成する際に逆合成の可能性が高い構造(アミド基やエステル基など11種類の部分構造)に基づいてフラグメント化する。(Retrosynthetic Combinatorial Analysis Procedure)
- BRICS:RECAPで着目していた11種類の構造を16種類に拡張したもの。(Breaking of Retrosynthetically Interesting Chemical Substructures)
前準備
モジュールのインストール
conda install -c conda-forge rdkit
メインコード
モジュールのインポート
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw, BRICS, Recap
データの準備
suppl = Chem.SDMolSupplier('logSdataset1290_2d.sdf')
mols_list = [mol for mol in suppl if mol is not None]
RECAPによるフラグメント化
1分子のみ分解してみる(お試し)
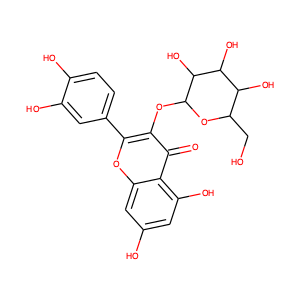
mol_test = Chem.MolFromSmiles('O=c1c(OC2OC(CO)C(O)C(O)C2O)c(-c2ccc(O)c(O)c2)oc2cc(O)cc(O)c12')
Draw.MolToFile(mol_test,'mol_test.png')
# RECAPの実行分解
decomp = Chem.Recap.RecapDecompose(mol_test)
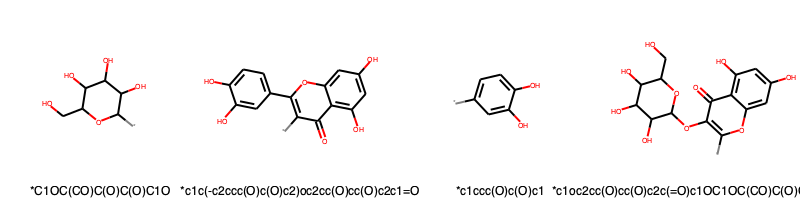
# 1段階目のフラグメント化で得られた分子を描画してみる。
first_gen = [node.mol for node in decomp.children.values()]
img = Draw.MolsToGridImage(first_gen, molsPerRow=4, legends=[Chem.MolToSmiles(m) for m in first_gen])
img.save('first_gen.png')
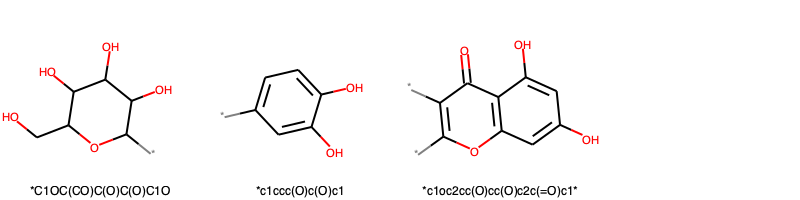
# leaf(これ以上分解できないフラグメント)を描画する
leaves = [leaf.mol for leaf in decomp.GetLeaves().values()]
img = Draw.MolsToGridImage(leaves, molsPerRow=4,legends=[Chem.MolToSmiles(m) for m in leaves])
img.save('leaves.png')
1290分子を分解する
leaves_list = []
for mol in mols_list:
decomp = Chem.Recap.RecapDecompose(mol)
leaves = [leaf.mol for leaf in decomp.GetLeaves().values()]
leaves_list = leaves_list + leaves
# smilesに変換して重複を削除する
leaves_list = list(set([Chem.MolToSmiles(mol) for mol in leaves_list]))
print('number of fragments:', len(leaves_list))
# >>>number of fragments: 537
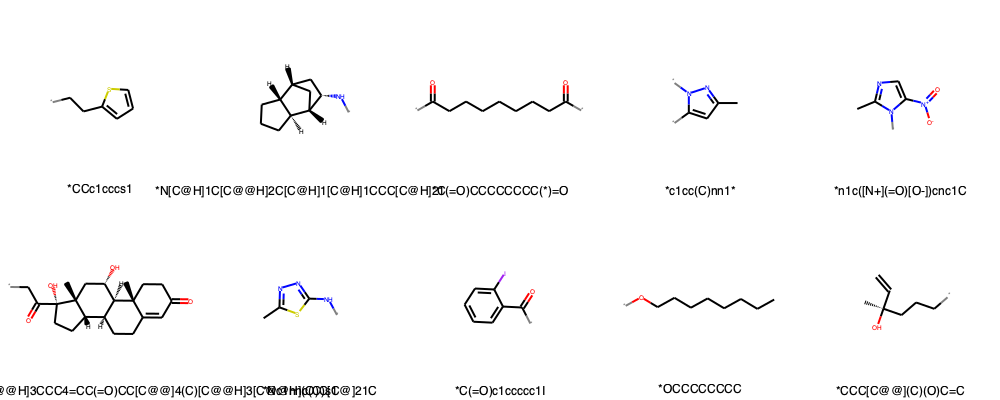
# 先頭10フラグメントだけ描画してみる
fragments10 = [Chem.MolFromSmiles(smiles) for smiles in leaves_list[:10]]
img = Draw.MolsToGridImage(fragments10, molsPerRow=5,legends=leaves_list[:10])
img.save('fragments10_RECAP.png')
BRICSによるフラグメント化
1分子のみ分解してみる(お試し)
mol_test = Chem.MolFromSmiles('O=c1c(OC2OC(CO)C(O)C(O)C2O)c(-c2ccc(O)c(O)c2)oc2cc(O)cc(O)c12')
Draw.MolToFile(mol_test,'mol_test.png')
# BRICSの実行
decomp = BRICS.BRICSDecompose(mol_test)
fragments = [Chem.MolFromSmiles(smiles) for smiles in decomp]
img = Draw.MolsToGridImage(fragments, molsPerRow=5,legends=leaves_list[:10])
img.save('fragments_BRICS.png')
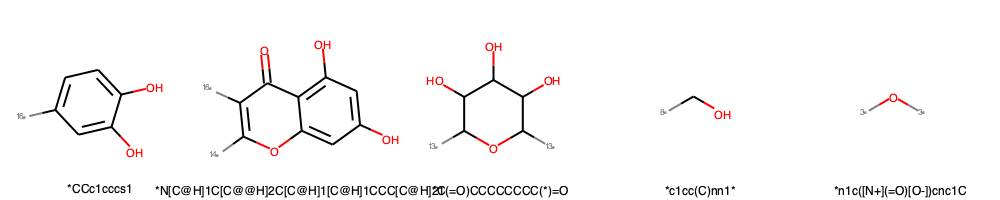
切断位置にダミーアトム([1*]や[2*]など)を挿入したSMILESのsetが得られる。
また、分解の際に考慮する構造の種類が多いため、RECAPの場合と比べて生成したフラグメントの数が多い。
1290分子を分解する
fragment_set = set()
for mol in mols_list:
fragment = BRICS.BRICSDecompose(mol)
fragment_set = fragment_set | fragment
fragment_list = list(fragment_set)
print('number of fragments:', len(leaves_list))
# >>>number of fragments: 1121
# 先頭10フラグメントだけ描画してみる
fragments10 = [Chem.MolFromSmiles(smiles) for smiles in fragment_list[:10]]
img = Draw.MolsToGridImage(fragments10, molsPerRow=5,legends=fragment_list[:10])
img.save('fragments10_BRICS.png')
RECAPでは537個のフラグメントを生成したのに対して、BRICSでは1121個ものフラグメントが生成できた。
(ダミーアトムのナンバリングが異なるフラグメントは、同一構造だとみなされていない可能性があるので要チェック)