本家ブログ(実践ケモインフォマティクス)もよろしくお願いします。
原子ごとに類似度の寄与率を可視化することができるメソッドSimilarityMapsを使って、TPSAなどの分子記述子に対する各原子の寄与を可視化する方法についてまとめた。
(本記事は「化学の新しいカタチ」の内容を簡潔にまとめたものです。より詳しい内容は、そちらに載っています。
準備
モジュールのインストール
conda install -c conda-forge rdkit
コード
分子の準備
練習用分子としてカフェイン分子を使用する。
import numpy as np
import matplotlib.pyplot as plt
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw, rdMolDescriptors
from rdkit.Chem.Draw import SimilarityMaps
mol = Chem.MolFromSmiles('Cn1c(=O)c2c(ncn2C)n(C)c1=O')
smiles = Chem.MolToSmiles(mol)
img = Draw.MolToImage(mol)
img.save('caffeine_molecule.png',bbox_inches='tight')
img
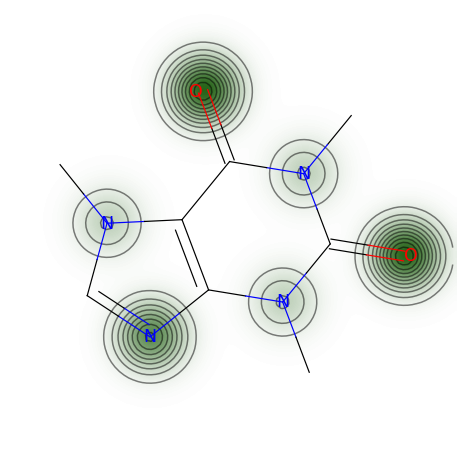
MolLogPへの寄与の計算と可視化
#_CalcCrippenContribs:各原子ごとに(MolLogPへの寄与,MolMRへの寄与)を格納したタプルが得られる。
crippen = rdMolDescriptors._CalcCrippenContribs(mol)
mol_log = []
mol_mr = []
for x, y in crippen:
mol_log.append(x)
mol_mr.append(y)
fig = SimilarityMaps.GetSimilarityMapFromWeights(mol,
mol_log,
colorMap='PRGn',
alpha=0.5)
fig.savefig('MollogP.png',bbox_inches='tight')
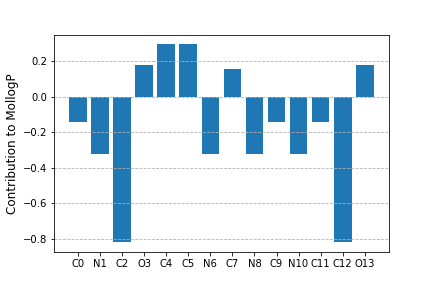
棒グラフでの寄与可視化
定量的に分かるように棒グラフでも可視化してみた。
#ついでに各原子の寄与を棒グラフでも表現してみる
atom_list = ['{}{}'.format(atom.GetSymbol(),atom.GetIdx()) for atom in mol.GetAtoms()]
#棒グラフの作成
x = np.arange(len(atom_list))
width = 0.8
fig, ax = plt.subplots()
bar = ax.bar(x, mol_log, width)
ax.set_xticks(x)
ax.set_xticklabels(atom_list)
ax.set_ylabel('Contribution to MollogP',fontsize=12)
plt.grid(linestyle='--',axis='y')
plt.savefig('bar_graph_mollogp.png')
plt.show()
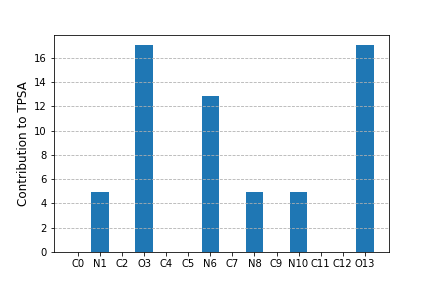
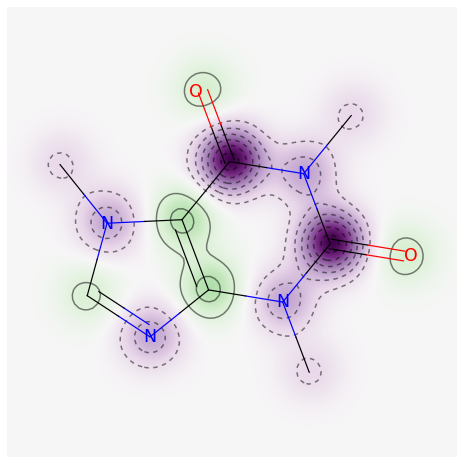
TPSAへの寄与の計算と可視化
#_CalcTPSAContribs(mol)::各原子ごとにのTPSAへの寄与を格納したタプルが得られる。
tpsa = rdMolDescriptors._CalcTPSAContribs(mol)
fig = SimilarityMaps.GetSimilarityMapFromWeights(mol,
weights=tpsa)
fig.savefig('tpsa.png',bbox_inches='tight')
棒グラフでの寄与可視化
MolLogPと同様に棒グラフでも可視化してみた。
#ついでに各原子の寄与を棒グラフでも表現してみる
atom_list = ['{}{}'.format(atom.GetSymbol(),atom.GetIdx()) for atom in mol.GetAtoms()]
#棒グラフの作成
x = np.arange(len(atom_list))
width = 0.8
fig, ax = plt.subplots()
bar = ax.bar(x, tpsa, width)
ax.set_xticks(x)
ax.set_xticklabels(atom_list)
ax.set_ylabel('Contribution to TPSA',fontsize=12)
plt.grid(linestyle='--',axis='y')
plt.savefig('bar_graph_tpsa.png')
plt.show()