背景
NCBIのgenomeデータベースを利用すると任意の細菌のゲノム配列やアノテーション情報(GenBankファイル)をブラウザからダウンロードできます。ただ、同じ細菌種の株ごとの違いを検証したいときなど、複数のデータをブラウザから手動でダウンロードするのは大変です。そこで、NCBIから細菌ゲノムの登録情報が記載されたファイルを取得し、ターミナルのコマンドでGenBankファイルを自動・網羅的に取得してみます。
手法
任意の細菌種を検索し、登録されている株の登録情報を取得する。
- NCBIのgenomeデータベースでダウンロードしたい細菌名を検索します。ここでは大腸菌(Escherichia coli)とします。
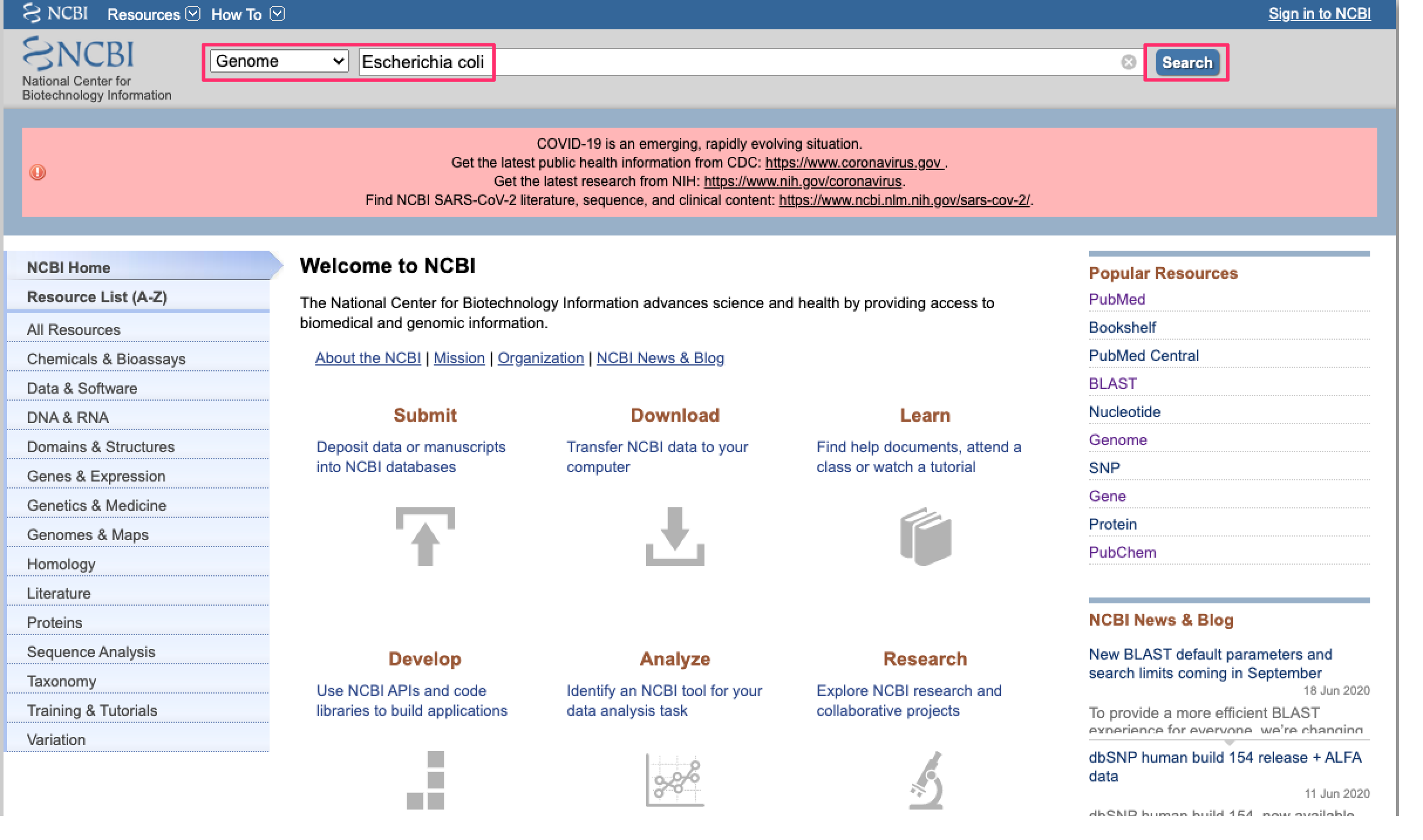
- 基準株の情報が画面上部に出てきます。「Browse the list」のlistをクリックします。
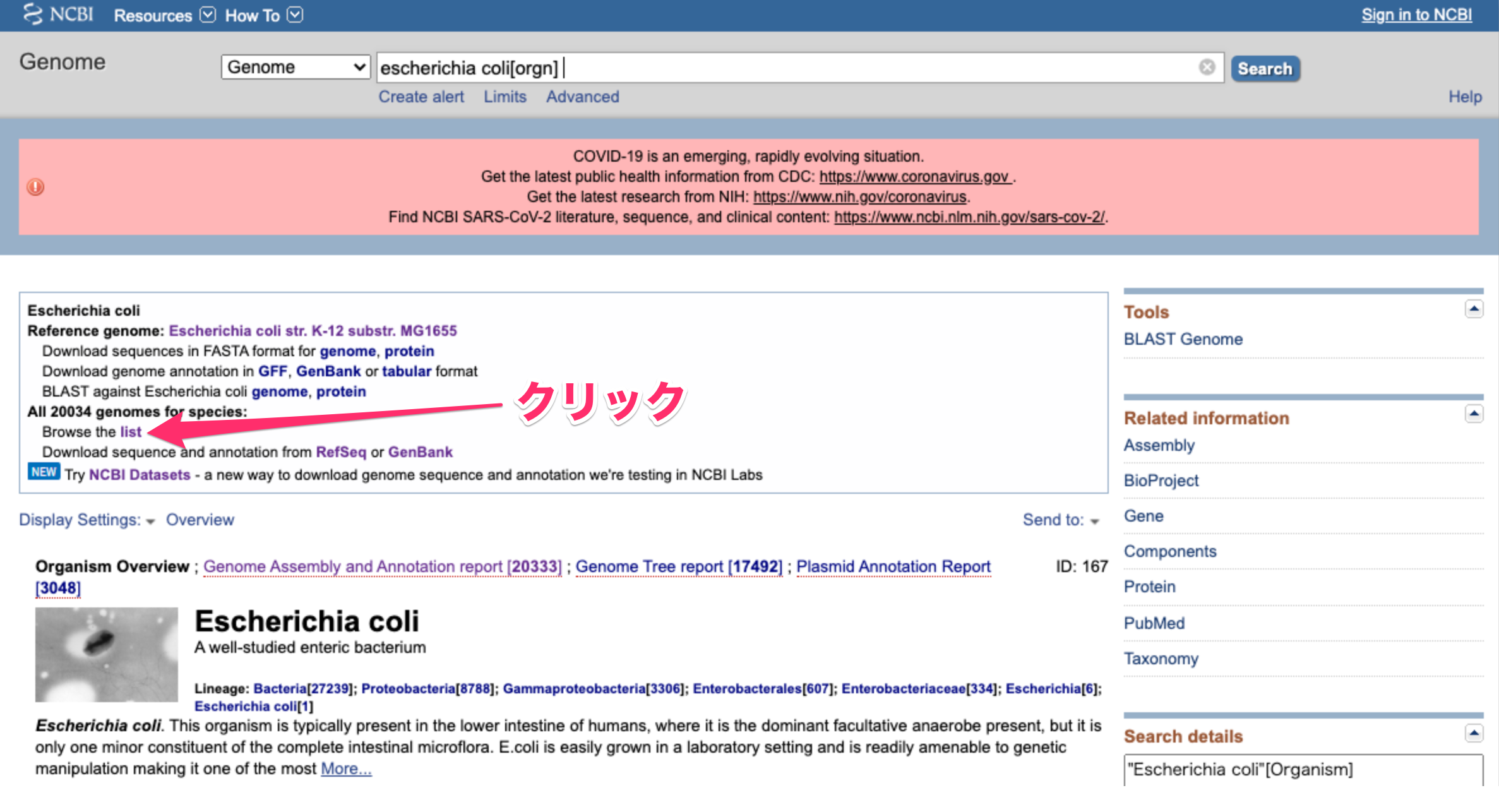
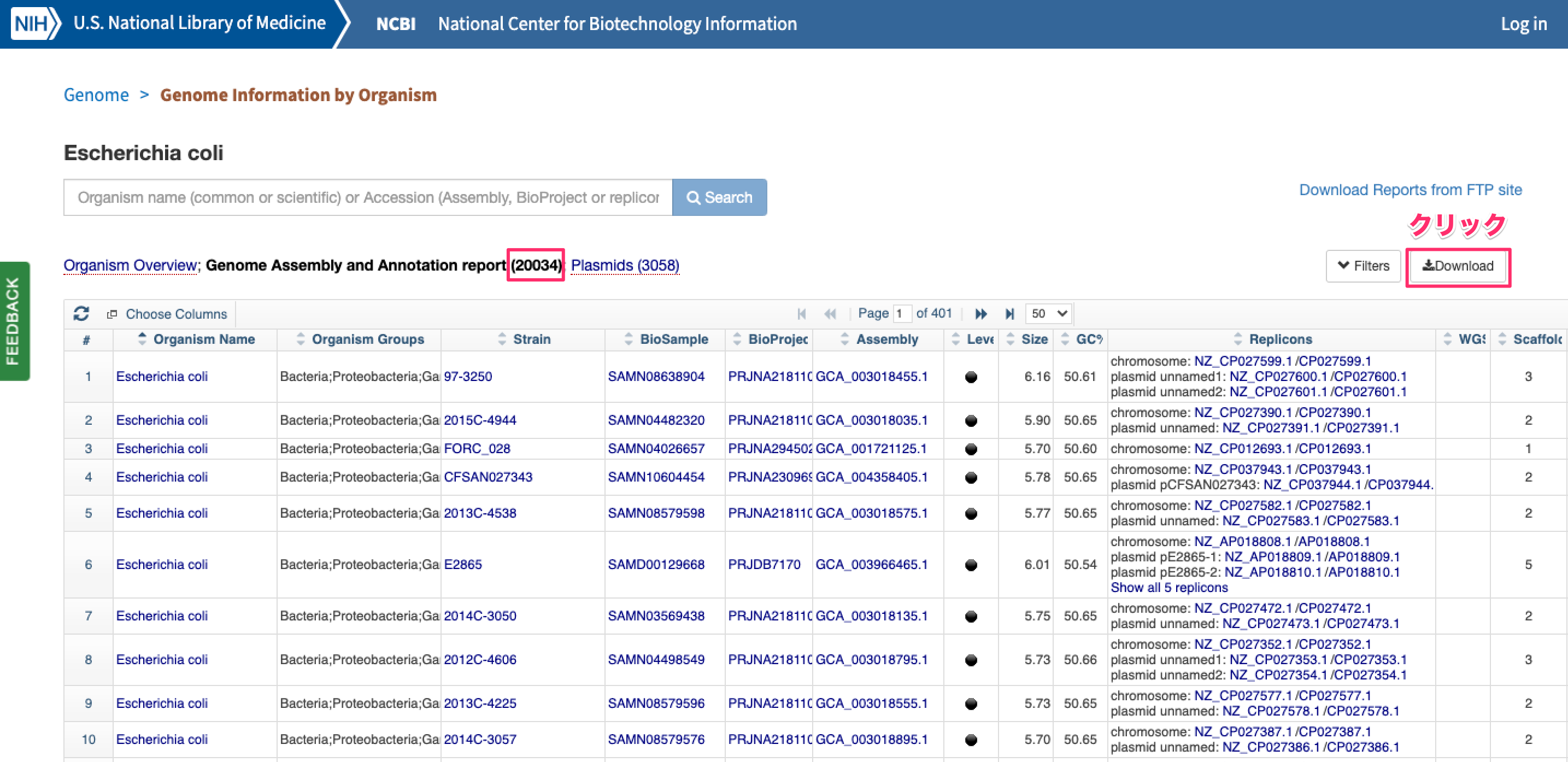
- 登録されている大腸菌ゲノムの一覧が表示されます。2020年6月23日現在で20,034の大腸菌ゲノムが登録されているようです。検索ボックスにキーワードを入力すれば、さらに絞り込むこともできます。
- 「Download」をクリックすると、20,034の大腸菌ゲノムの登録情報が記載されたcsvファイルがダウンロードされます。
-
エクセルなどで開きます。株名やゲノムサイズ、CDS数などが一目で分かります。今回使用するのは右から2番目にある「GenBank FTP」列です。


-
試しに1番上の「Escherichia coli str. K-12 substr. MG1655」の「ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCA/000/005/845/GCA_000005845.2_ASM584v2 」をブラウザに入力してみます。
-
「Escherichia coli str. K-12 substr. MG1655」に関するファイルが格納されたディレクトリにアクセスできます。「GCA_000005845.2_ASM584v2_genomic.fna.gz」をクリックすればゲノム配列(fasta)、「GCA_000005845.2_ASM584v2_genomic.gbff.gz」をクリックすればGenBankファイルがダウンロードできます。
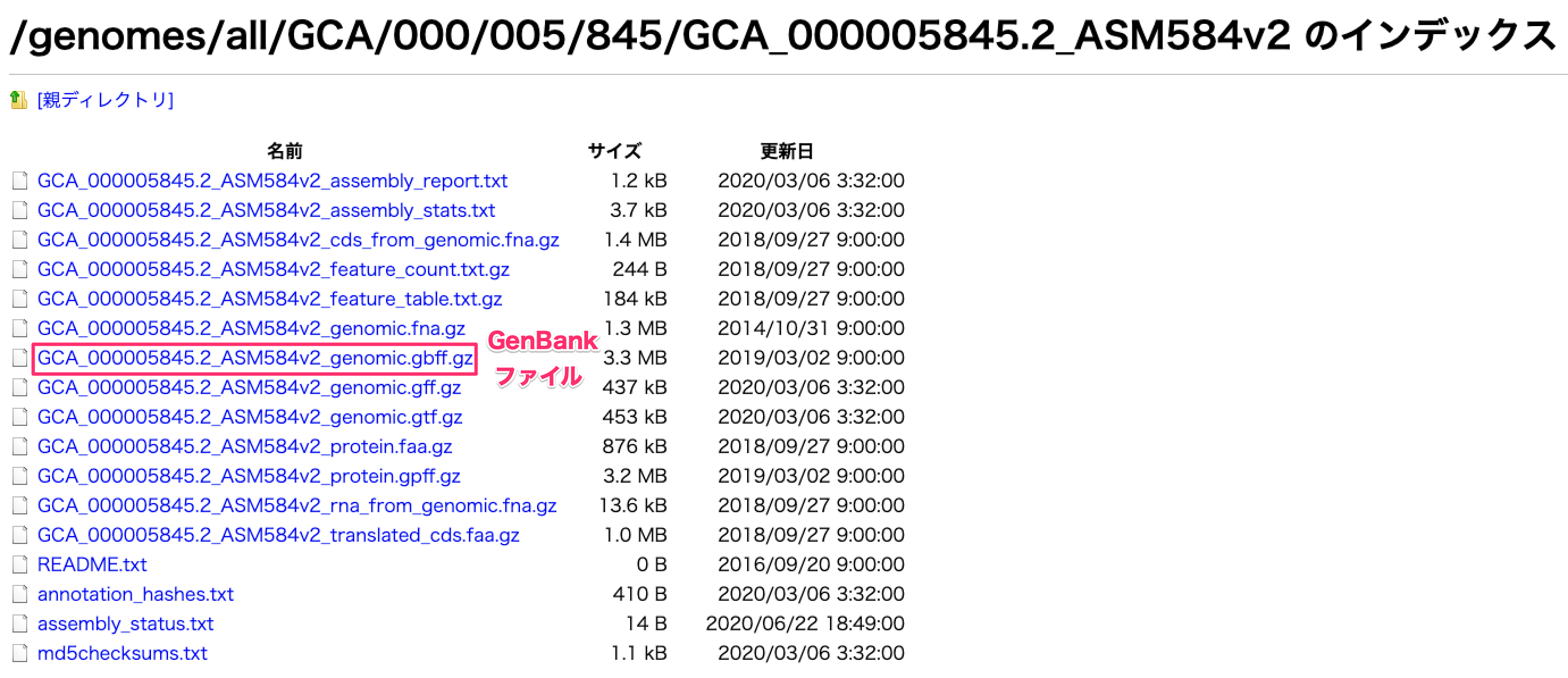
- 以上から、csvファイルに記載のあるFTPサーバー情報から入手したいファイルのURLを作成することで、ある細菌種のデータファイルを取得できることが分かります。
取得するファイルのURLを作成する。
- ここではGenBankファイル(gbff)を取得することにします。csvファイルに記載されているFTPサーバー情報は「ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCA/000/005/845/GCA_000005845.2_ASM584v2
」、実際のGenBnakファイルのアドレスは(ブラウザ上で右クリック→「リンクのアドレスをコピー」で取得できます)「ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCA/000/005/845/GCA_000005845.2_ASM584v2/GCA_000005845.2_ASM584v2_genomic.gbff.gz
」です。 - 見比べるとcsvファイルのアドレスに「/GCA_000005845.2_ASM584v2_genomic.gbff.gz」を追記すれば良いことが分かります。このうち「GCA_000005845.2_ASM584v2」は本ファイルが格納されているディレクトリ、すなわち最下層のディレクトリ名です。そのため、作成したいURLは「csvファイルのFTPサーバーアドレス + "/" + 最下層のディレクトリ名 + "_genomic.gbff.gz"」となります。
- エクセルの関数を使用しても作成できますが、ここではRを用いて作成してみます。
# 環境
sessionInfo()
R version 3.4.2 (2017-09-28)
Platform: x86_64-apple-darwin15.6.0 (64-bit)
Running under: macOS 10.14.1
# データの読み込み
dt <- read.csv("ダウンロードしたcsvファイル", stringsAsFactors = F)
# 最も下層のディレクトリ名の抽出
dirVec <- sapply(dt$GenBank.FTP, FUN = function(x) {
split <- strsplit(x, "/")[[1]]
return(split[length(split)])
})
# 取得するファイルのURLを作成
gbffVec <- paste(dt$GenBank.FTP, "/", dirVec,
"_genomic.gbff.gz", sep = "")
# 書き出し
write.table(gbffVec, "gbff_table.txt", quote = F, row.names = F,
col.names = F)
gbff_table.txtの中身は下記の通りです(20,034は多いので一部間引いています)。

ファイルのダウンロード
-
R上でダウンロードすることも可能ですが、ここではターミナルのwgetコマンドを用いてダウンロードしてみます。
-
wgetコマンドは-iオプションの後ろにファイル名を記載することで、ファイルに記載のあるURLにアクセスしてくれます。ここで先ほど作成したgbff_table.txtを指定します。また、-Pオプションでダウンロード先のディレクトリを指定することができます。
wget -i gbff_table.txt -P gbff/
```

- 後はgunzipコマンドでファイルを解凍すれば目的のGenBankファイルを利用することができます。ファイル数が多い場合はgunzipを並列に行えるpigzコマンドがお勧めです。
【pigz】Linux環境でマルチコアをフル活用して圧縮・解凍する方法
補足
もっと良い方法あれば教えて下さい。