背景
antiSMASHは、細菌および真菌ゲノム中の二次代謝物生合成遺伝子クラスターのゲノムワイドな同定やアノテーションが行えるツールです。Web版もありますが、ここではローカルにインストールします。
環境
PC: MacBook Pro 2020
OS: macOS Catalina (10.15.5)
shell: zsh (5.7.1)
方法
bioconda、Dockerを用いてインストールできます。マニュアルでインストールすることもできますが、依存関係にあるソフトが多いです。biocondaを用いる方法はすでに記事があるため(既知の二次代謝産物生合成遺伝子クラスターを検出する antiSMASH)、本記事ではDockerを使います。
参照
DockerをMacにインストールする
antiSMASH Documentation
Dockerが入っていれば非常に簡単です。
curl -q https://dl.secondarymetabolites.org/releases/latest/docker-run_antismash-full > ./run_antismash
chmod a+x ./run_antismash
# 必要であればPATHを通す
結果
試しに細菌のGenBankファイルをダウンロードして実行してみます。
# Streptomyces coelicolor A3(2)のGenBankファイルをダウンロード
curl -O https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/203/835/GCF_000203835.1_ASM20383v1/GCF_000203835.1_ASM20383v1_genomic.gbff.gz
gunzip GCF_000203835.1_ASM20383v1_genomic.gbff.gz
# 実行 run_antismash GenBankファイル アウトプットディレクトリ
./run_antismash GCF_000203835.1_ASM20383v1_genomic.gbff out_dir
アウトプットディレクトリに以下のようなファイルが作成されます。
index.htmlをブラウザで開きます。
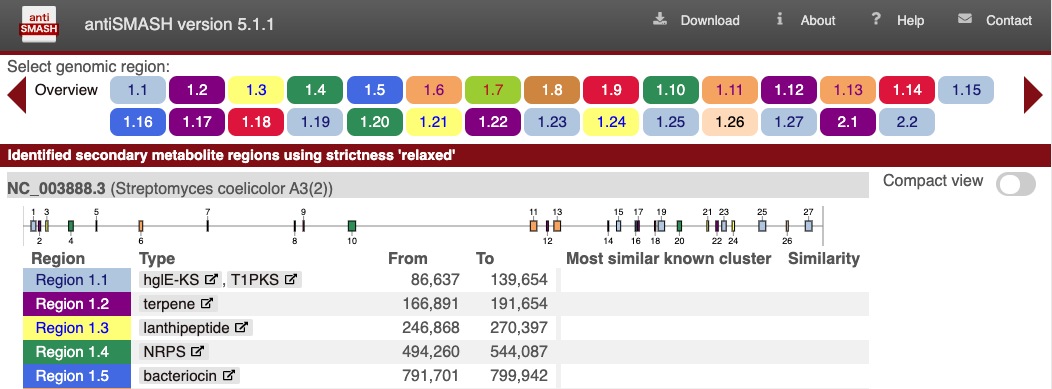
上部からOverview、ゲノム上の位置、タイプが示されます。
考察
Streptomyces coelicolor A3(2)はWeb版の「example output」としても使用されています(下記画像)。
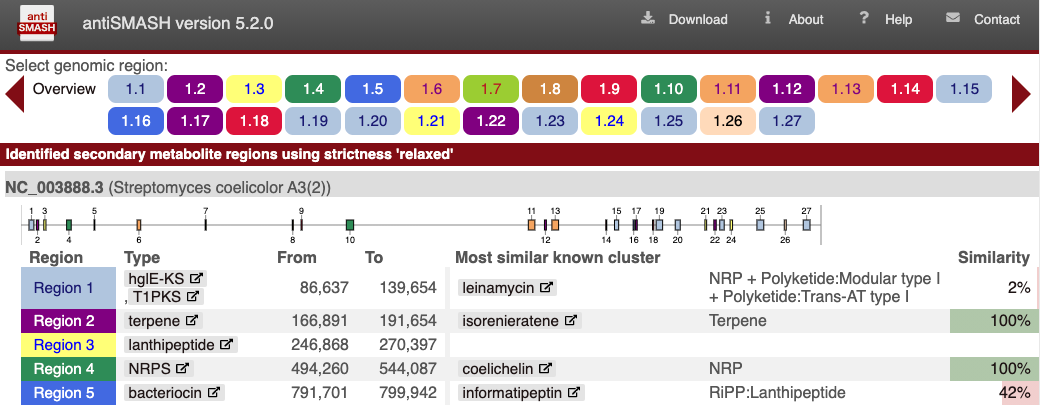
比較するとDocker版は5.1.1、Web版は5.2.0とweb版の方が新しいようです。検出されたクラスターの数と位置、クラスターのタイプは同じでした(Docer版の2.1、2.2はプラスミドです)。また、web版の方はMost similar known clusterとそのsimilarityについても表示してくれるようです(Docerでも実行時のオプションで設定できるのかもしれませんが)。
condaによるインストール(210217追記)
公式ドキュメントでは下記のコマンドでインストールできると記載されているが実際にやってみるとエラーになる。
conda create -n antismash antismash
conda activate antismash
download-antismash-databases
Preparing transaction: done
Verifying transaction: done
Executing transaction: done
ERROR conda.core.link:_execute(698): An error occurred while installing package 'bioconda::antismash-5.1.2-py_3'.
Rolling back transaction: done
LinkError: post-link script failed for package bioconda::antismash-5.1.2-py_3
location of failed script: /opt/anaconda3/envs/antismash/bin/.antismash-post-link.sh
==> script messages <==
<None>
# 以下省略
どうやらbiopythonのバージョンが競合しているらしい。
参考:antismash installation failing #292
インストール時にpythonとbiopythonのバージョンを指定することで解決した。
conda create -n antismash
conda activate antismash
conda install antismash biopython=1.76 python=3.7
download-antismash-databases
なお、condaのバージョンが古いと仮想環境のbinフォルダにpythonが存在しない旨のエラーが出る。condaのバージョンが古いのが原因でアップデート(4.3→4.9)すると解決した。
conda update -n base conda