はじめに
分子生物学の研究ではしばしばqPCRやトランスクリプトーム解析をすると思うのですが、そのさいにqPCRの増幅効率の計算や、qPCRによる濃度定量を行うと思います。それをPython3を使って計算し、可視化するスクリプトを書きました。
今回はトランスクリプトーム用のcDNAライブラリーを想定して書きますが、普通のqPCRの増幅効率計算にも使えます。
素人なので途中でめちゃめちゃ効率悪いことしてますが、改善案をコメントしていただけると嬉しいです。
とりあえずデータ準備
以下のようなqPCRのCqと、qPCRで計算された濃度の表を準備します。Libraryはサンプル名、Arb.concはcDNAサンプルの希釈倍率を示しています。今回は1000〜8000倍希釈のシークエンスを作っています。
| Library | Arb. conc. | Cq | Concentration |
|---|---|---|---|
| 1 | 8000 | 8.90 | 6.50 |
| 1 | 4000 | 10.00 | 3.20 |
| 1 | 2000 | 10.90 | 1.70 |
| 1 | 1000 | 12.20 | 1.00 |
| 2 | 8000 | 8.20 | 9.80 |
| 2 | 4000 | 10.00 | 3.20 |
| 2 | 2000 | 11.00 | 1.70 |
| 2 | 1000 | 12.20 | 0.80 |
更に、cDNAライブラリーの濃度をcDNAの配列長でノーマライズするために、Bioanalyzerなどで測定した各ライブラリーごとの平均長のデータを準備します。
| Library | Ave. Size |
|---|---|
| 1 | 310 |
| 2 | 330 |
| 3 | 325 |
| 4 | 320 |
| 5 | 324 |
| 6 | 340 |
| 7 | 320 |
| 8 | 345 |
| 9 | 350 |
pandasで読み込み
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from matplotlib import lines
import seaborn as sns
# cqのfileを読み込み
df = pd.read_csv('cq.csv')
# 平均長のファイルを読み込み
df_size = pd.read_csv('Ave_length.csv')
希釈濃度をCqと対応させるために常用対数にします。
また、今回は9つのライブラリーをいっぺんに描画したいので、dfを9つに分割します (色んな意味で余裕でもっと効率よくやれそうですが思いつきませんでした.......)。
# 希釈倍率をlog10変換
Log = np.log10(df['Arb. conc.'])
df['Log conc.'] = Log
# 分けたいカラムのリスト
cl = list(df.drop_duplicates(['Library'])['Library'])
# データフレームをLibraryの番号ごとに分ける
df_1= pd.DataFrame(index=[], columns=[])
df_2= pd.DataFrame(index=[], columns=[])
df_3= pd.DataFrame(index=[], columns=[])
df_4= pd.DataFrame(index=[], columns=[])
df_5= pd.DataFrame(index=[], columns=[])
df_6= pd.DataFrame(index=[], columns=[])
df_7= pd.DataFrame(index=[], columns=[])
df_8= pd.DataFrame(index=[], columns=[])
df_9= pd.DataFrame(index=[], columns=[])
for i, c in df.iterrows():
if c['Library'] == cl[0]:
df_1 = df_1.append(c)
if c['Library'] == cl[1]:
df_2 = df_2.append(c)
if c['Library'] == cl[2]:
df_3 = df_3.append(c)
if c['Library'] == cl[3]:
df_4 = df_4.append(c)
if c['Library'] == cl[4]:
df_5 = df_5.append(c)
if c['Library'] == cl[5]:
df_6 = df_6.append(c)
if c['Library'] == cl[6]:
df_7 = df_7.append(c)
if c['Library'] == cl[7]:
df_8 = df_8.append(c)
if c['Library'] == cl[8]:
df_9 = df_9.append(c)
描画と濃度計算
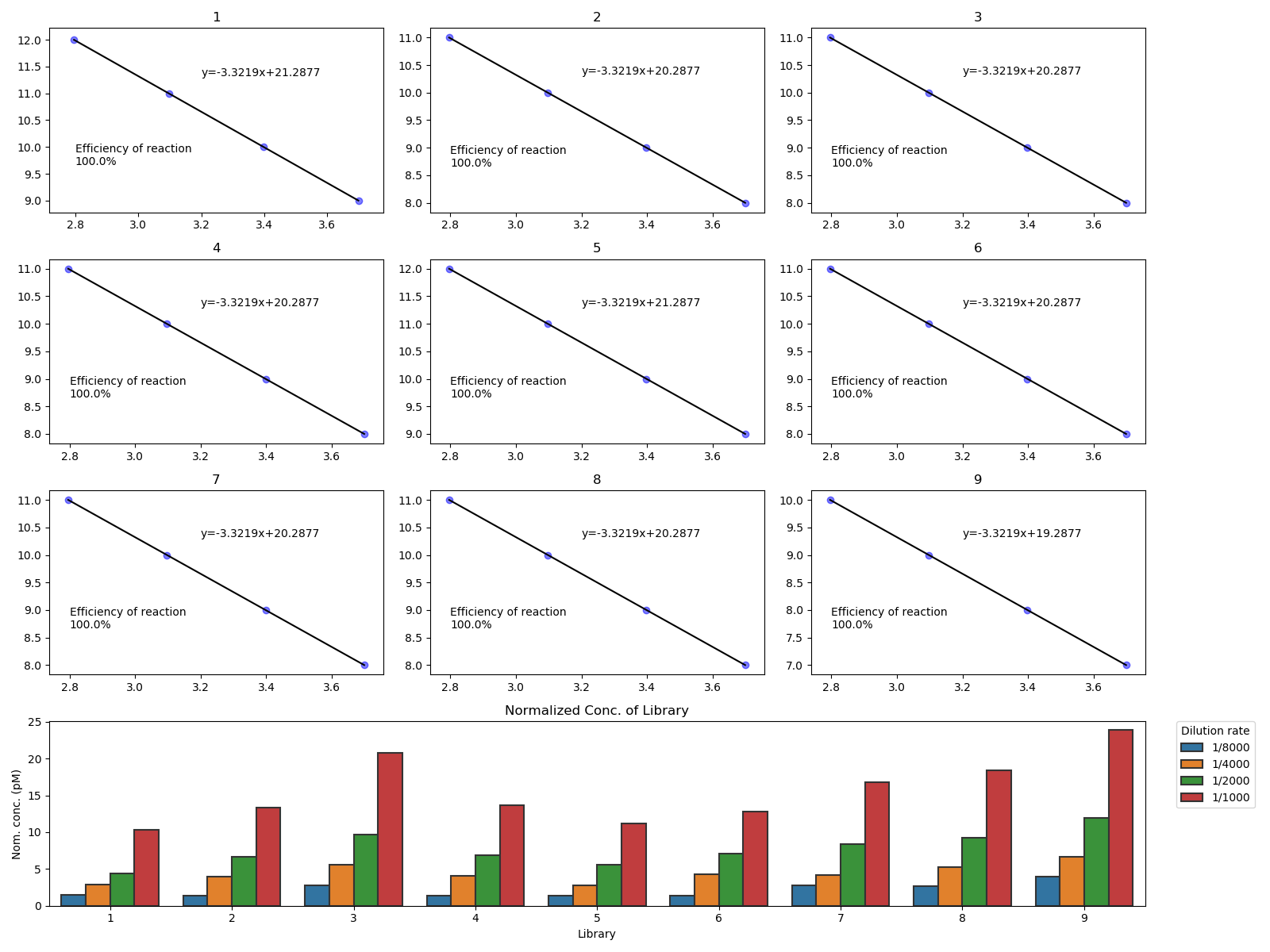
増幅効率を計算して描画し、更にノーマライズした濃度を棒グラフで出力します。
増幅効率の計算法はThermoのサイトなどがわかりやすいです。
# dfのリストと参照用の辞書を作る
df_l=['df_1', 'df_2', 'df_3', 'df_4','df_5', 'df_6', 'df_7','df_8', 'df_9']
df_dec={'df_1':df_1, 'df_2':df_2, 'df_3':df_3, 'df_4':df_4,'df_5':df_5, 'df_6':df_6, 'df_7':df_7,'df_8':df_8, 'df_9':df_9}
# ループでプロットの設定。enumerateでリスト長をiで参照している
# numpyで傾きを計算
fig = plt.figure()
for i, c in enumerate(df_l):
ax = fig.add_subplot(4, 3, i+1)
x = np.array(df_dec[c]['Log conc.']) # listで与えるとエラーが出るのでnumpy配列に変換
y = np.array(df_dec[c]['Cq'])
a, b = np.polyfit(x, y, 1)
y2 = a * x + b
ax.scatter(x,y,alpha=0.5,color="Blue",linewidths="1")
ax.plot(x, y2,color='black')
# 増幅効率の計算(a=-1/log10(1+e))
e=(10**(-1/a))-1
ax.text(3.2,a*3+b, 'y='+ str(round(a,4)) +'x+'+str(round(b,4)), fontsize=10)
ax.text(2.8,a*3.5+b, 'Efficiency of reaction'+'\n' + str(round(e*100,1))+'%', fontsize=10)
plt.title(i+1)
fig.set_size_inches(16, 12)
# 配列長でLibrary濃度の補正
nc_list =[]
for i, c in df.iterrows():
for n in range(20):
if c['Library'] == n:
nc = c['Concentration']*452/df_size.iat[n-1, 1]
nc_list.append(nc)
df['Nom. conc.'] = nc_list
# ノーマライズした濃度を棒グラフで表示
ax2 = fig.add_subplot(4, 1, 4)
g= sns.barplot(x = 'Library', y ='Nom. conc.',hue='Arb. conc.',data=df, dodge=True,capsize=.2, errwidth=1.5,linewidth=1.5, edgecolor=".2", ax=ax2)
plt.ylabel("Nom. conc. (pM)")
# legendのハンドルを取得
handles, _ = g.get_legend_handles_labels()
# レジェンドの名前変える
plt.legend(handles, ["1/8000", "1/4000", "1/2000", "1/1000"], bbox_to_anchor=(1.1,1), loc=0, borderaxespad=0, title= 'Dilution rate')
plt.subplots_adjust(wspace=.2, hspace=.4, left=0.08, bottom=.26, right=0.7, top=None)
plt.title('Normalized Conc. of Library')
plt.tight_layout()
plt.show()
すると以下の様な図が表示されます。
増幅効率100%になるようにデータを作ったので、Efficiency of reaction = 100%が表示されてます。
最後に、normalizeした濃度はdfに格納されているので、csvに出力して終了です。
df.to_csv("Library_Normconc.csv")
おわり
えらいニッチな記事を書きましたが大量にcDNAライブラリー作成してる人とかに役立てば幸いです。