はじめに
グラフ中に化合物の構造式や物性値を埋め込む方法を紹介します。例えばグラフを見ながらディスカッションする際に、あるプロットにカーソルを合わせるだけで、そのプロットの詳細(どのような構造式なのか、どのような特徴量を持つのか)を知ることができます。いちいちパワーポイントでプロットの詳細を書き込むという煩雑な作業から解放されます。
環境
- windos10
- anaconda
- python 3.9.17
- rdkit 2022.03.5
- bokeh 3.2.2
- numpy 1.22.3
- pandas 1.2.4
- xlsxwriter 3.1.2
RDKitのインストール方法は以下を参考にしました。
https://datachemeng.com/rdkit_install_import/
データの読み込み
以下のようなサンプル名やSMILES、物性値が書かれたcsvファイル(sample_data2.csv)を使ってグラフを描いていくことにします。
# データの読み込み
import pandas as pd
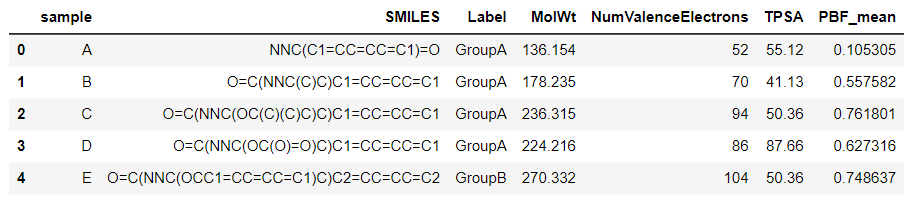
df = pd.read_csv("sample_data2.csv", encoding="shift_jis")
df.head()
構造式を付与する
以下のコードを実行することでSMILESを構造式(画像)に変換できます。
# ライブラリーのインストール
from IPython.display import SVG
from rdkit.Chem import AllChem, Draw, Descriptors, PandasTools
# SMILES→構造可視化
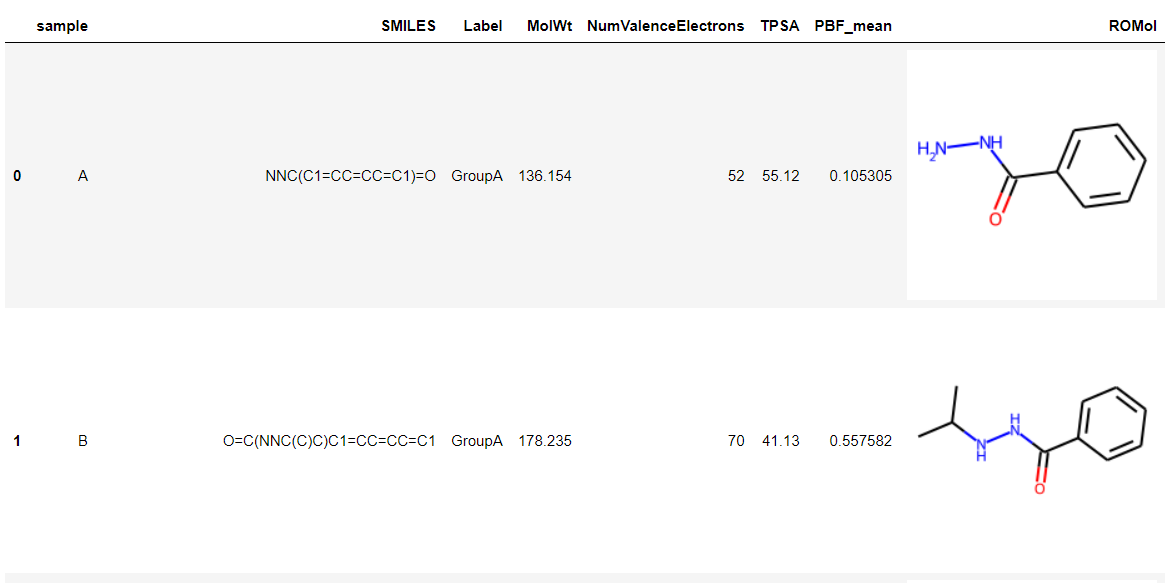
PandasTools.AddMoleculeColumnToFrame(df, "SMILES")
# データの表示
df.head()
※画像は先頭2行分だけ表示
# エクセルに保存(エラー回避のため欠損値は0埋めしておく)
import xlsxwriter
PandasTools.SaveXlsxFromFrame(df.fillna(0), "sample_data2_ROMol.xlsx", size=(150, 150))
可視化する
# ライブラリーのインポート
from bokeh.io import output_notebook, show
from bokeh.models import ColumnDataSource, HoverTool
from bokeh.plotting import figure
output_notebook()
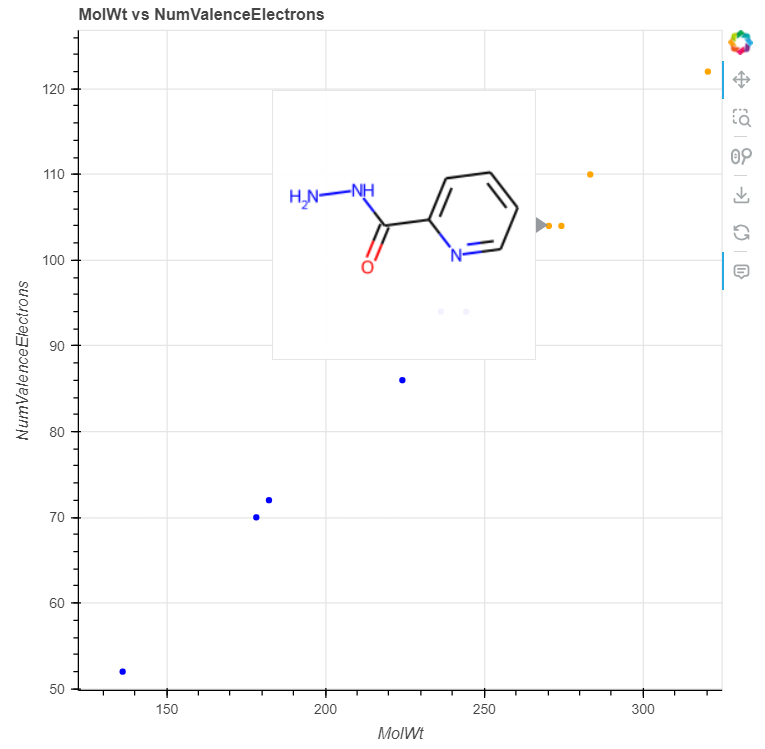
Label列でGroupAとGroupBとグループ分けしたデータをそれぞれ色分けして、横軸にMolWt、縦軸にNumValenceElectronsをとって各化合物をプロットしていきます。
# 色分けするためにデータをそれぞれ定義
df_A = df[df["Label"] == "GroupA"]
df_B = df[df["Label"] == "GroupB"]
# プロットするデータの定義
import base64
df_A['base64image'] = df_exp['ROMol'].map(
lambda x: str(x)[str(x).find('src="')+5:str(x).rfind('" alt')])
A_source = ColumnDataSource(df_A[['MolWt', 'NumValenceElectrons', 'base64image']])
df_B['base64image'] = df_exp['ROMol'].map(
lambda x: str(x)[str(x).find('src="')+5:str(x).rfind('" alt')])
B_source = ColumnDataSource(df_B[['MolWt', 'NumValenceElectrons', 'base64image']])
TOOLS = 'hover,save,pan,box_zoom,reset,wheel_zoom'
p = figure(title='MolWt vs NumValenceElectrons', tools=TOOLS)
p.scatter(x='MolWt', y='NumValenceElectrons', source=A_source, line_color=None, fill_color='blue', size=5)
p.scatter(x='MolWt', y='NumValenceElectrons', source=B_source, line_color=None, fill_color='orange', size=5)
p.xaxis.axis_label = 'MolWt'
p.yaxis.axis_label = 'NumValenceElectrons'
p.select_one(HoverTool).tooltips = """
<div>
<div>
<img src=@base64image></img>
</div>
</div>
"""
# グラフの表示
show(p)
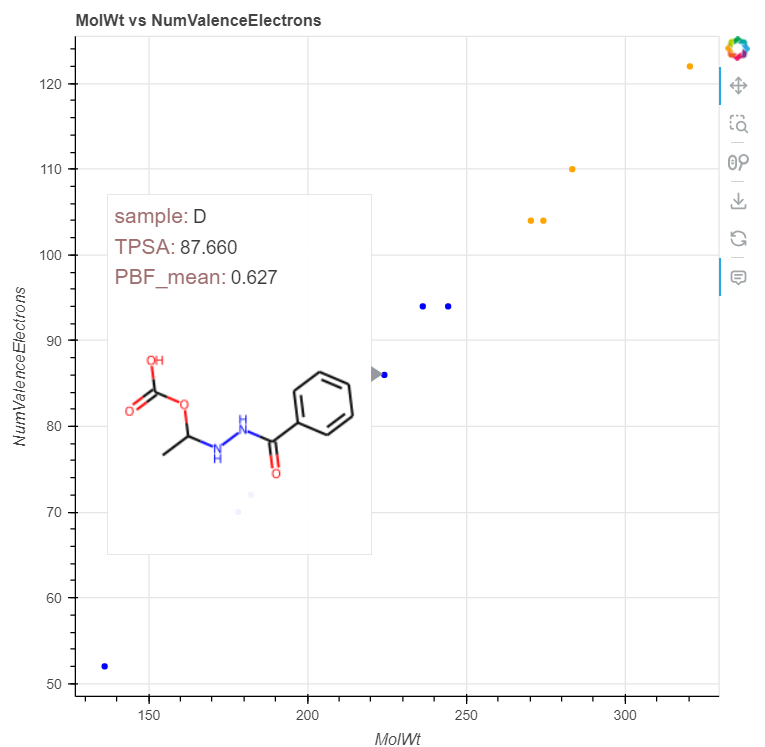
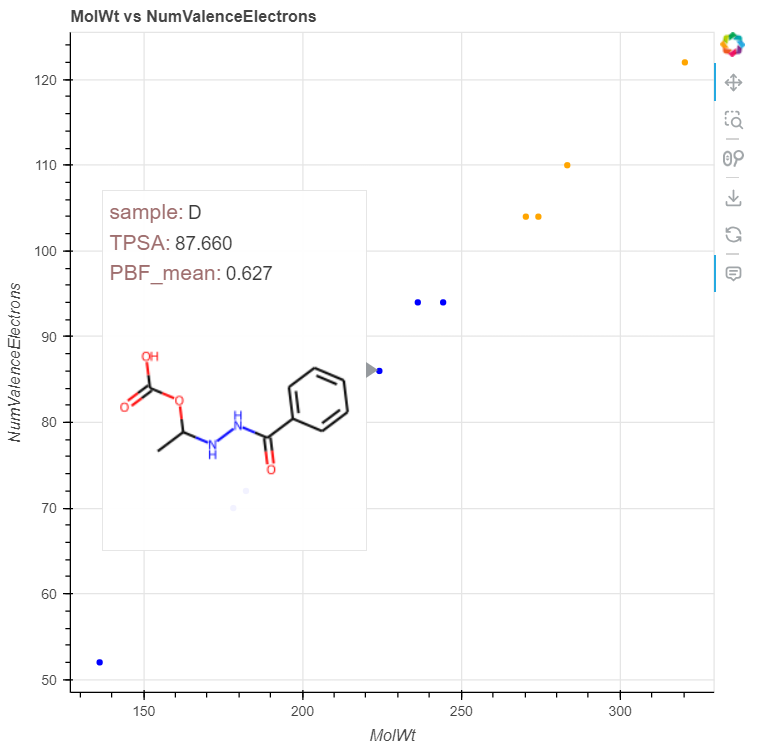
続いて先ほどのプロットに各化合物の物性値(TPSAとPBF_mean)の情報を埋め込んで表示させます。
# プロットするデータの定義
df_A['base64image'] = df_A['ROMol'].map(
lambda x: str(x)[str(x).find('src="')+5:str(x).rfind('" alt')])
A_source = ColumnDataSource(df_A[["sample", "MolWt", "NumValenceElectrons", "TPSA", "PBF_mean", "base64image"]])
df_B['base64image'] = df_B['ROMol'].map(
lambda x: str(x)[str(x).find('src="')+5:str(x).rfind('" alt')])
B_source = ColumnDataSource(df_B[["sample", "MolWt", "NumValenceElectrons", "TPSA", "PBF_mean", "base64image"]])
TOOLS = 'hover,save,pan,box_zoom,reset,wheel_zoom'
p = figure(title='MolWt vs NumValenceElectrons', tools=TOOLS)
p.scatter(x='MolWt', y='NumValenceElectrons', source=A_source, line_color=None, fill_color='blue', size=5)
p.scatter(x='MolWt', y='NumValenceElectrons', source=B_source, line_color=None, fill_color='orange', size=5)
p.xaxis.axis_label = 'MolWt'
p.yaxis.axis_label = 'NumValenceElectrons'
p.select_one(HoverTool).tooltips = """
<div>
<div>
<span style="font-size: 17px; color: #966;">sample:</span>
<span style="font-size: 15px; ">@sample</span>
</div>
<div>
<span style="font-size: 17px; color: #966;">TPSA:</span>
<span style="font-size: 15px; ">@TPSA</span>
</div>
<div>
<span style="font-size: 17px; color: #966;">PBF_mean:</span>
<span style="font-size: 15px; ">@PBF_mean</span>
</div>
<div>
<img src=@base64image></img>
</div>
</div>
"""
# グラフの表示
show(p)