はじめに
システム開発者であれば、一度は「システム関連図」を自動化できないかと考えたことがあるはずです。私も、過去の投稿記事でPDFや画像認識を使って自動生成できないか試みましたが、精度や汎用性に限界を感じました。最終的に、Excelのシェープ(円や四角)を利用して関連性を基づき作図する方法に行き着きました。結果としては十分な精度が得られたものの、シェープ同士の関係性を手作業で定義するのは非常に手間がかかり、現実的ではありません。
過去の試行錯誤については、以下の投稿記事にまとめています。
一方で、システム開発者として、もう一つ気になるのが「プログラム構造」です。例えば、「この関数に引数を追加したらどの部分に影響が出るのか?」や、「メソッド名を変更した際、どれだけの箇所を修正する必要があるのか?」といった悩みは、日常的に直面する問題です。もちろん、grepなどの検索ツールもありますが、それだけでは不十分だということを多くの技術者は痛感しているでしょう。
そこで今回は、「システム関連図」の代わりに「プログラム構造解析」に焦点を当て、Neo4jとgpt-4oを使ったPythonプログラムを作成してみようと思います。ここで思い浮かんだのが、かつて利用していたDoxygenです。最近は使っていませんでしたが、クラスやファイルの依存関係を高い精度で文書化でき、さらにGraphvizと連携してリッチなクラス図や依存関係図を生成できる優れたツールです。
今回は、Neo4j、gpt-4o、そしてDoxygenを組み合わせて、プログラムの構造を解析・可視化するプロセスに挑戦していきます。
Doxygenとは
Doxygenは、ソースコードのコメントをもとにドキュメントを自動生成するツールです。規定フォーマットに沿ったコメントを挿入することで、クラスや関数の関係性を視覚化したドキュメントを作成できます。特にGraphvizと組み合わせることで、関数やクラス間の依存関係を直感的に把握できる図を生成できるのが強力です。
詳しい設定方法については、@wakaba130さんの投稿を参考にさせていただきました。読みやすく、非常に参考になりましたので、感謝いたします。
今回は、「htmlファイル」と「xmlファイル」の2種類の出力を行います。htmlは依存関係を可視化するため、視覚的に正確な関連性を確認できるようにします。一方、xmlはこの情報を加工してgpt-4oと連携させるためのデータとして使用します。
Doxygen処理実行
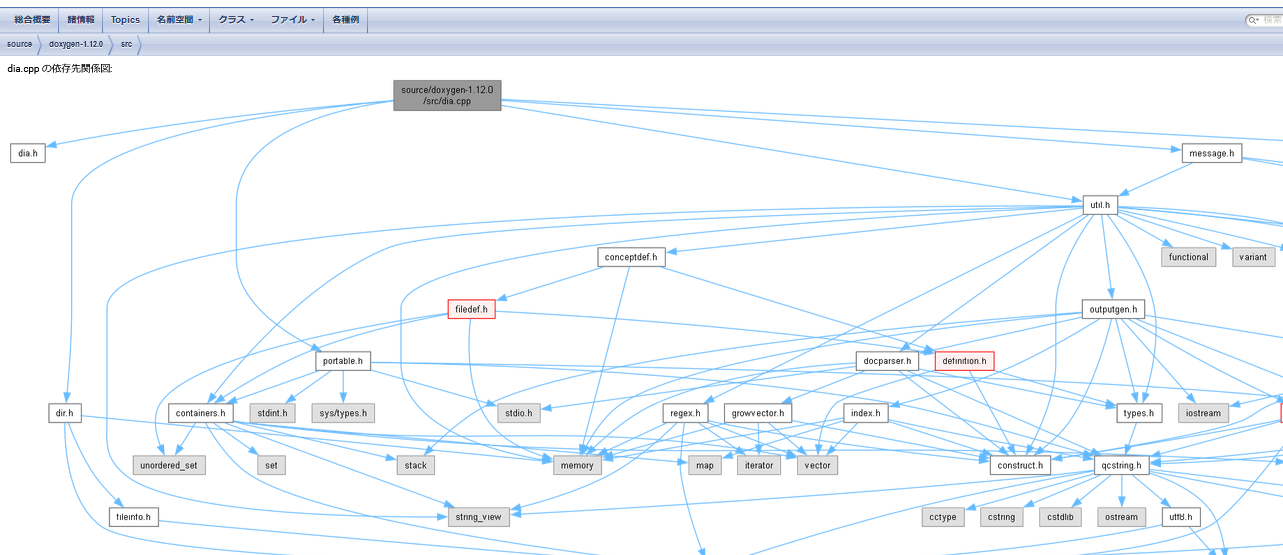
「木乃伊(ミイラ)取りが木乃伊になる」という表現を借りると、今回の試行はまさにそんな感じです。Doxygenを使い、実際にcppファイルを解析して依存関係を可視化しました。今回のターゲットはdia.cppというソースファイルで、Doxygenの解析結果として得られたhtmlファイルが以下の通りです。この図は、dia.cppの依存関係を視覚化したものです。
このhtmlファイルを確認することで、関数やクラス間の関係性が一目でわかり、プログラム構造の把握が容易になります。
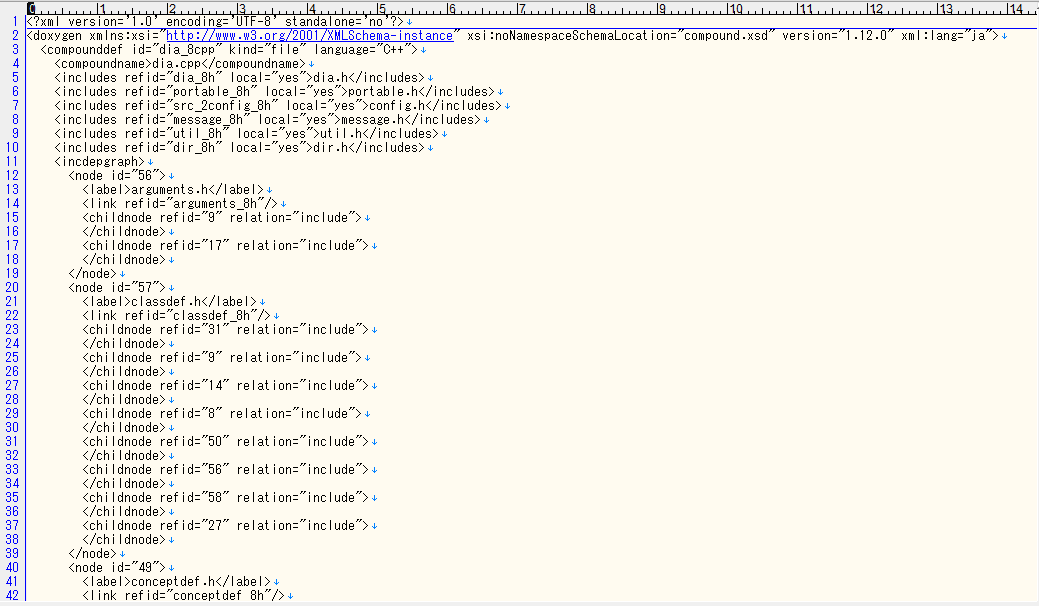
また、次に示すのは同じdia.cppに関連する「xmlファイル」のサンプルです。
このxmlデータを活用し、今後gpt-4oを用いたプログラム構造解析を進めていく予定です。xml形式の出力は、単にドキュメント生成にとどまらず、他のツールとの連携や自動処理にも適しています。
プログラム
処理概要
今回のプログラムの全体処理は以下のように整理できます。システム開発者がよく使用するデ・マルコ式のDFDにまとめてみました。
| 項目 | 概要説明 |
|---|---|
| Doxygen実行 | Doxygenを実行し、プログラムの依存関係を解析した「htmlファイル」と「xmlファイル」を生成します。 |
| 関係性抽出 | xmlファイルからファイル間の依存関係を解析し、txtファイルにその情報を登録します。 |
| Cypher形式ファイル生成 | 解析結果のtxtファイルをgpt-4oに入力し、Neo4jで使用するためのCypher形式のクエリを生成します。 |
| Neo4j登録 | 生成されたCypherクエリをNeo4jに登録し、グラフ図で依存関係を視覚化します。 |
プログラム(関係性抽出)
Doxygenから出力されるxmlファイルを解析し、ファイル間の依存関係を抽出するプロセスに着手しました。しかし、xmlファイルのフォーマットについて公式な詳細が見つからなかったため、実際のファイルを確認しながら帰納t的にでフォーマットを推測しました。
XMLファイル構造
例えば、次のようなxml構造がありました。
<incdepgraph>
<node id="56">
<label>arguments.h</label>
<link refid="arguments_8h"/>
<childnode refid="9" relation="include">
</childnode>
<childnode refid="17" relation="include">
</childnode>
</node>
<node id="57">
<label>classdef.h</label>
<link refid="classdef_8h"/>
<childnode refid="31" relation="include">
</childnode>
<childnode refid="9" relation="include">
</childnode>
<childnode refid="14" relation="include">
</childnode>
.....
このように、nodeタグにはidが設定され、ファイル名を示すラベルや、他のファイルとの依存関係を示すchildnodeタグが含まれています。refid属性によって関連ファイルが指定され、依存関係にあるファイルが子ノードとして表現されていることがわかります。次に、refid="9"に対応するノードを確認すると、以下のようにvectorというラベルが付いていることが確認できます。
<node id="9">
<label>vector</label>
</node>
これを基に、プログラムでファイル間の依存関係を抽出していきます。
Pythonコード
import glob
import os
import re
from dotenv import load_dotenv
import xml.etree.ElementTree as ET
from lxml import etree
load_dotenv()
files = glob.glob(os.path.join('C:/Users/ogiki/Desktop/doxygen/xml', "**/*.*"), recursive=True)
def execute():
with open("output.txt", "w", encoding="utf-8") as f:
for file in files:
# フルパスからファイル名を取得
filename_with_ext = os.path.basename(file) # ファイル名+拡張子
base, ext = os.path.splitext(filename_with_ext) # ファイル名と拡張子に分離
if ext == '.xml' and 'cpp' in base and not re.match(r'^\d{3}', base): # ファイル名の頭3文字が数字のものは除外
print(file)
tree = etree.parse(file)
root = tree.getroot() # ルート要素を取得
# 全てのノードをイテレーションしてタグ名を表示
for node in root.iter():
if node.tag == 'node':
# --- 子要素を取得 ---
children = node.getchildren()
for child in children:
# --- ラベルの取得 ---
if child.tag == 'label':
parent_label = child.text
# --- 子ノードの取得 ---
if child.tag == 'childnode':
refid = child.attrib
child_label = get_label(root, refid['refid'])
# 親子関係を標準出力
# print(f"{parent_label} → {child_label}")
f.write(f"{parent_label} → {child_label}\n")
def get_label(root, refid):
# 全てのノードをイテレーションしてタグ名を表示
for node in root.iter():
if node.tag == 'node':
if refid == node.attrib['id']:
# --- 子要素を取得 ---
children = node.getchildren()
for child in children:
if child.tag == 'label':
label = child.text
return label
return ''
if __name__ == "__main__":
execute()
このスクリプトでは、Doxygenが生成したXMLファイルを解析し、ファイル間の依存関係を抽出して、親ファイルと子ファイルの関係をテキストファイルに書き出します。
テキストファイルへの書き込み
今回は、関連情報を一旦テキストファイルに出力することにしました。この処理には、以前私が投稿したプログラムを再利用しています。
with open("output.txt", "w", encoding="utf-8") as f:
処理ファイルの選定
Doxygenによって生成されるXMLファイルの中には、「サマリ」を示すものが含まれており、これらはファイル名の先頭に3桁の数字が付いています。これらのファイルは処理対象外とし、さらにcppファイルのみを対象にしました。その結果、以下のような条件文になりました。
f ext == '.xml' and 'cpp' in base and not re.match(r'^\d{3}', base):
<node>タグでは子ノードの「id」を取得できますが、ノードの「ラベル」(名称)を直接取得することはできません。そこで、取得した子ノードのidを基に、再度rootから子ノードの「ラベル」を取得する処理を実装しました。
以下は、関数を呼び出している部分です。
child_label = get_label(root, refid['refid'])
以下、実際の関数です。
def get_label(root, refid):
# 全てのノードをイテレーションしてタグ名を表示
for node in root.iter():
if node.tag == 'node':
if refid == node.attrib['id']:
# --- 子要素を取得 ---
children = node.getchildren()
for child in children:
if child.tag == 'label':
label = child.text
return label
return ''
処理の結果は次のようになります。依存元と依存先をシンプルに「→」で示し、列挙しています。
source/doxygen-1.12.0/src/aliases.cpp → unordered_map
source/doxygen-1.12.0/src/aliases.cpp → cassert
source/doxygen-1.12.0/src/aliases.cpp → message.h
source/doxygen-1.12.0/src/aliases.cpp → aliases.h
source/doxygen-1.12.0/src/aliases.cpp → containers.h
source/doxygen-1.12.0/src/aliases.cpp → config.h
source/doxygen-1.12.0/src/aliases.cpp → regex.h
source/doxygen-1.12.0/src/aliases.cpp → textstream.h
source/doxygen-1.12.0/src/aliases.cpp → util.h
source/doxygen-1.12.0/src/aliases.cpp → debug.h
source/doxygen-1.12.0/src/aliases.cpp → stringutil.h
aliases.h → string
aliases.h → string_view
arguments.h → vector
arguments.h → qcstring.h
classdef.h → memory
classdef.h → vector
classdef.h → unordered_set
classdef.h → containers.h
classdef.h → definition.h
classdef.h → arguments.h
classdef.h → membergroup.h
classdef.h → configvalues.h
conceptdef.h → memory
conceptdef.h → definition.h
conceptdef.h → filedef.h
プログラム(Cypher形式ファイル生成 + Neo4jへの登録)
先ほどお話しした通り、このプログラムは以前私が投稿したものをほぼそのまま再利用しています。
from langchain_community.graphs import Neo4jGraph
from langchain.chat_models import ChatOpenAI
from langchain_experimental.graph_transformers import LLMGraphTransformer
from new4j_loader import load_py, load_fig, load_pdf, load_excel_fig_text, load_doxygen_text, split_text
# modelは「3.5-turbo」もしくは「4o」のみ。「4o-mini」はNG
# output.txt → neo4j用テキスト
llm = ChatOpenAI(model= "gpt-4o")
import os
# 自前のNeo4j
os.environ["NEO4J_URI"] = "bolt://localhost:7687"
os.environ["NEO4J_USERNAME"] = "neo4j"
os.environ["NEO4J_PASSWORD"] = "******"
graph = Neo4jGraph()
# 最初に全てクリアしておく
graph.query("MATCH (n) DETACH DELETE n;")
docs = load_doxygen_text()
tgt_chunks = split_text(docs)
llm_transformer = LLMGraphTransformer(llm=llm)
graph_documents = llm_transformer.convert_to_graph_documents(tgt_chunks)
graph.add_graph_documents(graph_documents)
特に大きな変更はありませんが、1つ注目すべき点は「自前のNeo4j」を使用していることです。Neo4j Auraは時間が経過したため、現在は利用できなくなっています。
import glob
from langchain_community.document_loaders import TextLoader
from langchain.docstore.document import Document
def load_doxygen_text(path: str= "C:/Users/ogiki/neo4j_pj/demodata/doxygen_output.txt") -> list:
doxygen_resources = []
for file in glob.glob(path):
print(file)
loader = TextLoader(file, encoding='utf-8')
pages = loader.load_and_split()
file_text = ''.join([x.page_content for x in pages])
doc = Document(page_content=file_text, metadata={'source': file})
doxygen_resources.append(doc)
return doxygen_resources
作成した「関係性抽出テキストファイル」をgpt-4oに渡すと、Cypher形式の文が返ってきます。それをNeo4jに登録する処理を行います。
結果
Neo4jでの結果を確認してみました。嬉しいことに、初めて動画を載せることができました♪ いろいろと大変でしたが、その甲斐がありました。

うまくいったようです。ただし、fileは緑色、Libraryは茶色、header fileは赤色ですが、***.hが緑色(file)になっているため、精度にはまだ改善の余地がありそうです。それでも、全体的に見て、関連性はしっかりと表現できているようでした。
おわりに
少しずつではありますが、「プログラム構造解析」が実現できました。この手法により、Cypher形式で属性やオブジェクトをSQLのように簡単に追加できる可能性が広がり、将来的に大いに期待できると思っています。