はじめに
streamlit上で分子の描画に困ることがあります。
今回利用するmols2gridというライブラリを利用するとsmiles記法による分子の構造式を描画することが可能です。
こちらにgithubリンクを張っています。
環境
- python3.9.16
- rdkit 2022.9.3
- streamlit 1.12
- mols2grid 1.1.0
コード中身
app.py
import mols2grid
import pandas as pd
import streamlit as st
import streamlit.components.v1 as components
#アプリタイトル
st.title("分子構造描画アプリ")
# smilesのカラムを含むデータフレームを作成。今回はQM9のデータの一部を利用
df_qm9 = pd.read_excel("qm9.xlsx")
# streamlit上に表示
st.write(df_qm9)
# mols2gridを利用して構造式をstreamlit上に描画
raw_html = mols2grid.display(df_qm9, smiles_col="smiles")._repr_html_()
components.html(raw_html, width=900, height=900, scrolling=True)
結果
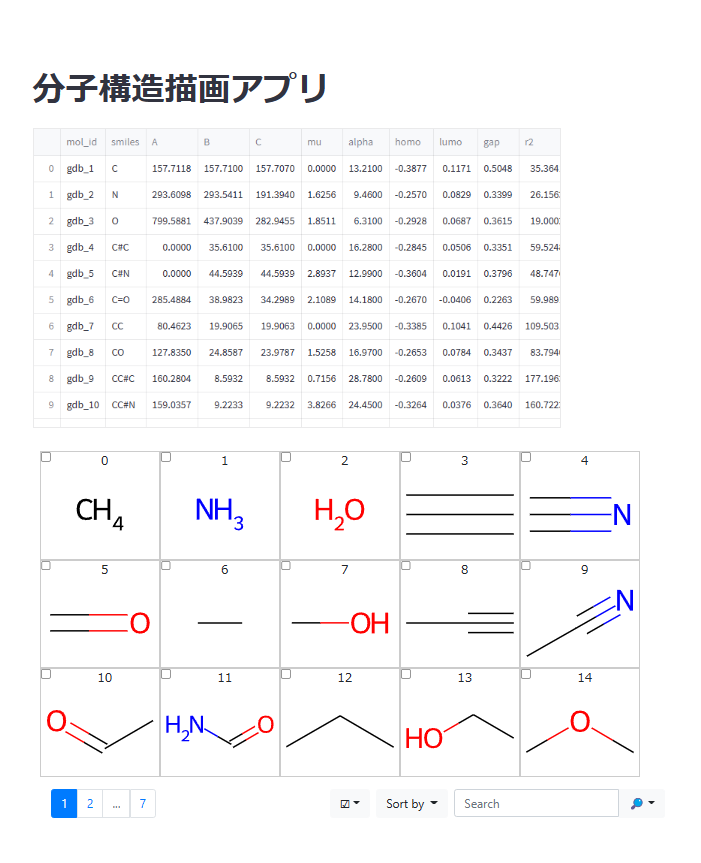
このような画面が出力されます。
右下のSort byをクリックすることで、任意のカラムの情報を用いてソートすることができます。
今回はpandasのデータフレームを使いましたが、そのほかにもrdkitのmolオブジェクトやsdfファイルを読み込むことが可能です。
import mols2grid
# sdfファイルを読み込む場合
mols2grid.display("molecules.sdf")
# molオブジェクトを読み込む場合
mols2grid.display(mols)
本記事では割愛しますが、そのほかに3D構造を表示したり、popupを表示させたりすることも可能です。
この辺りはdocumentに詳しく書かれているのでそちらを参照してください。