はじめに
- 急遽小さく立ち上がるAI/ML案件のための、個別にサクッと低コスト、でも最低限必要な運用機能は備えた開発環境の構成とその運用ルールを考えてみました。
- Not リッチに機能テンコ盛りでイケイケで本番稼働な大規模自社サービス用の機械学習基盤
- 実案件やってみた反省を踏まえて考えましたが、これから実際に運用してみて、随時ブラッシュアップしていくことになると思います。
今回のスコープ
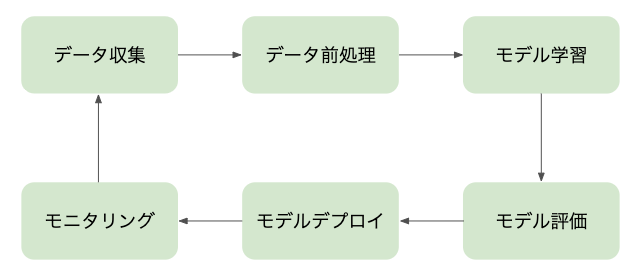
- 一般的な機械学習ワークフロー全体
-> 今回は、データ収集 ~ モデル評価あたりをスコープとしています。
困っていたこと
〜個別所有のローカル開発マシンだけで無邪気に実験していると、後から困ることあるある〜
- 学習でGPU使い切っちゃったから暇になったなぁ。(暇じゃない)
- そういえば、このときのこのデータって、どうやって作ったんでしたっけ?
- そういえば、このときのこのモデルって、どうやって作ったんでしたっけ?
- そういえば、このときのこの検証結果って、どういう条件で検証したんでしたっけ?
- あの検証結果どこでしたっけ?
開発環境に最低限望むこと
- 学習・評価が並行で実行できること
- 特にハイパーパラメータチューニングのときに必要
- 評価も、なんやかんや色んな条件で実施する
-
再現性を担保してくれること
- 前処理
- 元データ <-> 前処理スクリプト <-> 加工済データ
- 学習
- 加工済みデータ(学習用) <-> 学習スクリプト、ハイパーパラメータ <-> モデル
- 評価
- 加工済みデータ(評価用) <-> 評価コード <-> モデル <-> 評価結果(サマリ表、画像出力結果など)
- 前処理
- 低コストであること
- 費用が安い
-
データサイエンティスト/データエンジニアの作業コストが低い
- -> 例えば、極力ノートブックで。(画像を扱う案件とかで、定性評価のためにノートブックに画像を出力して確認したかったりするので)
- 自由と統制のバランスをいい感じに!(難しい)
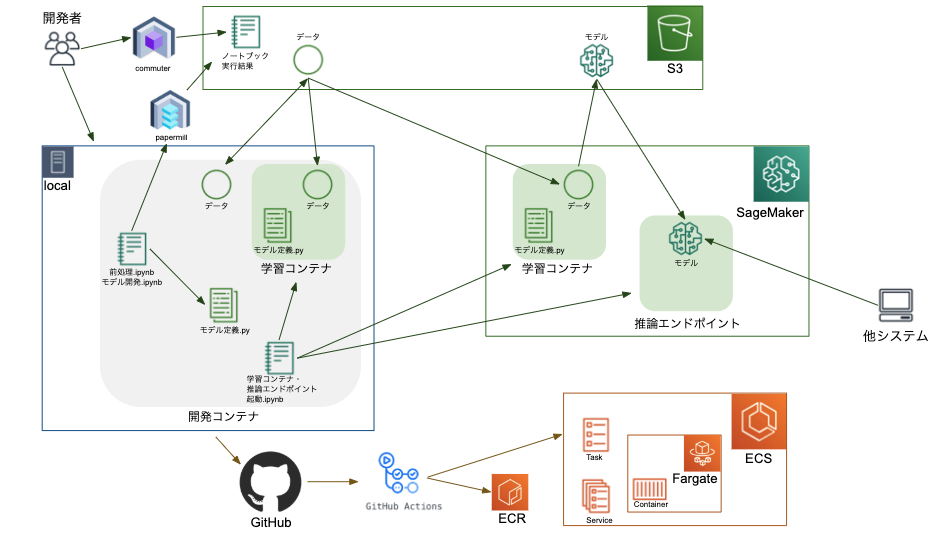
構成概要
- データはS3に集める。ローカルにしかない、がないように。
- データ前処理とかモデル評価のノートブックは、papermillを使って並行で実行したりS3に格納したりするようにする -> commuterで結果確認するようにする
- ローカルマシンで開発しつつ、モデルをガンガン学習させる段階になったら、SageMakerを使って学習を並行で処理 + 結果管理するようにする
- データとソースの整合性が取れていることを確認するため、CIも回す。GitHub Actions + Fargate。(Fargate上コンテナからSageMaker学習コンテナを起動したり。)
環境構築手順
※ソース全量は別途GitHubに上げておきます。あくまで説明用に一部抜粋して記載します。
S3バケット作成
- S3バケット作成
- バージョニングを有効化
-
フォルダ構成は最終的に下記のようにする(アップロード時に作成する)
- Cookiecutter Data Scienceを参考にしています。
- rawは受領したデータとかをそのまま格納して、触らない。
- interimとprocessedは必ずrawから加工されたデータを格納する。
- reportsにノートブック実行結果を格納する。ノートブックごとにディレクトリを作成する。
フォルダ構成
├── data
│ ├── raw <- The original, immutable data dump.
│ │ └── dataType_date
│ │ ├── image
│ │ └── csv
│ ├── interim <- Intermediate data that has been transformed.
│ │ └── ${rawDir}_howToProcess
│ │ ├── image
│ │ └── csv
│ ├── processed <- The final, canonical data sets for modeling.
│ │ └── ${rawDir}_howToProcess
│ │ ├── image
│ │ └── csv
│ └── external <- Data from third party sources.
│ └── dataType_date
│ ├── image
│ └── csv
├── reports
│ └── notebookName
IAMユーザ作成
- S3の権限を付与
- ユーザごとに、アクセスできるバケットを制限
IAMロール作成
- S3とSageMakerの権限を付与
コンテナ環境構築
- Dockerfile作成、build、run
- フォルダ構成は『【Kaggleのフォルダ構成や管理方法】タイタニック用のGitHubリポジトリを公開しました』を参考に作成しました。
Dockerfile
FROM 520713654638.dkr.ecr.ap-northeast-1.amazonaws.com/sagemaker-pytorch:1.1.0-cpu-py3
# apt install
RUN apt-get update && \
apt-get install -y \
curl \
git \
vim \
groff-base
# pip install
COPY requirements.txt /tmp/
RUN pip install --upgrade pip && \
pip install -r /tmp/requirements.txt
# install docker
RUN curl https://get.docker.com | sh
# make dir
RUN mkdir -p /opt/config \
/opt/data/raw \
/opt/data/interim \
/opt/data/processed \
/opt/data/external \
/opt/features \
/opt/logs \
/opt/models \
/opt/notebooks \
/opt/reports \
/opt/scripts \
/opt/utils
# cd
WORKDIR /opt
# expose
EXPOSE 8888
CI環境構築
- 下記を参考に、GitHub Action・ECR・ECSの設定
環境利用手順
データ収集
- データ収集ノートブック作成
- 上記で作成したrawフォルダ(s3)に受領したデータとかを格納する
- 今回はサンプルとして、下記コードにてcifar10のデータを格納する
-
ノートブックの書き方ルール(共通)
* parameters
* パラメータを記載。papermillでコマンドラインから渡すことを想定。
* -> check existence of OUTPUT_DATA_PATH
* すでにS3にアウトプットがある場合はexit- -> download from INPUT_DATA_PATH
- ローカルにインプットがなければ、S3からデータをダウンロード
* -> run - メインの処理を記載
* -> upload to OUTPUT_DATA_PATH - S3にデータをアップロード
* -> notify slack - Slackに通知
- ローカルにインプットがなければ、S3からデータをダウンロード
- -> download from INPUT_DATA_PATH
cifar10_collect_data.ipynb
# parameters
S3_BUCKET_NAME = ''
# INPUT_DATA_PATH_LOCAL = ''
# INPUT_DATA_PATH_S3 = ''
OUTPUT_DATA_PATH_LOCAL = '../data/raw/cifar10'
OUTPUT_DATA_PATH_S3 = 'data/raw/cifar10'
# check existence of OUTPUT_DATA_PATH
import boto3
import sys
s3 = boto3.resource('s3')
bucket = s3.Bucket(S3_BUCKET_NAME)
key = OUTPUT_DATA_PATH_S3
objs = list(bucket.objects.filter(Prefix=key))
if len(objs) > 0 and (objs[0].key == key or objs[0].key == key + '/'):
print("OUTPUT FILE Exists!")
sys.exit(0)
# download from INPUT_DATA_PATH
import subprocess
import os.path
if os.path.exists(INPUT_DATA_PATH_LOCAL) == False:
cmd = 'aws s3 cp s3://{0}/{1} {2} --recursive'.format(S3_BUCKET_NAME, INPUT_DATA_PATH_S3, INPUT_DATA_PATH_LOCAL)
subprocess.call(cmd.split())
# run
# collect data
import torch
import torchvision
import torchvision.transforms as transforms
trainset = torchvision.datasets.CIFAR10(root=OUTPUT_DATA_PATH_LOCAL, train=True, download=True)
testset = torchvision.datasets.CIFAR10(root=OUTPUT_DATA_PATH_LOCAL, train=False,download=True)
# upload to OUTPUT_DATA_PATH
import subprocess
cmd = 'aws s3 cp {0} s3://{1}/{2} --recursive'.format(OUTPUT_DATA_PATH_LOCAL, S3_BUCKET_NAME, OUTPUT_DATA_PATH_S3)
subprocess.call(cmd.split())
# notify slack
import requests
def slack_notify(msg = NOTEBOOK_NAME + 'has completed'):
slack_user_id = SLACK_USER_ID
slack_webhook_url = SLACK_WEBHOOK_URL
requests.post(slack_webhook_url, json={"text":f"<@{slack_user_id}> {msg}"})
- データ収集ノートブック実行
コマンド
$ papermill cifar10_acquisition.ipynb s3://${S3_BUCKET_NAME}/reports/cifar10_collect_data/cifar10_collect_data_`date "+%Y%m%d%H%M%S"`.ipynb
データ前処理
- データ前処理ノートブック作成
- 今回は略
- データ前処理ノートブック実行
- 今回は略
モデル学習
- モデル学習ノートブック作成
cifar10_train_model
# parameters
S3_BUCKET_NAME = 'sagemaker-sample-bucket01'
INPUT_DATA_PATH_LOCAL = '../data/raw/cifar10'
INPUT_DATA_PATH_S3 = 'data/raw/cifar10'
OUTPUT_DATA_PATH_LOCAL = '../data/processed/cifar10'
OUTPUT_DATA_PATH_S3 = 'data/processed/cifar10'
# check existence of OUTPUT_DATA_PATH
import boto3
import sys
s3 = boto3.resource('s3')
bucket = s3.Bucket(S3_BUCKET_NAME)
key = OUTPUT_DATA_PATH_S3
objs = list(bucket.objects.filter(Prefix=key))
if len(objs) > 0 and (objs[0].key == key or objs[0].key == key + '/'):
print("OUTPUT FILE Exists!")
sys.exit(0)
# download from INPUT_DATA_PATH
import subprocess
import os.path
if os.path.exists(INPUT_DATA_PATH_LOCAL) == False:
cmd = 'aws s3 cp s3://{0}/{1} {2} --recursive'.format(S3_BUCKET_NAME, INPUT_DATA_PATH_S3, INPUT_DATA_PATH_LOCAL)
subprocess.call(cmd.split())
# run
# train model
import sagemaker
import subprocess
from sagemaker.pytorch import PyTorch
sagemaker_session = sagemaker.Session()
role = sagemaker.get_execution_role() # ex. arn:aws:iam::xxxxxxxxxxxx:role/service-role/...
instance_type = 'local'
# instance_type = 'ml.m5.large'
# estimator
hyper_param = {
'workers': 2,
'epochs':2,
'batch_size': 4,
'lr': 0.001,
'momentum': 0.9,
}
cifar10_estimator = PyTorch(entry_point='../models/cifar10_sagemaker.py',
hyperparameters=hyper_param,
role=role,
framework_version='1.1.0',
train_instance_count=1,
train_instance_type=instance_type)
# fit
cifar10_estimator.fit(
inputs='s3://' + S3_BUCKET_NAME + '/' + INPUT_DATA_PATH_S3,
)
# upload to OUTPUT_DATA_PATH
# import subprocess
# cmd = 'aws s3 cp {0} s3://{1}/{2} --recursive'.format(OUTPUT_DATA_PATH_LOCAL, S3_BUCKET_NAME, OUTPUT_DATA_PATH_S3)
# subprocess.call(cmd.split())
# notify slack
import requests
def slack_notify(msg = NOTEBOOK_NAME + 'has completed'):
slack_user_id = SLACK_USER_ID
slack_webhook_url = SLACK_WEBHOOK_URL
requests.post(slack_webhook_url, json={"text":f"<@{slack_user_id}> {msg}"})
- モデル学習ノートブック実行
コマンド実行
$ papermill cifar10_train_model.ipynb s3://${S3_BUCKET_NAME}/reports/cifar10_train_model/cifar10_train_model_model_`date "+%Y%m%d%H%M%S"`.ipynb
モデル評価
- モデル評価ノートブック作成
- 今回は略
- モデル評価ノートブック実行
- 今回は略
CI実行
- developブランチが更新されると、CIが走るようになっているので、結果を見て(S3に上がったノートブックをcommuterで + SageMakerの管理画面から)、masterにマージするようにする
おわりに
- 随時修正していきます。re:Inventで発表された機能を取り入れたり、AWSに限らず他のサービスもどんどん試していきたいと思います。