はじめに
新型コロナウイルス新規感染者数の報道を見るたびに「年齢層の内訳をもっと詳しく教えてほしいなあ」と思っていたら、東京都が陽性患者のデータを公開していることを今更ながら知りました。
ここでは、Pythonおよびpandas, seaborn, Matplotlibを使って東京都が公開しているデータを分析・可視化する方法を紹介します。
本記事の主旨は「今後はこうなる」「こういう対策をすべし」という予測や提言ではなく、「こうすると簡単にデータを可視化できるからみんなもやってみてね」ということです。自分で試してみると理解が深まるので、みなさんも是非やってみてください。
なお、グラフのレイアウトや軸の書式などの細部にこだわるとMatplotlibの面倒な処理が必要なので、ここでは深追いしません(最後に少し触れる程度)。広く公開するための見栄えの良いグラフを作るというよりも、自分自身でデータを確認して傾向をつかむための可視化をゴールとします。
サンプルコードはGitHubにもおいてあります。Jupyter Notebook(.ipynb)のほうが見やすい部分もあるので、あわせてご参照ください。
データの概要
東京都
東京都の陽性患者データは以下で公開されている。
東京都の新型コロナウイルス感染症対策サイトのオープンデータを入手のリンクから辿り着ける。
履歴を見ると、平日の10〜15時くらいに更新されている模様。
その他の都道府県
東京都の対策サイトをフォークしたサイトの一覧が以下にまとめられている。
東京都のようにオープンデータへのリンクがあるサイトもある。以下は北海道と神奈川県の例。
サイト上にリンクがない場合も、データ自体はどこかで公開されているはずなので探せば見つかるかもしれない。
以降のサンプルコードでは東京都のデータを使う。他の都道府県のデータは項目などが異なる場合があるが、基本的な扱いは同じ。
その他のデータ
そのほか、新型コロナウイルス関連のデータとしては厚生労働省が発表している国内のPCR検査実施人数や陽性者数、入院者数、死者数などの集計データがある。
ライブラリのバージョン
以降のサンプルコードでの各ライブラリおよびPython本体のバージョンは以下の通り。バージョンが異なると挙動が異なる可能性があるので注意。
import math
import sys
import pandas as pd
import matplotlib as mpl
import matplotlib.pyplot as plt
import seaborn as sns
print(pd.__version__)
# 1.0.5
print(mpl.__version__)
# 3.3.0
print(sns.__version__)
# 0.10.1
print(sys.version)
# 3.8.5 (default, Jul 21 2020, 10:48:26)
# [Clang 11.0.3 (clang-1103.0.32.62)]
データの確認と前処理
pd.read_csv()にダウンロードしたCSVファイルへのパスを指定してDataFrameとして読み込む。2020年7月31日までのデータを例として使う。
df = pd.read_csv('data/130001_tokyo_covid19_patients_20200731.csv')
pd.read_csv()の引数にはURLを直接指定することもできるが、試行錯誤する段階では何度も無駄にアクセスすることになってしまうのでローカルにダウンロードするほうが無難。
# df = pd.read_csv('https://stopcovid19.metro.tokyo.lg.jp/data/130001_tokyo_covid19_patients.csv')
行数・列数および先頭・末尾のデータは以下の通り。
print(df.shape)
# (12691, 16)
print(df.head())
# No 全国地方公共団体コード 都道府県名 市区町村名 公表_年月日 曜日 発症_年月日 患者_居住地 患者_年代 患者_性別 \
# 0 1 130001 東京都 NaN 2020-01-24 金 NaN 湖北省武漢市 40代 男性
# 1 2 130001 東京都 NaN 2020-01-25 土 NaN 湖北省武漢市 30代 女性
# 2 3 130001 東京都 NaN 2020-01-30 木 NaN 湖南省長沙市 30代 女性
# 3 4 130001 東京都 NaN 2020-02-13 木 NaN 都内 70代 男性
# 4 5 130001 東京都 NaN 2020-02-14 金 NaN 都内 50代 女性
#
# 患者_属性 患者_状態 患者_症状 患者_渡航歴の有無フラグ 備考 退院済フラグ
# 0 NaN NaN NaN NaN NaN 1.0
# 1 NaN NaN NaN NaN NaN 1.0
# 2 NaN NaN NaN NaN NaN 1.0
# 3 NaN NaN NaN NaN NaN 1.0
# 4 NaN NaN NaN NaN NaN 1.0
print(df.tail())
# No 全国地方公共団体コード 都道府県名 市区町村名 公表_年月日 曜日 発症_年月日 患者_居住地 患者_年代 \
# 12686 12532 130001 東京都 NaN 2020-07-31 金 NaN NaN 70代
# 12687 12558 130001 東京都 NaN 2020-07-31 金 NaN NaN 70代
# 12688 12563 130001 東京都 NaN 2020-07-31 金 NaN NaN 70代
# 12689 12144 130001 東京都 NaN 2020-07-31 金 NaN NaN 80代
# 12690 12517 130001 東京都 NaN 2020-07-31 金 NaN NaN 80代
#
# 患者_性別 患者_属性 患者_状態 患者_症状 患者_渡航歴の有無フラグ 備考 退院済フラグ
# 12686 男性 NaN NaN NaN NaN NaN NaN
# 12687 男性 NaN NaN NaN NaN NaN NaN
# 12688 男性 NaN NaN NaN NaN NaN NaN
# 12689 女性 NaN NaN NaN NaN NaN NaN
# 12690 男性 NaN NaN NaN NaN NaN NaN
本データのようなカテゴリカル・データが主である場合、count()やnunique(), unique(), value_counts()などのメソッドを使うと概要がつかみやすい。
- pandas.DataFrame.count — pandas 1.1.0 documentation
- pandas.DataFrame.nunique — pandas 1.1.0 documentation
- pandas.Series.unique — pandas 1.1.0 documentation
- pandas.Series.value_counts — pandas 1.1.0 documentation
- pandasでユニークな要素の個数、頻度(出現回数)をカウント
count()は欠損値NaNではない要素の数を返す。プライバシー保護のためか、市区町村名や症状、属性などの細かい情報は公開されていない(データがない)ことが分かる。
print(df.count())
# No 12691
# 全国地方公共団体コード 12691
# 都道府県名 12691
# 市区町村名 0
# 公表_年月日 12691
# 曜日 12691
# 発症_年月日 0
# 患者_居住地 12228
# 患者_年代 12691
# 患者_性別 12691
# 患者_属性 0
# 患者_状態 0
# 患者_症状 0
# 患者_渡航歴の有無フラグ 0
# 備考 0
# 退院済フラグ 7186
# dtype: int64
nunique()はデータの種類の数を返す。東京都のデータなので全国地方公共団体コードや都道府県名はすべて同じ。
print(df.nunique())
# No 12691
# 全国地方公共団体コード 1
# 都道府県名 1
# 市区町村名 0
# 公表_年月日 164
# 曜日 7
# 発症_年月日 0
# 患者_居住地 8
# 患者_年代 13
# 患者_性別 5
# 患者_属性 0
# 患者_状態 0
# 患者_症状 0
# 患者_渡航歴の有無フラグ 0
# 備考 0
# 退院済フラグ 1
# dtype: int64
それぞれの列(= Series)に対してはunique(), value_counts()でユニークな要素およびその個数(出現頻度)が確認できる。
print(df['患者_居住地'].unique())
# ['湖北省武漢市' '湖南省長沙市' '都内' '都外' '―' '調査中' '-' "'-" nan]
print(df['患者_居住地'].value_counts(dropna=False))
# 都内 11271
# 都外 531
# NaN 463
# ― 336
# 調査中 85
# 湖北省武漢市 2
# 湖南省長沙市 1
# '- 1
# - 1
# Name: 患者_居住地, dtype: int64
print(df['患者_性別'].unique())
# ['男性' '女性' "'-" '―' '不明']
print(df['患者_性別'].value_counts())
# 男性 7550
# 女性 5132
# '- 7
# 不明 1
# ― 1
# Name: 患者_性別, dtype: int64
今回は分析対象を公表年月日、患者の年代、退院済フラグに絞る。便宜上、rename()で列名を変更しておく。
df = df[['公表_年月日', '患者_年代', '退院済フラグ']].copy()
df.rename(columns={'公表_年月日': 'date_str', '患者_年代': 'age_org', '退院済フラグ': 'discharged'},
inplace=True)
print(df)
# date_str age_org discharged
# 0 2020-01-24 40代 1.0
# 1 2020-01-25 30代 1.0
# 2 2020-01-30 30代 1.0
# 3 2020-02-13 70代 1.0
# 4 2020-02-14 50代 1.0
# ... ... ... ...
# 12686 2020-07-31 70代 NaN
# 12687 2020-07-31 70代 NaN
# 12688 2020-07-31 70代 NaN
# 12689 2020-07-31 80代 NaN
# 12690 2020-07-31 80代 NaN
#
# [12691 rows x 3 columns]
ここでcopy()を使っているのはSettingWithCopyWarningを防ぐため。今回の場合、データを更新するわけではないので放っておいても問題はない。
年代の列を見てみると、不明や'-といったデータが含まれている。
print(df['age_org'].unique())
# ['40代' '30代' '70代' '50代' '60代' '80代' '20代' '10歳未満' '90代' '10代' '100歳以上'
# '不明' "'-"]
print(df['age_org'].value_counts())
# 20代 4166
# 30代 2714
# 40代 1741
# 50代 1362
# 60代 832
# 70代 713
# 80代 455
# 10代 281
# 90代 214
# 10歳未満 200
# 不明 6
# 100歳以上 5
# '- 2
# Name: age_org, dtype: int64
数が少ないので、ここでは除外してしまうことにする。
df = df[~df['age_org'].isin(['不明', "'-"])]
print(df)
# date_str age_org discharged
# 0 2020-01-24 40代 1.0
# 1 2020-01-25 30代 1.0
# 2 2020-01-30 30代 1.0
# 3 2020-02-13 70代 1.0
# 4 2020-02-14 50代 1.0
# ... ... ... ...
# 12686 2020-07-31 70代 NaN
# 12687 2020-07-31 70代 NaN
# 12688 2020-07-31 70代 NaN
# 12689 2020-07-31 80代 NaN
# 12690 2020-07-31 80代 NaN
#
# [12683 rows x 3 columns]
print(df['age_org'].unique())
# ['40代' '30代' '70代' '50代' '60代' '80代' '20代' '10歳未満' '90代' '10代' '100歳以上']
年代の区分が細かいので、もう少し粗くする。右辺全体を括弧()で囲んでいるのは途中で改行するため。
df['age'] = (
df['age_org'].replace(['10歳未満', '10代'], '0-19')
.replace(['20代', '30代'], '20-39')
.replace(['40代', '50代'], '40-59')
.replace(['60代', '70代', '80代', '90代', '100歳以上'], '60-')
)
print(df['age'].unique())
# ['40-59' '20-39' '60-' '0-19']
print(df['age'].value_counts())
# 20-39 6880
# 40-59 3103
# 60- 2219
# 0-19 481
# Name: age, dtype: int64
日時(公表年月日)の列date_strは文字列。今後の処理のため、datetime64[ns]型に変換した列dateを追加しておく。
df['date'] = pd.to_datetime(df['date_str'])
print(df.dtypes)
# date_str object
# age_org object
# discharged float64
# age object
# date datetime64[ns]
# dtype: object
前処理はここまで。ここからは実際にデータを分析・可視化する例を示す。
年代別の新規陽性患者数の推移
ここでは年代別の新規陽性患者数の推移を見る。新規陽性患者数の総数についてはMatplotlibで処理する例として最後に述べる。
積み上げ棒グラフ
pd.crosstab()で日時(公表年月日)と年代のクロス集計を行う。
df_ct = pd.crosstab(df['date'], df['age'])
print(df_ct)
# age 0-19 20-39 40-59 60-
# date
# 2020-01-24 0 0 1 0
# 2020-01-25 0 1 0 0
# 2020-01-30 0 1 0 0
# 2020-02-13 0 0 0 1
# 2020-02-14 0 0 1 1
# ... ... ... ... ...
# 2020-07-27 5 79 34 13
# 2020-07-28 13 168 65 20
# 2020-07-29 9 160 56 25
# 2020-07-30 11 236 83 37
# 2020-07-31 10 332 82 39
#
# [164 rows x 4 columns]
print(type(df_ct.index))
# <class 'pandas.core.indexes.datetimes.DatetimeIndex'>
datetime64[ns]型に変換した列が新たにインデックスとなり、DatetimeIndexとして扱われる。出力が同じでも文字列型の日時を指定した場合はDatetimeIndexにならないので注意。
resample()で週ごとに集計する。resample()はDatetimeIndexでないと実行できない。
df_ct_week = df_ct.resample('W', label='left').sum()
print(df_ct_week)
# age 0-19 20-39 40-59 60-
# date
# 2020-01-19 0 1 1 0
# 2020-01-26 0 1 0 0
# 2020-02-02 0 0 0 0
# 2020-02-09 0 2 5 9
# 2020-02-16 0 1 3 6
# 2020-02-23 0 2 3 5
# 2020-03-01 2 5 9 9
# 2020-03-08 0 5 10 11
# 2020-03-15 0 10 27 12
# 2020-03-22 7 100 88 102
# 2020-03-29 16 244 198 148
# 2020-04-05 21 421 369 271
# 2020-04-12 30 350 375 280
# 2020-04-19 32 286 267 264
# 2020-04-26 29 216 165 260
# 2020-05-03 7 105 69 120
# 2020-05-10 2 46 16 46
# 2020-05-17 3 22 10 15
# 2020-05-24 4 43 16 21
# 2020-05-31 2 89 34 22
# 2020-06-07 5 113 17 26
# 2020-06-14 6 177 29 28
# 2020-06-21 10 236 65 23
# 2020-06-28 34 460 107 51
# 2020-07-05 79 824 191 68
# 2020-07-12 66 1006 295 117
# 2020-07-19 78 1140 414 171
# 2020-07-26 48 975 320 134
plot()で可視化。簡単に積み上げ棒グラフが作成できる。
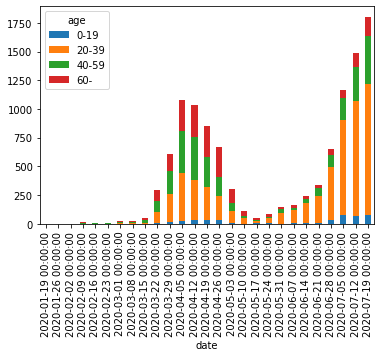
df_ct_week[:-1].plot.bar(stacked=True)
[:-1]で最終行(最終週)のデータを除外している。今回のデータでは最終週は土曜日(2020年8月1日)の分が含まれておらず他の週と比較するのは適切でないため除外した。
Jupyter Notebookではグラフが出力セルに表示される。画像ファイルとして保存したい場合はplt.savefig()を使う。Jupyter Notebookの出力を右クリックで保存することもできる。
plt.figure()
df_ct_week[:-1].plot.bar(stacked=True)
plt.savefig('image/bar_chart.png', bbox_inches='tight')
plt.close('all')
発生条件が不明だが、保存時にX軸ラベルが切れてしまうという不具合があった。以下を参考にbbox_inches='tight'としたら解消した。
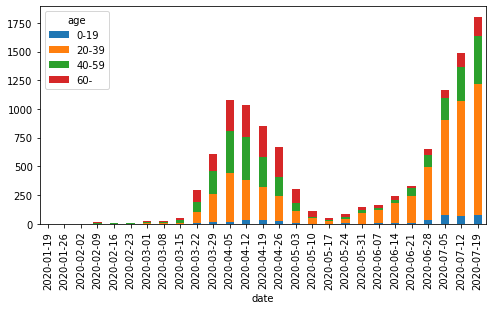
上の例のように、そのまま棒グラフを作成すると、X軸ラベルに時刻が表示されてしまう。最も簡単な解決策はインデックスを任意のフォーマットの文字列に変換してしまうこと。
df_ct_week_str = df_ct_week.copy()
df_ct_week_str.index = df_ct_week_str.index.strftime('%Y-%m-%d')
df_ct_week_str[:-1].plot.bar(stacked=True, figsize=(8, 4))
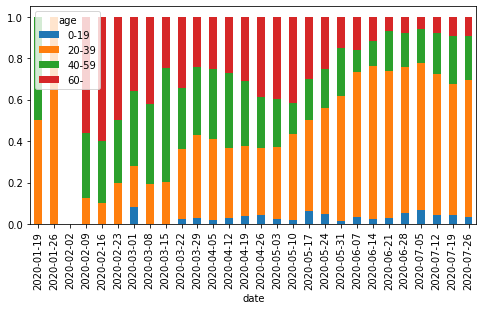
全体を規格化して年代の比率の推移を見る。Tは転置(行と列の入れ替え)。転置して合計値で割り算、再度転置してもとに戻すことで規格化できる。
6月以降は若年者(20〜30代)が大部分を締めているが、直近は中高齢者(40代以降)の割合も増えつつある。
df_ct_week_str_norm = (df_ct_week_str.T / df_ct_week_str.sum(axis=1)).T
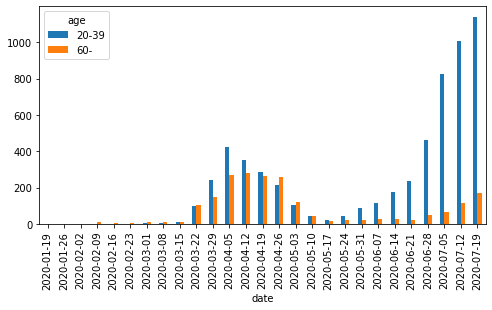
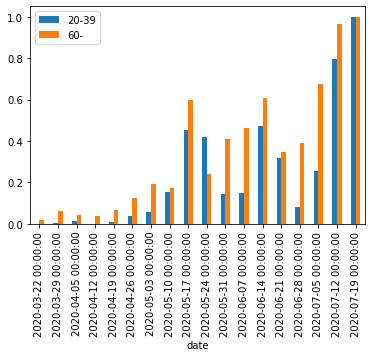
若年者(20〜30代)と高齢者(60代以降)の推移は以下の通り。高齢者の絶対数も3月末のレベル程度まで増えている。
df_ct_week_str[:-1][['20-39', '60-']].plot.bar(figsize=(8, 4))
折れ線グラフ(前週比)
感染拡大の勢いを見るため、前週比を算出する。
shift()でデータをずらして割ればよい。
df_week_ratio = df_ct_week / df_ct_week.shift()
print(df_week_ratio)
# age 0-19 20-39 40-59 60-
# date
# 2020-01-19 NaN NaN NaN NaN
# 2020-01-26 NaN 1.000000 0.000000 NaN
# 2020-02-02 NaN 0.000000 NaN NaN
# 2020-02-09 NaN inf inf inf
# 2020-02-16 NaN 0.500000 0.600000 0.666667
# 2020-02-23 NaN 2.000000 1.000000 0.833333
# 2020-03-01 inf 2.500000 3.000000 1.800000
# 2020-03-08 0.000000 1.000000 1.111111 1.222222
# 2020-03-15 NaN 2.000000 2.700000 1.090909
# 2020-03-22 inf 10.000000 3.259259 8.500000
# 2020-03-29 2.285714 2.440000 2.250000 1.450980
# 2020-04-05 1.312500 1.725410 1.863636 1.831081
# 2020-04-12 1.428571 0.831354 1.016260 1.033210
# 2020-04-19 1.066667 0.817143 0.712000 0.942857
# 2020-04-26 0.906250 0.755245 0.617978 0.984848
# 2020-05-03 0.241379 0.486111 0.418182 0.461538
# 2020-05-10 0.285714 0.438095 0.231884 0.383333
# 2020-05-17 1.500000 0.478261 0.625000 0.326087
# 2020-05-24 1.333333 1.954545 1.600000 1.400000
# 2020-05-31 0.500000 2.069767 2.125000 1.047619
# 2020-06-07 2.500000 1.269663 0.500000 1.181818
# 2020-06-14 1.200000 1.566372 1.705882 1.076923
# 2020-06-21 1.666667 1.333333 2.241379 0.821429
# 2020-06-28 3.400000 1.949153 1.646154 2.217391
# 2020-07-05 2.323529 1.791304 1.785047 1.333333
# 2020-07-12 0.835443 1.220874 1.544503 1.720588
# 2020-07-19 1.181818 1.133201 1.403390 1.461538
# 2020-07-26 0.615385 0.855263 0.772947 0.783626
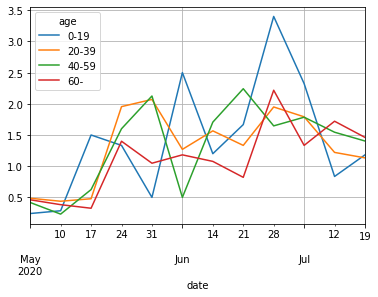
df_week_ratio['2020-05-03':'2020-07-25'].plot(grid=True)
7月に入って前週比は各年代とも減少傾向にある。
なお、棒グラフとは異なり、plot()(またはplot.line())で折れ線グラフを作成する場合は上の例のようにX軸の日時データが適当にフォーマットされる。後述のように日時データの内容によってはフォーマットされない場合もあるので注意。
ヒートマップ
年代別の新規陽性患者数の推移を把握するための別のアプローチとして、ヒートマップを作成する。
ここでは細かい年代区分をそのまま使う。積み上げ棒グラフの例と同じくpd.crosstab()でクロス集計。resample()を使わないので文字列型の日時date_strを指定している。横軸を日時にするためTで転置し、縦軸の下側を若くするため[::-1]で転置後の行の並びを反転している。
df['age_detail'] = df['age_org'].replace(
{'10歳未満': '0-9', '10代': '10-19', '20代': '20-29', '30代': '30-39', '40代': '40-49', '50代': '50-59',
'60代': '60-69', '70代': '70-79', '80代': '80-89', '90代': '90-', '100歳以上': '90-'}
)
df_ct_hm = pd.crosstab(df['date_str'], df['age_detail']).T[::-1]
ヒートマップを作成するにはseabornの関数heatmap()が便利。
plt.figure(figsize=(15, 5))
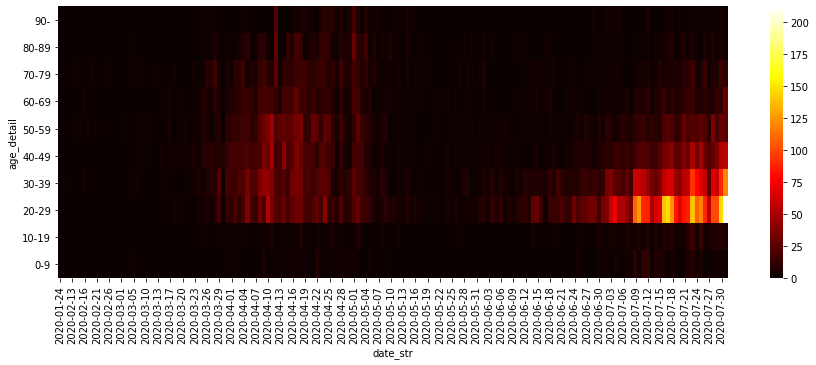
sns.heatmap(df_ct_hm, cmap='hot')
6月以降、徐々に高齢者に感染が広がっているのが確認できる。
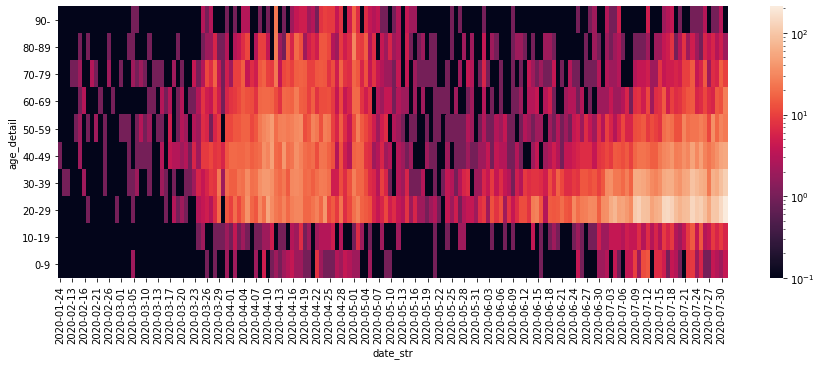
ログスケールのヒートマップは以下を参照。Warningが出たがとりあえず動いた。
データに0があるとエラーになるため0を0.1に置き換えるという大雑把な処理をしているので要注意。
df_ct_hm_re = df_ct_hm.replace({0: 0.1})
min_value = df_ct_hm_re.values.min()
max_value = df_ct_hm_re.values.max()
log_norm = mpl.colors.LogNorm(vmin=min_value, vmax=max_value)
cbar_ticks = [math.pow(10, i) for i in range(math.floor(math.log10(min_value)),
1 + math.ceil(math.log10(max_value)))]
plt.figure(figsize=(15, 5))
sns.heatmap(df_ct_hm_re, norm=log_norm, cbar_kws={"ticks": cbar_ticks})
ちなみに、ヒートマップで可視化するというアイデアは以下の記事のフロリダの例を見て知った。
フロリダのグラフを作成した@zorinaqさんが、ヒートマップ以外にも今後の予測など様々なグラフを作成するコードを公開されている。ある程度Pythonの知識がないと難しそうだが、興味があれば見てみるといいかもしれない。
退院済フラグ
注意点
上で示したcount()の結果の通り、公開データで退院済フラグが1になっているのは7186件だが、東京都の新型コロナウイルス感染症対策サイトの「退院等(療養期間経過を含む)」は9615人となっている(2020年7月31日 20:30 更新時点)。
データの反映が遅れているだけなのか、なにか理由があるのかわからないが、公開データの退院済フラグは現状とズレがある可能性があることは留意されたい。
積み上げ棒グラフ
年代別陽性者数の推移の例と同様に、退院済フラグの推移を積み上げ棒グラフで見る。前処理として欠損値NaNを0に置き換えている。
print(df['discharged'].unique())
# [ 1. nan]
df['discharged'] = df['discharged'].fillna(0).astype('int')
print(df['discharged'].unique())
# [1 0]
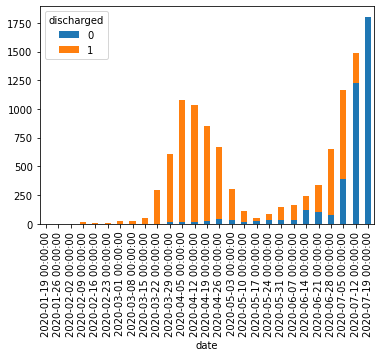
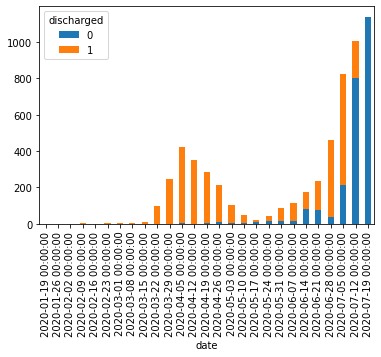
print(pd.crosstab(df['date'], df['discharged']).resample('W', label='left').sum()[:-1].plot.bar(stacked=True))
X軸に時刻が表示されるのが気になる場合は、年代別陽性者数の推移の例のように、インデックスの日時を文字列に変換すればよい。ここではそのままにしている。以降の例も同じ。
このグラフは公表年月日別の退院済フラグの割合を表している。当然ながら、陽性と確認されてから時間が経っている(= 公表年月日が古い)人の多くは退院している(= 退院済フラグが1になっている)。
年代別で確認する。pd.crosstab()ではリストで複数の列を指定するとマルチインデックスとして結果が得られる。
df_dc = pd.crosstab(df['date'], [df['age'], df['discharged']]).resample('W', label='left').sum()
print(df_dc)
# age 0-19 20-39 40-59 60-
# discharged 0 1 0 1 0 1 0 1
# date
# 2020-01-19 0 0 0 1 0 1 0 0
# 2020-01-26 0 0 0 1 0 0 0 0
# 2020-02-02 0 0 0 0 0 0 0 0
# 2020-02-09 0 0 0 2 0 5 0 9
# 2020-02-16 0 0 0 1 0 3 0 6
# 2020-02-23 0 0 0 2 0 3 0 5
# 2020-03-01 0 2 0 5 0 9 0 9
# 2020-03-08 0 0 0 5 0 10 1 10
# 2020-03-15 0 0 0 10 0 27 0 12
# 2020-03-22 0 7 0 100 0 88 2 100
# 2020-03-29 0 16 1 243 4 194 9 139
# 2020-04-05 0 21 5 416 1 368 11 260
# 2020-04-12 1 29 0 350 6 369 10 270
# 2020-04-19 2 30 3 283 6 261 17 247
# 2020-04-26 1 28 8 208 4 161 33 227
# 2020-05-03 1 6 6 99 5 64 23 97
# 2020-05-10 0 2 7 39 3 13 8 38
# 2020-05-17 2 1 10 12 2 8 9 6
# 2020-05-24 3 1 18 25 8 8 5 16
# 2020-05-31 0 2 13 76 8 26 9 13
# 2020-06-07 1 4 17 96 7 10 12 14
# 2020-06-14 3 3 84 93 13 16 17 11
# 2020-06-21 4 6 75 161 18 47 8 15
# 2020-06-28 4 30 37 423 19 88 20 31
# 2020-07-05 44 35 211 613 92 99 46 22
# 2020-07-12 62 4 803 203 250 45 113 4
# 2020-07-19 78 0 1140 0 414 0 171 0
# 2020-07-26 48 0 975 0 320 0 134 0
若年者と高齢者をそれぞれグラフにすると以下の通り。
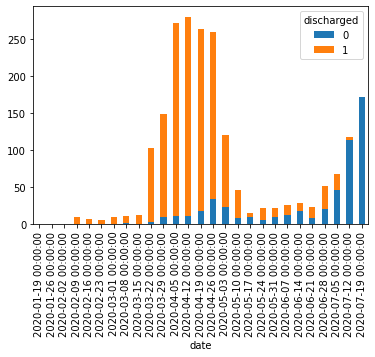
df_dc[:-1]['20-39'].plot.bar(stacked=True)
df_dc[:-1]['60-'].plot.bar(stacked=True)
これも想像通りだが、高齢者のほうが公表年月日が古い人でも退院済フラグが1になっていない割合が大きく、入院が長引きやすいようである。
割合を見やすくするために規格化する。
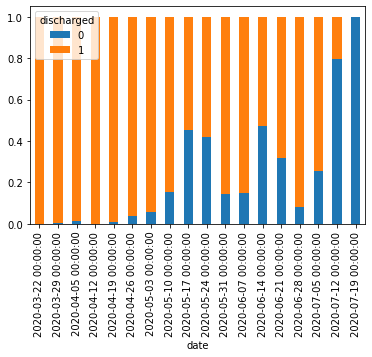
x_young = df_dc[9:-1]['20-39']
x_young_norm = (x_young.T / x_young.sum(axis=1)).T
print(x_young_norm)
# discharged 0 1
# date
# 2020-03-22 0.000000 1.000000
# 2020-03-29 0.004098 0.995902
# 2020-04-05 0.011876 0.988124
# 2020-04-12 0.000000 1.000000
# 2020-04-19 0.010490 0.989510
# 2020-04-26 0.037037 0.962963
# 2020-05-03 0.057143 0.942857
# 2020-05-10 0.152174 0.847826
# 2020-05-17 0.454545 0.545455
# 2020-05-24 0.418605 0.581395
# 2020-05-31 0.146067 0.853933
# 2020-06-07 0.150442 0.849558
# 2020-06-14 0.474576 0.525424
# 2020-06-21 0.317797 0.682203
# 2020-06-28 0.080435 0.919565
# 2020-07-05 0.256068 0.743932
# 2020-07-12 0.798211 0.201789
# 2020-07-19 1.000000 0.000000
x_young_norm.plot.bar(stacked=True)
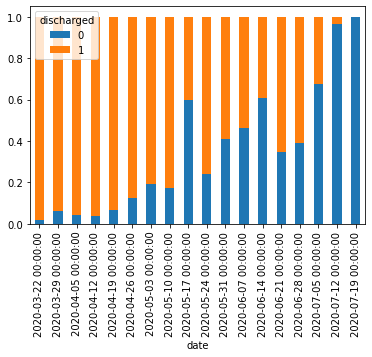
x_old = df_dc[9:-1]['60-']
x_old_norm = (x_old.T / x_old.sum(axis=1)).T
print(x_old_norm)
# discharged 0 1
# date
# 2020-03-22 0.019608 0.980392
# 2020-03-29 0.060811 0.939189
# 2020-04-05 0.040590 0.959410
# 2020-04-12 0.035714 0.964286
# 2020-04-19 0.064394 0.935606
# 2020-04-26 0.126923 0.873077
# 2020-05-03 0.191667 0.808333
# 2020-05-10 0.173913 0.826087
# 2020-05-17 0.600000 0.400000
# 2020-05-24 0.238095 0.761905
# 2020-05-31 0.409091 0.590909
# 2020-06-07 0.461538 0.538462
# 2020-06-14 0.607143 0.392857
# 2020-06-21 0.347826 0.652174
# 2020-06-28 0.392157 0.607843
# 2020-07-05 0.676471 0.323529
# 2020-07-12 0.965812 0.034188
# 2020-07-19 1.000000 0.000000
x_old_norm.plot.bar(stacked=True)
若年者と高齢者の退院済フラグが1になっていない割合を並べて示すと以下の通り。やはり高齢者のほうが入院が長引いている傾向を示している。
pd.DataFrame({'20-39': x_young_norm[0], '60-': x_old_norm[0]}).plot.bar()
Matplotlibで処理
これまでの例はDataFrameのplot()やseabornの関数を使って比較的簡単に処理できたが、場合によってはMatplotlibで処理する必要がある。
新規陽性者の総数の推移を例とする。
ここではvalue_counts()で日時の列をカウントして公表年月日ごとの新規陽性者の総数を算出する。sort_index()でソートしないと多い順に並ぶので注意。
- pandas.DataFrame.sort_index — pandas 1.1.0 documentation
- pandas.DataFrame, Seriesをソートするsort_values, sort_index
s_total = df['date'].value_counts().sort_index()
print(s_total)
# 2020-01-24 1
# 2020-01-25 1
# 2020-01-30 1
# 2020-02-13 1
# 2020-02-14 2
# ...
# 2020-07-27 131
# 2020-07-28 266
# 2020-07-29 250
# 2020-07-30 367
# 2020-07-31 463
# Name: date, Length: 164, dtype: int64
print(type(s_total))
# <class 'pandas.core.series.Series'>
print(type(s_total.index))
# <class 'pandas.core.indexes.datetimes.DatetimeIndex'>
これまでの例とは異なり、DataFrameではなくSeriesだが考え方はどちらでも同じ。
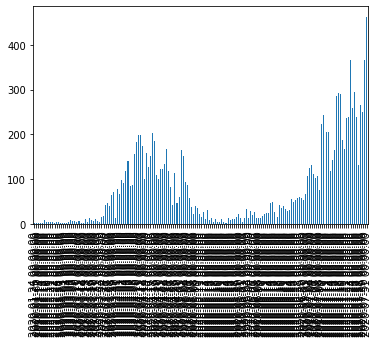
plot.bar()で棒グラフを生成すると、以下のようにX軸が重なってしまう。
s_total.plot.bar()
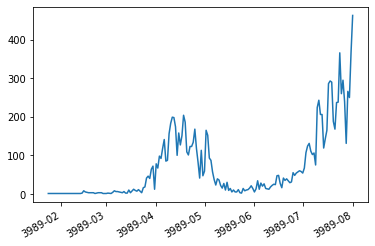
前週比の例でplot()で折れ線グラフを生成する場合は日時を適当にフォーマットしてくれると書いたが、この場合はうまくいかない。
s_total.plot()
plot()でもうまくいかないのは、インデックスに設定された日時データが周期的ではないからだと思われる(詳しく調べていないので違ってたらすみません)。
前週比の例では週ごとのデータが抜けなく存在していたが、この例では2020-01-26や2020-01-27などの陽性者数が0の日時はデータが存在していない。
reindex()とpd.date_range()を利用して、陽性者数が0の日時にも値を0としてデータを追加する。
- pandas.DataFrame.reindex — pandas 1.1.0 documentation
- pandas.date_range — pandas 1.1.0 documentation
- pandas.DataFrameの行・列を任意の順に並べ替えるreindex
s_total_re = s_total.reindex(
index=pd.date_range(s_total.index[0], s_total.index[-1]),
fill_value=0
)
print(s_total_re)
# 2020-01-24 1
# 2020-01-25 1
# 2020-01-26 0
# 2020-01-27 0
# 2020-01-28 0
# ...
# 2020-07-27 131
# 2020-07-28 266
# 2020-07-29 250
# 2020-07-30 367
# 2020-07-31 463
# Freq: D, Name: date, Length: 190, dtype: int64
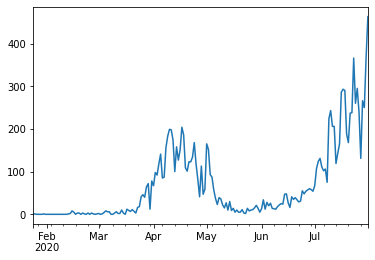
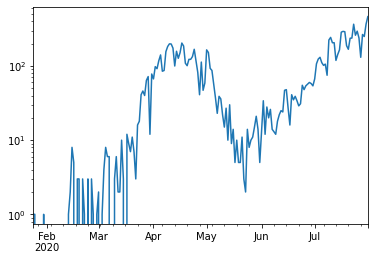
こうすると、plot()で日時が適当にフォーマットされる。ついでに書いておくとログスケールにしたい場合はlogy=True。
s_total_re.plot()
s_total_re.plot(logy=True)
この場合でもplot.bar()はダメ。
s_total_re.plot.bar()
折れ線グラフ以外の種類の場合は、Matplotlibで処理する必要がある。
Formatter, Locatorを設定し、Matplotlibのbar()でグラフを生成する。
- matplotlib.axis - Formatters and Locators — Matplotlib 3.3.0 documentation
- matplotlib.dates — Matplotlib 3.3.0 documentation
- matplotlib.axes.Axes.bar — Matplotlib 3.3.0 documentation
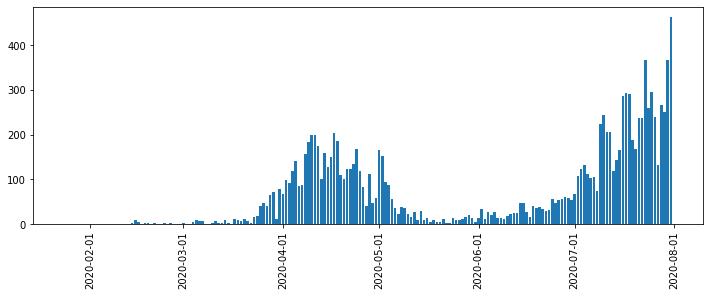
fig, ax = plt.subplots(figsize=(12, 4))
ax.xaxis.set_major_locator(mpl.dates.AutoDateLocator())
ax.xaxis.set_major_formatter(mpl.dates.DateFormatter('%Y-%m-%d'))
ax.xaxis.set_tick_params(rotation=90)
ax.bar(s_total.index, s_total)
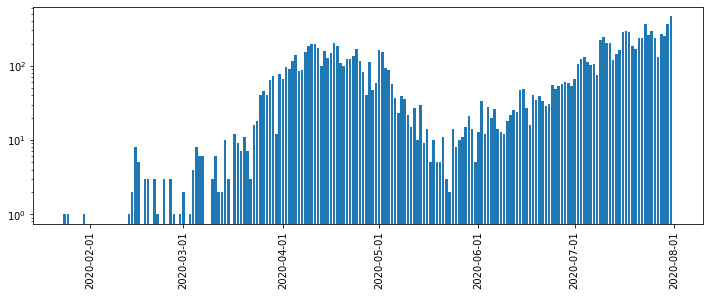
ログスケールにしたい場合はset_yscale('log')。
fig, ax = plt.subplots(figsize=(12, 4))
ax.xaxis.set_major_locator(mpl.dates.AutoDateLocator())
ax.xaxis.set_major_formatter(mpl.dates.DateFormatter('%Y-%m-%d'))
ax.xaxis.set_tick_params(rotation=90)
ax.set_yscale('log')
ax.bar(s_total.index, s_total)
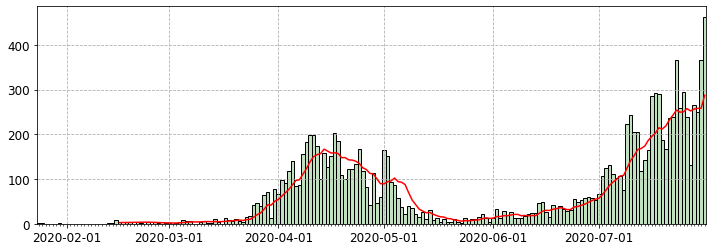
移動平均線を追加したい場合は、rolling()を用いる。
print(s_total.rolling(7).mean())
# 2020-01-24 NaN
# 2020-01-25 NaN
# 2020-01-30 NaN
# 2020-02-13 NaN
# 2020-02-14 NaN
# ...
# 2020-07-27 252.285714
# 2020-07-28 256.428571
# 2020-07-29 258.142857
# 2020-07-30 258.285714
# 2020-07-31 287.285714
# Name: date, Length: 164, dtype: float64
fig, ax = plt.subplots(figsize=(12, 4))
ax.xaxis.set_major_locator(mpl.dates.AutoDateLocator())
ax.xaxis.set_minor_locator(mpl.dates.DayLocator())
ax.xaxis.set_major_formatter(mpl.dates.DateFormatter('%Y-%m-%d'))
ax.xaxis.set_tick_params(labelsize=12)
ax.yaxis.set_tick_params(labelsize=12)
ax.grid(linestyle='--')
ax.margins(x=0)
ax.bar(s_total.index, s_total, width=1, color='#c0e0c0', edgecolor='black')
ax.plot(s_total.index, s_total.rolling(7).mean(), color='red')
上の例では参考までに設定をいくつか加えた。margins(x=0)は左右の余白の切り詰め。色の指定は名称でもカラーコードでも可能。
さいごに
公開されているデータは限られていますが、それでも自分であれこれ弄って試してみると理解が深まると思います。みなさんも是非やってみてください。