Kaggle - Data Science Bowl 20171の1位解法2の調査記事です.
[1位] Document and code of the grt123 team
Author: Liao
Kaggle Discussion: https://www.kaggle.com/c/data-science-bowl-2017/discussion/32555
Writeup: https://github.com/lfz/DSB2017/blob/master/solution-grt123-team.pdf
Code: https://github.com/lfz/DSB2017
開発環境
- Deep Learning Library: Pytorch
- GPU: 8個のTITAN X
流れ
- 前処理
- 肺結節の検出
- 悪性腫瘍の判別
前処理
- 生データの信号値はHU値とする.
肺野のマスクデータの抽出
- ボクセルサイズを1mmに設定
- 各スライス画像に対してガウスぼかし
- -600を閾値として2値化(0は空気)
- 連結成分数が30未満の領域は除外
- 各スライス画像に対して面積が$6000mm^2$以上かつスライス画像の中心からの距離の平均がが"62mm"以上の領域は除外.
- 残りの領域を抽出し肺野領域のマスクデータを構築.
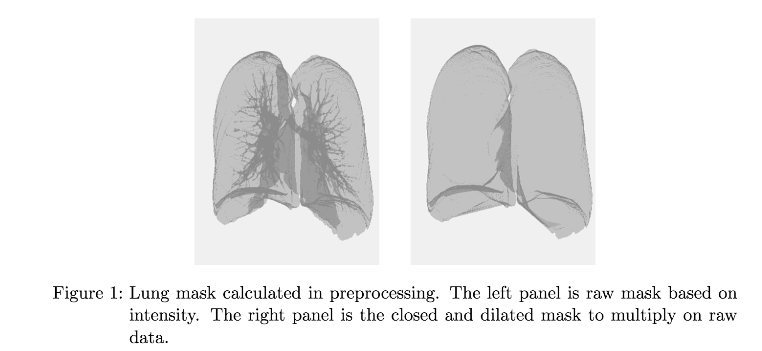
図1: 肺野部分のマスクデータ(文献3より引用)
Convex hull & Dilation
- 最も大きい2つの連結領域の体積が同じぐらいになるまでerode
- 各スライスに対してマスク領域の凸包を取りマスク領域を広げる.(ただし面積が50%以上増加する場合は実施しない)
- マスクデータは肺野より10ボクセルほど大きめになる.
信号値の正規化
- -1200〜600でHU値を[-1200,600]になるようclippingして,0〜255に線形変換
- mask領域外は170でpadding(170は組織と同等の信号強度)
- 信号値が210以上(大抵は骨)の領域も170に変更.
データセット
- LUNA16(以下LUNAと略)とKaggle Stage I(以下Kaggle1と略)を利用.
- LUNAには放射線科医による肺結節のアノテーションデータあり
- Kaggle1には症例ごとに悪性腫瘍か否かのラベルのみ存在
データセットの問題点
- LUNAには悪性腫瘍とは関係ないことが多い小さい肺結節が存在
- LUNAとKaggle1で肺結節に違いが存在(LUNAでは稀だがKaggle1に散見)
- 60mm以上の肺結節
- 気管支上の肺結節
回避策
- 6mm未満のLUNAの肺結節は除外
- Kaggle1の肺結節に手動でラベル付け
- 著者は肺がんに関する専門知識を持っていないが,後述の実験に役立った.
- さらに重要なこととして,悪性腫瘍の判断ははい結節の検出率が悪くても見つけ荒れるよう設計する必要があったので,ちょうど良かったのかも.
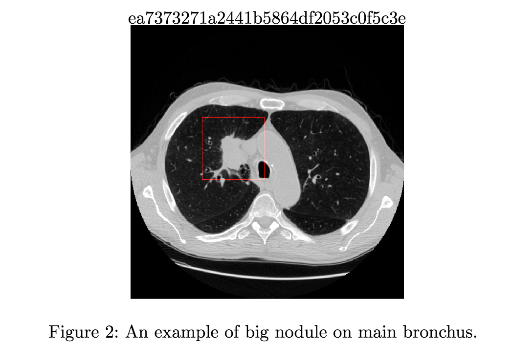
図2: 気管支上にへばりついている肺結節(文献3より引用)
肺結節の検出
- 肺結節の検出のために3Dの畳み込みニューラルネットを利用.
- 基本構造はproposal netの3次元版
訓練に対してPatch-based
- 訓練時に,症例ごとのボリュームデータをまるごと使いたかったが,GPUメモリの都合上断念.
- CTデータを128x128x128にクロップしたパッチ画像を入力とした.
- 下記の二種類のパッチ画像をランダムに抽出
- 少なくとも1つの結節影を含むデータ(入力の70%)
- 肺野からランダムに抽出(残りの入力30%)
- 肺の領域外までパッチ画像が及んだときは170でpadding
- Data Augmentation
- 左右の反転
- 0.8〜1.15で拡大縮小
- 他処理(回転,軸の入れ替え)は改善に繋がらず.
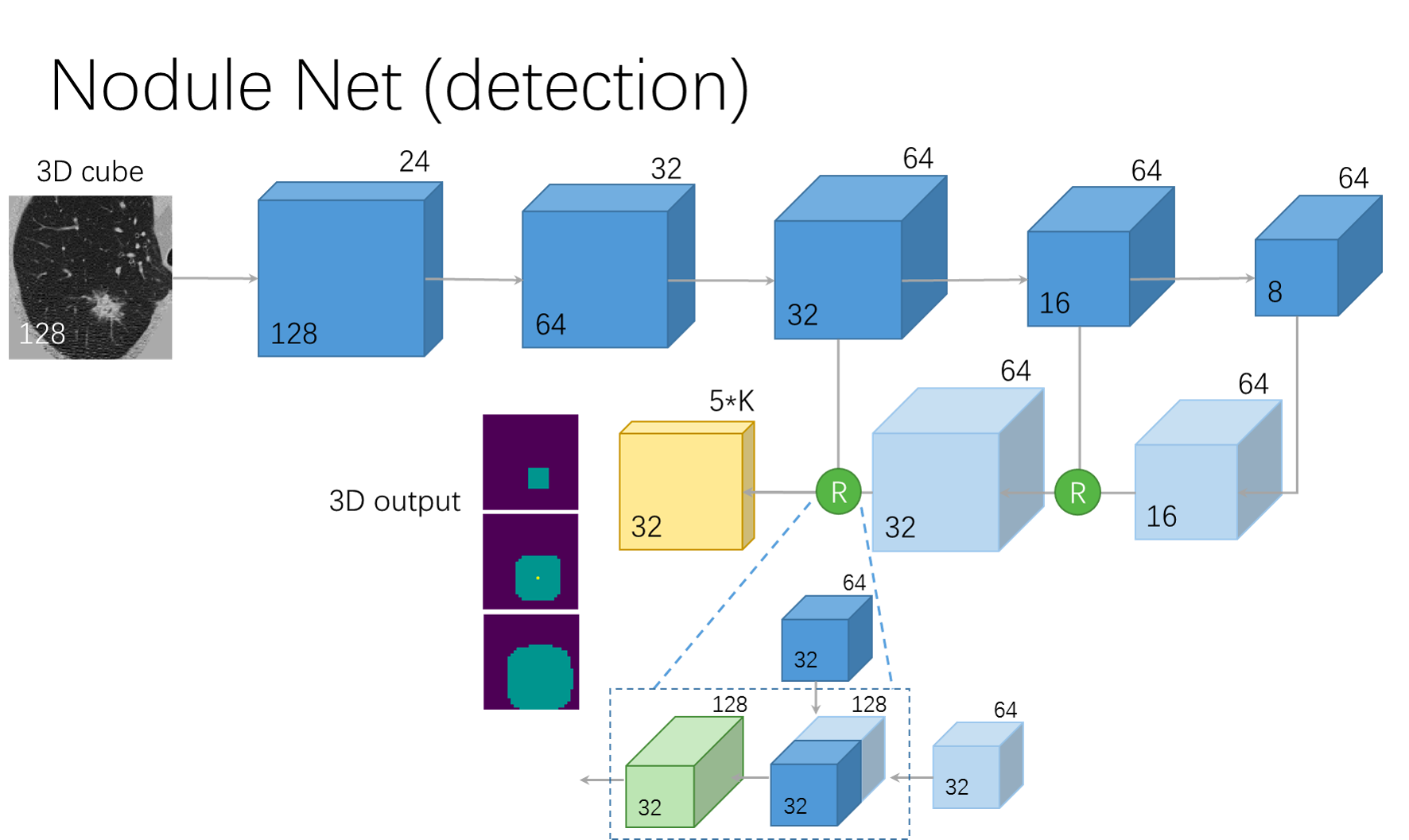
ネットワーク構造
- U-Netベースのネットワーク
- bounding box regressionとクラス分類スコアの算出
- ネットワークは10,30,60mmの異なるスケールのアンカーを持つ.
- 出力のマップサイズは32x32x5x3
- bounding box labelsは$(\frac{\delta x}{x}, \frac{\delta y}{y}, \frac{\delta z}{z}, \log(\delta r))$とL1 loss
- Intersection over Union(IOU)を用いてクラスを分類
- IOUが0.5以上なら正のサンプル,0.02以下なら負のサンプルとして扱った.
図3: 肺結節検出ネットワーク(文献3より引用)
正例の割合調整
- 小さい肺結節は大きい肺結節より数が多いので,大きい肺結節を抽出する割合を2倍に増やした.(大きい肺結節のほうが悪性腫瘍である可能性が高いため)
検出の難しい負例の探索
- 負のサンプル数は圧倒的に多いので,なるだけ正のサンプルと割合が近づくよう,判断の難しい負のサンプル(Hard
Negative4)を下記方法で抽出-
- ネットワークを用いて確信度を算出
-
- N個の負のサンプルが候補として挙げられる.
-
- 候補にある負のサンプルを確信度の高い順にソートして上位n件を判断の難しい負のサンプルとして抽出
-
悪性腫瘍の判別
ネットワーク構造
- 肺結節検出ネットワーク(以下N-Net)
- それぞれのproposalに対して,N-Netの最後の畳込み層を抽出
- N-Netの上位5件の候補を入力候補とした
- 入力サイズは128の特徴量からなる32x32x32のcubeで図4の緑色の箇所
- 2x2x2ボクセルでmax pooling
- 予測確率Pは$P=1 - (1-P_d) \Pi_i (1 - P_i)$とした.ここで$P_d$はダミーの肺結節の確率
- 上手く学習が進むよう,$P_i$が0.03より低い場合は損失として$-\ln(P_i)$を加算
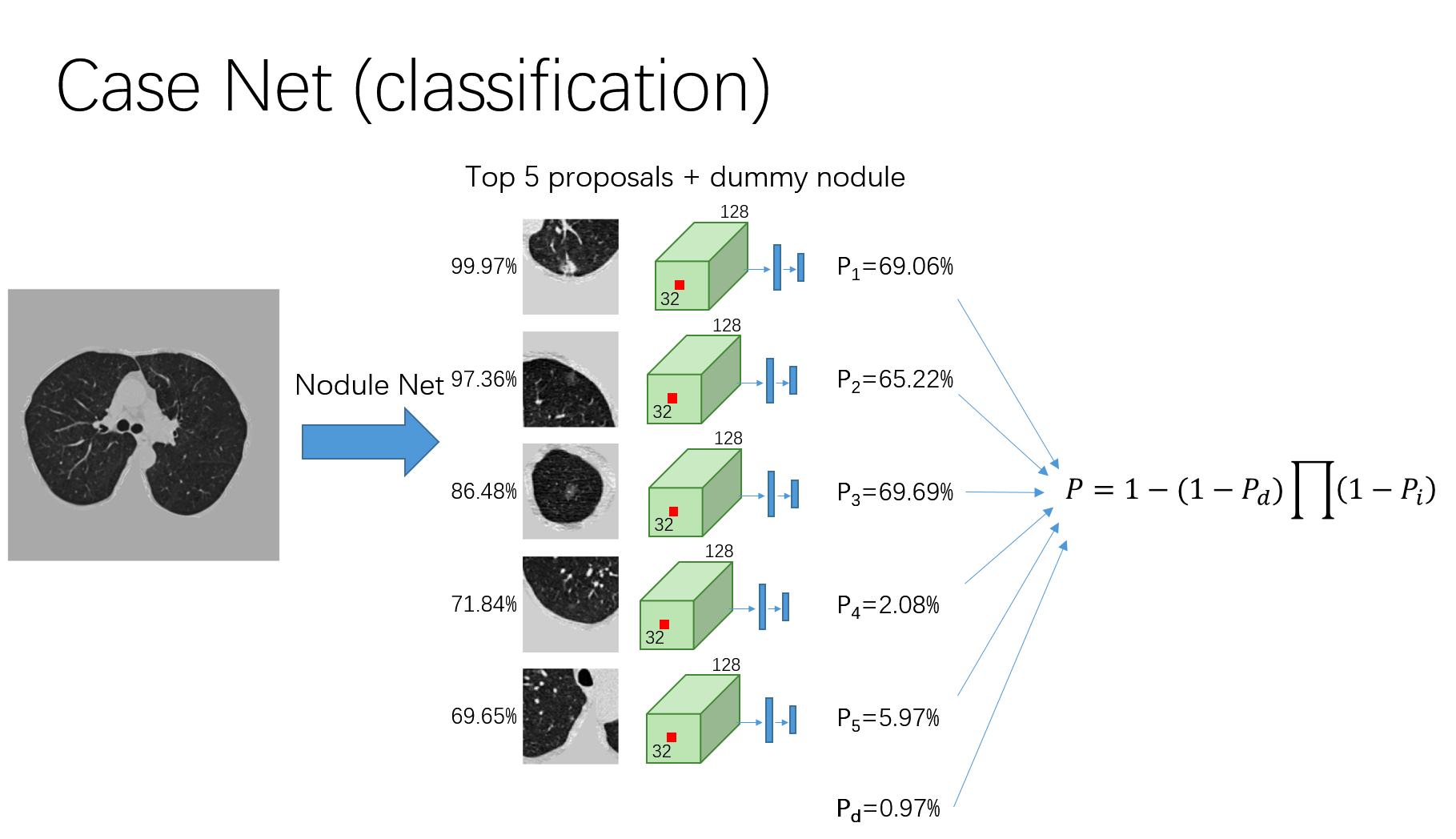
図4: 悪性腫瘍ネットワーク(文献3より引用)
工夫
- 肺結節検出用ネットワーク0の性能を最大限に活かせるよう,エポックごとに悪性腫瘍ネットワークも同時に学習
- 畳み込み層は両方のネットワークで重みを共有
- Data Augmentationとしては,flip, resize, swap, rotationを実施.
実装
- Optimizer: SGD(損失が減少しづらくなったら0.1倍にlearning rateを調整)
References
-
Kaggle, Data Science Bowl 2017, 2017. ↩
-
Liao, [Document and code of the grt123 team](Discussion: https://www.kaggle.com/c/data-science-bowl-2017/discussion/32555), 2017. ↩
-
grt123, Solution of the 'grt123' Team, 2017. ↩ ↩2 ↩3 ↩4
-
Harada, 知能情報論 物体検出, 2017. ↩