データ分析に使うライブラリとして古典的な機械学習がベースのScikit-Learnと数理モデルがベースのstatsmodelsがあります。
重回帰分析をやる分には結果が同じになりますが、ロジスティック回帰だと実は結果が異なるようになります。
決定境界の描画関数
Scikit-Learn用
from sklearn import preprocessing
import matplotlib.pyplot as plt
import numpy as np
def showline_clf(x, y, model, modelname, x0="x0", x1="x1"):
fig, ax = plt.subplots(figsize=(8, 6))
X, Y = np.meshgrid(np.linspace(*ax.get_xlim(), 1000), np.linspace(*ax.get_ylim(), 1000))
XY = np.column_stack([X.ravel(), Y.ravel()])
x = preprocessing.minmax_scale(x)
model.fit(x, y)
Z = model.predict(XY).reshape(X.shape)
plt.contourf(X, Y, Z, alpha=0.1, cmap="brg")
plt.scatter(x[:, 0], x[:, 1], c=y, cmap="brg")
plt.xlim(min(x[:, 0]), max(x[:, 0]))
plt.ylim(min(x[:, 1]), max(x[:, 1]))
plt.title(modelname)
plt.colorbar()
plt.xlabel(x0)
plt.ylabel(x1)
plt.show()
statsmodels用
from sklearn import preprocessing
import matplotlib.pyplot as plt
import numpy as np
def showline_clf2(x, y, model, modelname, x0="x0", x1="x1"):
fig, ax = plt.subplots(figsize=(8, 6))
X, Y = np.meshgrid(np.linspace(min(x[:, 1]), max(x[:, 1]), 1000), np.linspace(min(x[:, 2]), max(x[:, 2]), 1000))
XY = []
for i in range(len(X.ravel())):
XY.append([1, X.ravel()[i], Y.ravel()[i]])
Z = np.argmax(model.predict(XY), axis=1).reshape(X.shape)
plt.contourf(X, Y, Z, alpha=0.1, cmap="brg")
plt.scatter(x[:, 1], x[:, 2], c=y, cmap="brg")
plt.title(modelname)
plt.colorbar()
plt.xlabel(x0)
plt.ylabel(x1)
plt.show()
データの読み込み
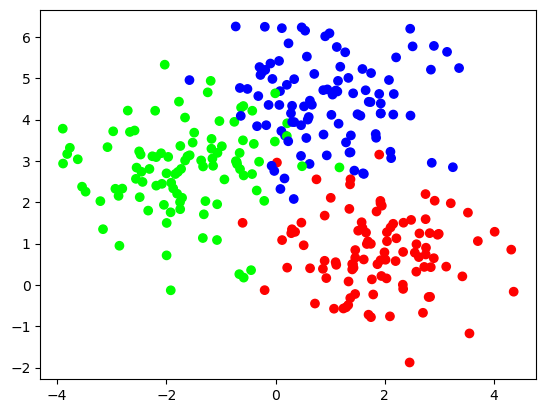
x, y = make_blobs(n_samples=300, centers=3, random_state=0, cluster_std=1.0)
plt.scatter(x[:, 0], x[:, 1], c=y, cmap="brg")
plt.show()
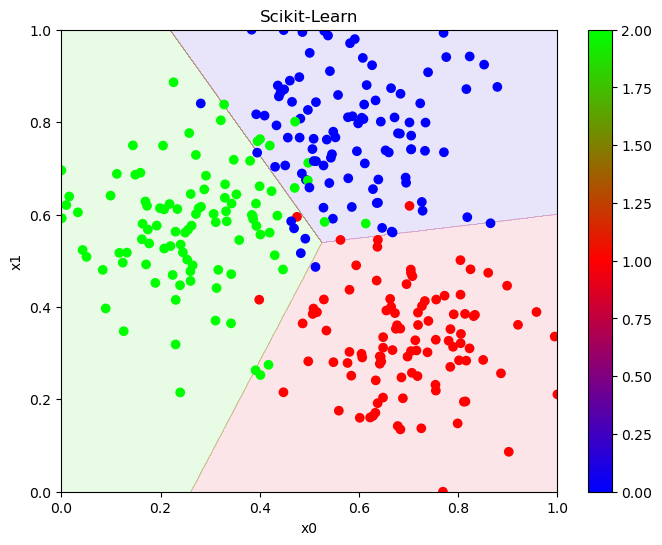
Scikit-Learnの決定境界
model1 = LogisticRegression()
showline_clf(x, y, model1, "Scikit-Learn", x0="x0", x1="x1")
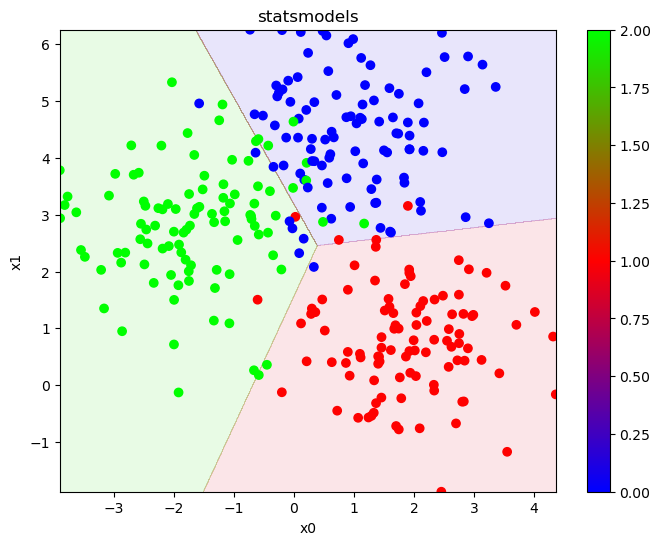
statsmodelsの決定境界
x = sm.add_constant(x)
model2 = sm.MNLogit(y, x).fit_regularized()
showline_clf2(x, y, model2, "statsmodels", x0="x0", x1="x1")
少しだけ決定境界が変わっている事が分かります。
なのでただ予測をする分には切片を追加する事を考慮する事と分類の結果の出力の仕方を追加する以外は特に変わりはありませんが、分析をするとなると少し変わります。
オッズ比の計算
ロジスティック回帰は自然対数に係数乗をするとオッズ比が算出されます。
では実際にオッズ比を見てみましょう。
クロス集計表の作成
df = pd.read_csv("LungCanser.csv")
df_cross = pd.crosstab(df["Smoke"], df["LungCanser"])
df_cross
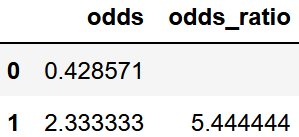
オッズ比の算出
cross = df_cross.values
odds = []
for i in range(len(cross)):
odds.append(cross[i][1]/cross[i][0])
odds_ratio = ["", odds[1]/odds[0]]
df_odds = pd.DataFrame(odds)
df_odds.columns = ["odds"]
df_odds_ratio = pd.DataFrame(odds_ratio)
df_odds_ratio.columns = ["odds_ratio"]
df_odds = pd.concat([df_odds, df_odds_ratio], axis=1)
df_odds
オッズ比は約5.4444となりました。
statsmodelsのロジスティック回帰でオッズ比算出
x = sm.add_constant(df["Smoke"])
y = df["LungCanser"]
model3 = sm.Logit(y, x).fit_regularized()
np.exp(model3.params["Smoke"])
5.444429034587729
一致する値になりました
Scikit-Learnのロジスティック回帰でオッズ比算出
model4 = LogisticRegression()
model4.fit(df["Smoke"].values.reshape(-1, 1), df["LungCanser"])
np.exp(model4.coef_[0][0])
4.707396266875112
先ほどの決定境界の時から分かる通り少し係数が異なるためオッズ比が異なります。
この係数の違いを大きいと見るか小さい誤差と見るかでどちらか楽な方を使えばいいと思いますが、正確な方を使いたいならstatsmodelsになります。
まとめ
統一してよ