以前書いた記事で自由に値を入れてシミュレーションするならVIFを計算して多重共線性を考慮した上で値を入れましょうと書いたことがあります。
今回はそのVIFについて書いていきます。
独立変数間の多重共線性を検出するための指標の1つ。独立変数間の相関係数行列の逆行列の対角要素であり、値が大きい場合はその変数を分析から除いた方がよいと考えられる。10を基準とすることが多い。
『統計WEB』より
分かりやすく言えば説明変数間で独立していない変数があります。でじゃあそういう変数ってどういうものかっていうと変数間で相関がある物だと考えてください。
当たり前ですけど変数間で相関があったらそのまま値を入れちゃダメで、相関に基づく回帰した値を入れないと実データと乖離する可能性があり再現性に難がある可能性があります。
そこでVIFを計算して変数間で相関のある変数を削除して全ての変数を独立させます。
ではここからは具体的にプログラムとデータを使ってやっていきましょう。
ライブラリのインポート
import statsmodels.api as sm
import matplotlib.pyplot as plt
import seaborn as sns
import pandas as pd
import numpy as np
データの読み込み
今回はScikit-Learnにある糖尿病のデータセットをCSVにしたものを読み込みます。
ただ、今回は値を見てもらえば分かる通り最初から標準化されています。
df = pd.read_csv("diabetes.csv")
df.head()
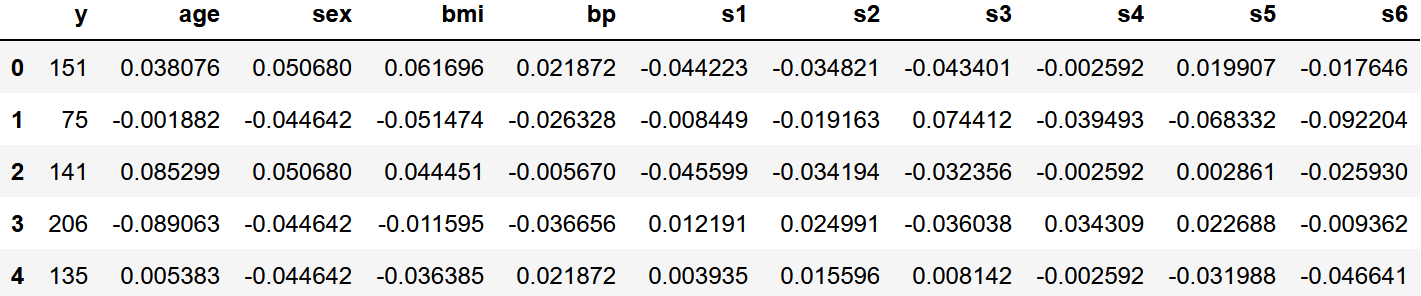
このデータからなんとなくs1からs6までのどこかで相関がありそうな気がしますよね?
説明変数と目的変数を分ける
相関係数とVIFは説明変数で計算しますので分離する必要があります。
y = df["y"]
x = df.drop("y", axis=1)
相関行列を出力
x.corr()
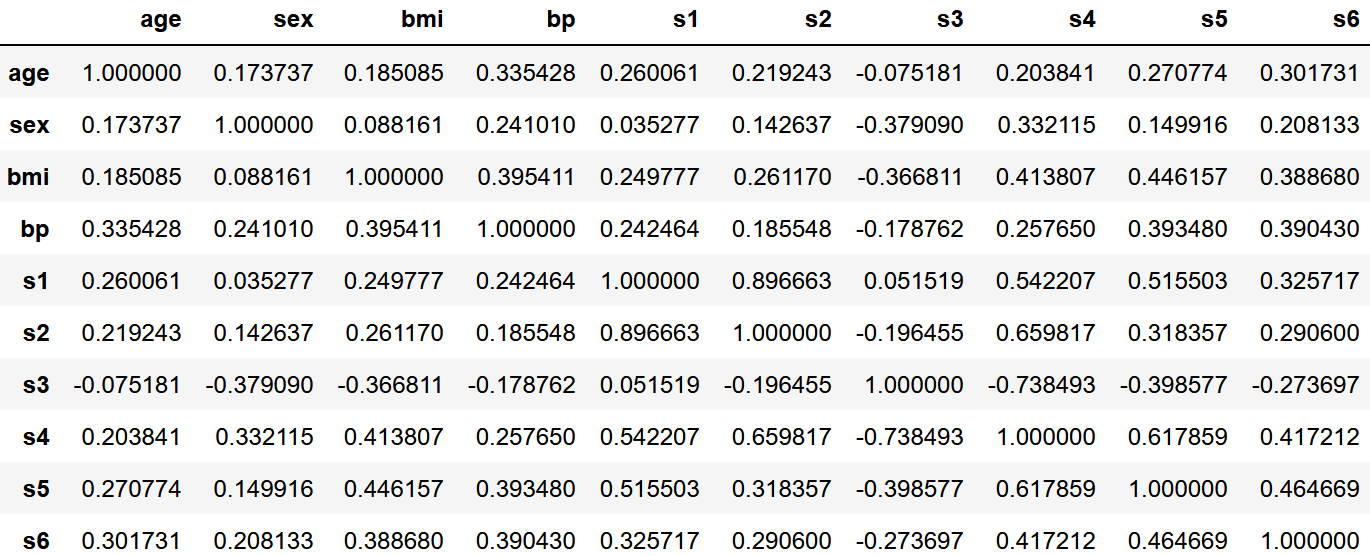
実際に見てみると予想通りs1からs6までで強い相関が見られるところがあります。
散布図を描画
データ分析の基本となりますが、相関係数だけでは外れ値や異常値は分からないので一応散布図も見ましょう。
sns.pairplot(x)
plt.show()
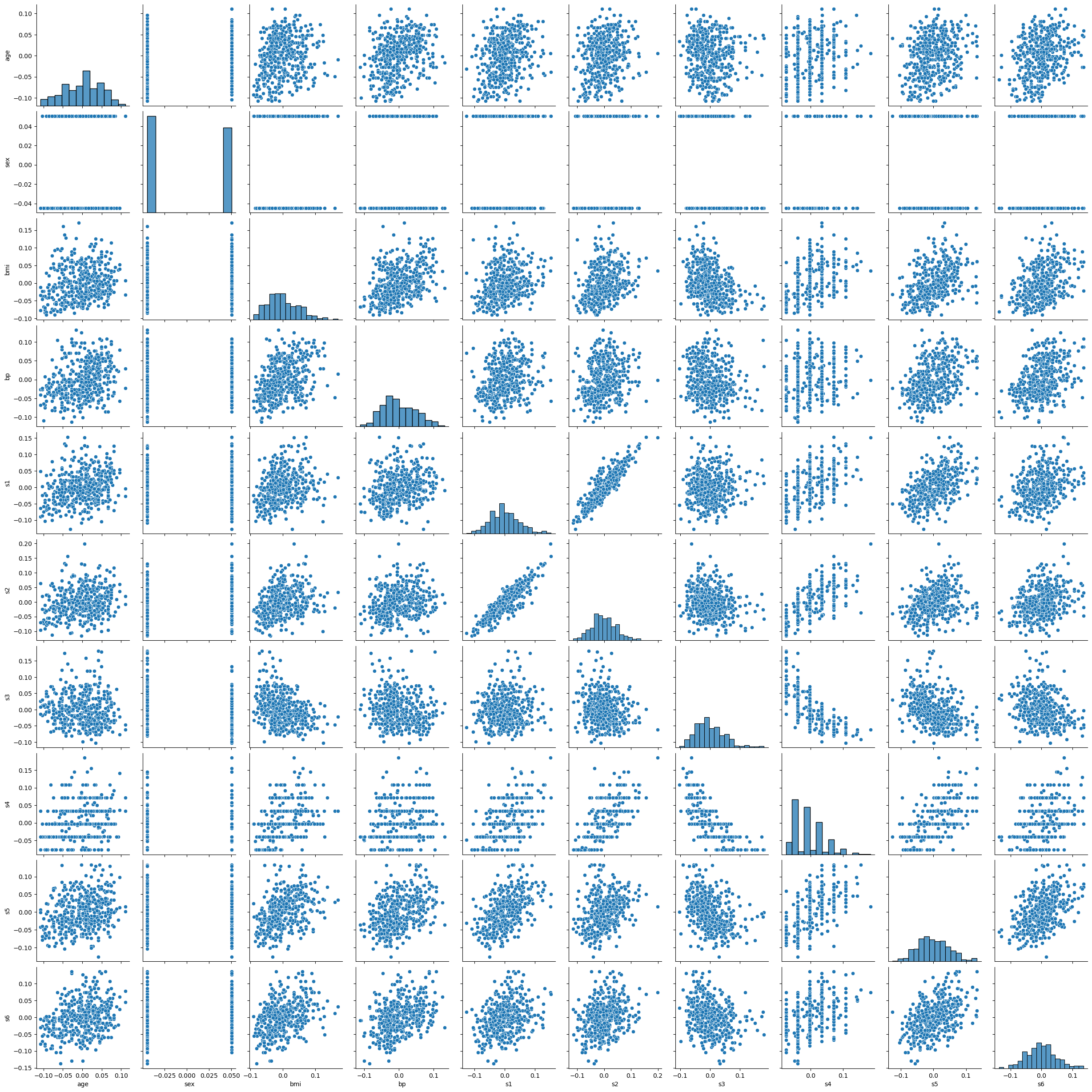
今回は特に外れ値や異常値の類いは見受けられないのでそのまま進めます。
VIFの算出
やり方は簡単で、相関行列の逆行列の対角要素取り出し、それをデータフレームにして変数名を入れて昇順・降順にします。この時、見方として10を超えるデータを削除対象としますが、それは絶対値で見ます。
vif = np.diag(np.linalg.inv(x.corr().values))
df_vif = pd.DataFrame(vif)
df_vif.columns = ["VIF"]
df_vif.index = x.corr().columns
df_vif.sort_values("VIF")
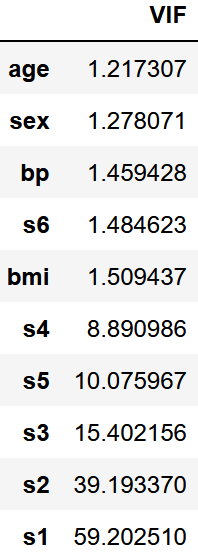
この結果から「s5」「s3」「s2」「s1」が削除対象になります。
これによってあとは学習モデルを使って学習させ、自由に値を入れる事でシミュレーションができます。
正確に書けば間違っていることもあると思いますが、あまり記事になっていなかったので今回書きました。
※ただし欠落性バイアスが発生する可能性も考慮してデータの削除を行ってください