前回の記事、
【超初心者向け】Kaggleのはじめかた part2
ではデータセットの取得からモデルの設計、結果の評価、kernelからの提出まで一通り経験してみました。
今回はMachine learningのlevel 2を解説していきたいと思います。
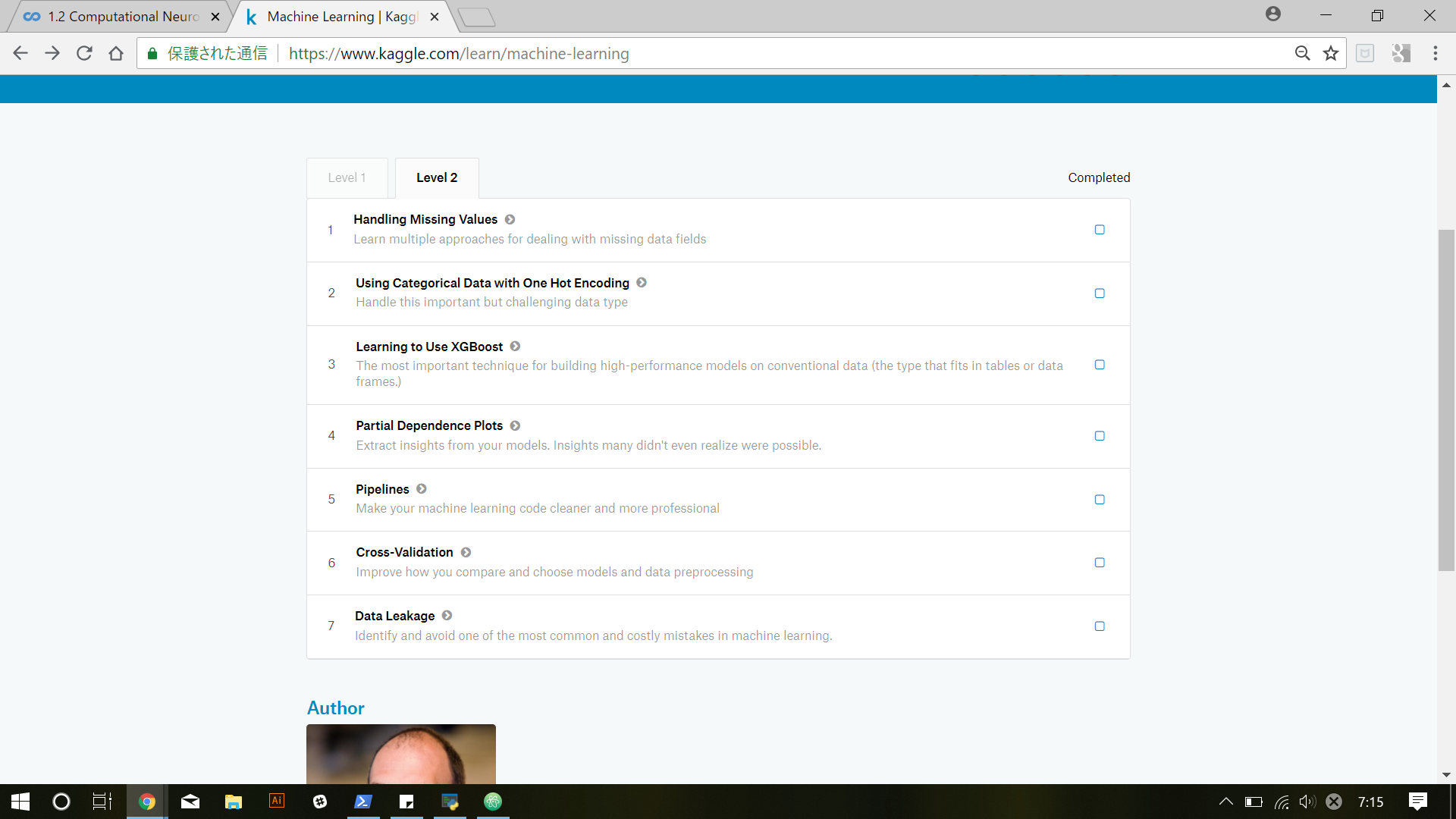
- Handling Missing Values
- Using Categorical Data with One Hot Encoding
- Learning to Use XGBoost
- Partial Dependence Plots
- Pipelines
- Cross-Validation
- Data Leakage
1.データセットに含まれる欠損値の取り扱い
(今回はここまで😅)
前回のlevel 1で使用したkernelを今回もそのまま使います
1. データセットに含まれる欠損値の取り扱い
前回使用したkernelの最後の行から始めます。
データに欠損値(not a number)があると、多くの一般的な機械学習ライブラリではerrorを引き起こしてしまうため、この問題をいかに解決するかは重要です。
まず、データセット上の欠損値を概観するには
print(data.isnull().sum())
を使用します
前回最後に使用したデータ名は、trainでしたので、
print(train.isnull().sum())
として実行してみると、
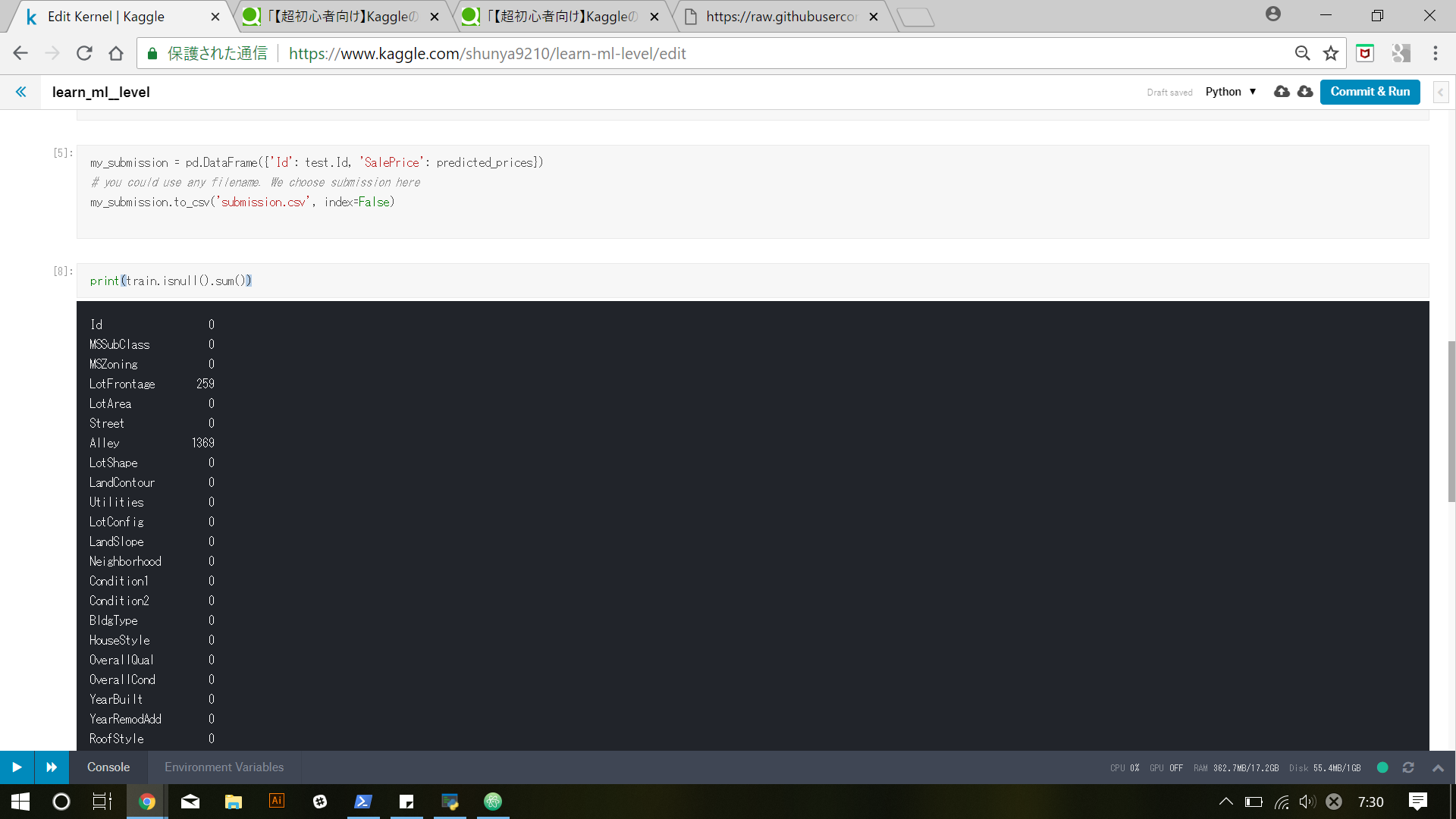
このようにカテゴリーごとにどれだけ欠損値が含まれているのかを確認できます
実際に
train
でデータを表示してみると、いたるところでNaNが確認できます。
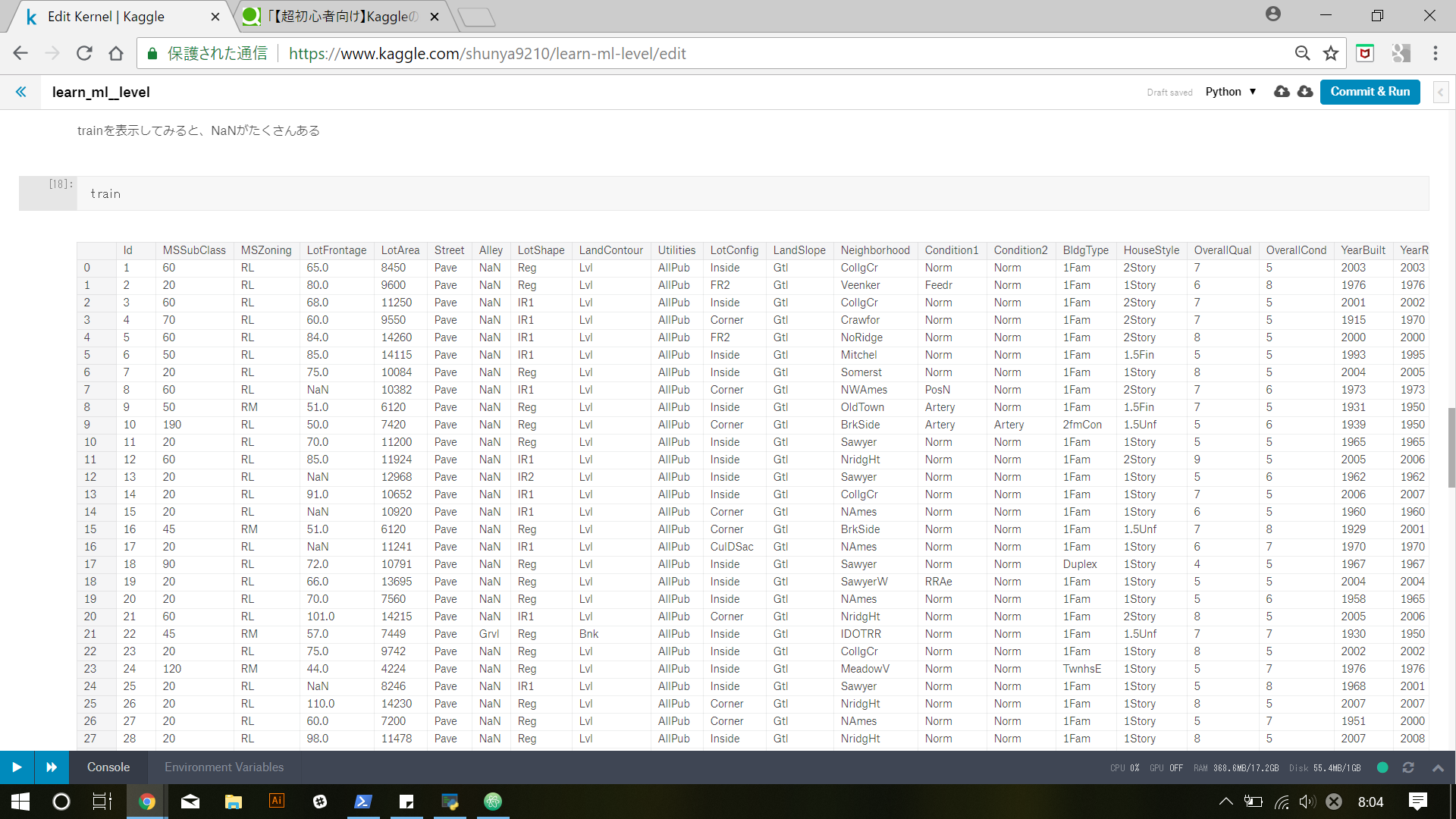
では、どのようにしてこれらの欠損値を処理するのか。ここでは3つの方法が紹介されています。
1) A Simple Opinion: Drop Columns with Missing Values
一つ目は少々強引なやり方で、欠損値を含むColumnsをそれごと取り除いてしまう方法です。
data_without_missing_values = train.dropna(axis=1)
data_without_missing_values
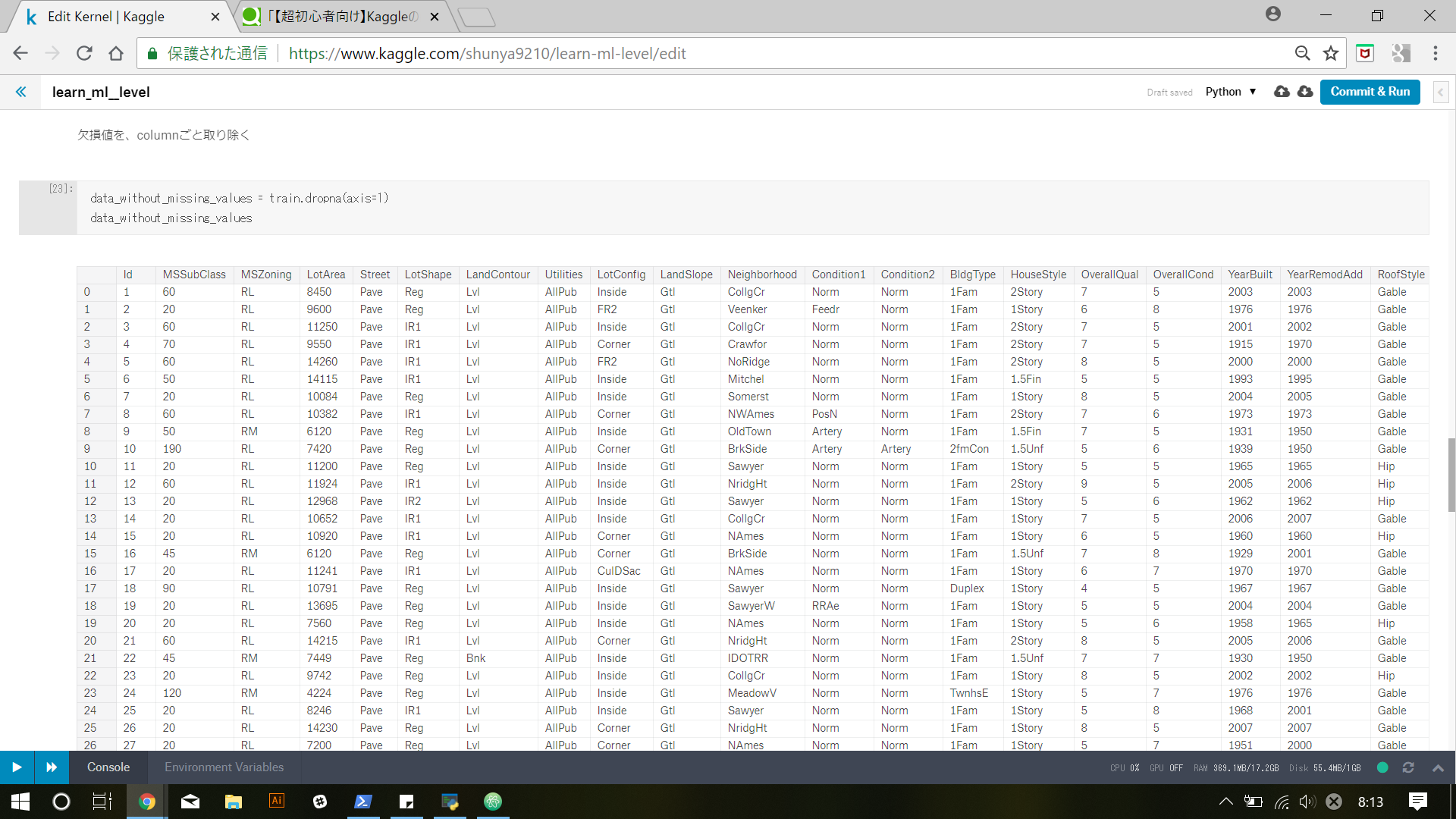
たしかに、columnごと取り除かれていますね。ただ、trainデータだけではなく、testデータからも同一のcolumnsを取り除く必要があるので、
cols_with_missing = [col for col in original_data.columns
if original_data[col].isnull().any()]
redued_original_data = original_data.drop(cols_with_missing, axis=1)
reduced_test_data = test_data.drop(cols_with_missing, axis=1)
test_dataやoriginal_dataのところを、自分が使うデータセット名に変えればそのまま使えますね。
2) A Better Opinion: Imputation
さすがにcolumnを1つまるごと落としてしまうと、重要な情報が失われ精度が低下してしまうためいくらかの値を埋め合わせます。
from sklearn.preprocessing import Imputer
my_imputer = Imputer()
data_with_imputed_values = my_imputer.fit_transform(original_data)
今回は文字列を含んでいるため、うまくできませんでした。。。後で勉強します😢
3) An Extension To Imputation
# データセットのコピーを作成
new_data = original_data.copy()
# 何を補填したかわかるように新しいcolumnsを作成して記録する
cols_with_missing = (col for col in new_data.columns
if new_data[c].isnull().any())
for col in cols_with_missing:
new_data[col + '_was_missing'] = new_data[col].isnull()
# 埋め合わせる
my_imputer = Imputer()
new_data = my_imputer.fit_transform(new_data)
今回はできませんでしたが、このようなやり方もあるようです
例
import pandas as pd
# Load data
melb_data = pd.read_csv('../input/melbourne-housing-snapshot/melb_data.csv')
from sklearn.ensemble import RandomForestRegressor
from sklearn.metrics import mean_absolute_error
from sklearn.model_selection import train_test_split
melb_target = melb_data.Price
melb_predictors = melb_data.drop(['Price'], axis=1)
# 初期データのコピーを取っておく
melb_numeric_predictors = melb_predictors.select_dtypes(exclude=['object'])
最初にこれを実行すると、
FileNotFoundError: File b'../input/melbourne-housing-snapshot/melb_data.csv' does not exist
というエラーが出たので、同じkernel内のdataタブをクリックして、add dataボタンをさらにクリックしてmelbourne housing snapshotを追加しておきます。
分かりにくいですが、右上の"<"に埋め込まれています。
1) columnまるまる除去
cols_with_missing = [col for col in X_train.columns
if X_train[col].isnull().any()]
reduced_X_train = X_train.drop(cols_with_missing, axis=1)
reduced_X_test = X_test.drop(cols_with_missing, axis=1)
print("Mean Absolute Error from dropping columns with Missing Values:")
print(score_dataset(reduced_X_train, reduced_X_test, y_train, y_test))
自分はlevel1の途中をすっぽかして提出用のkernelを作成していたので、X_trainが定義されていませんでした。前回のqiitaの記事に戻り確認します。
def score_dataset(X_train, X_test, y_train, y_test):
model = RandomForestRegressor()
model.fit(X_train, y_train)
preds = model.predict(X_test)
return mean_absolute_error(y_test, preds)
まず、のちに使うscore_datasetという関数を定義してあげます。
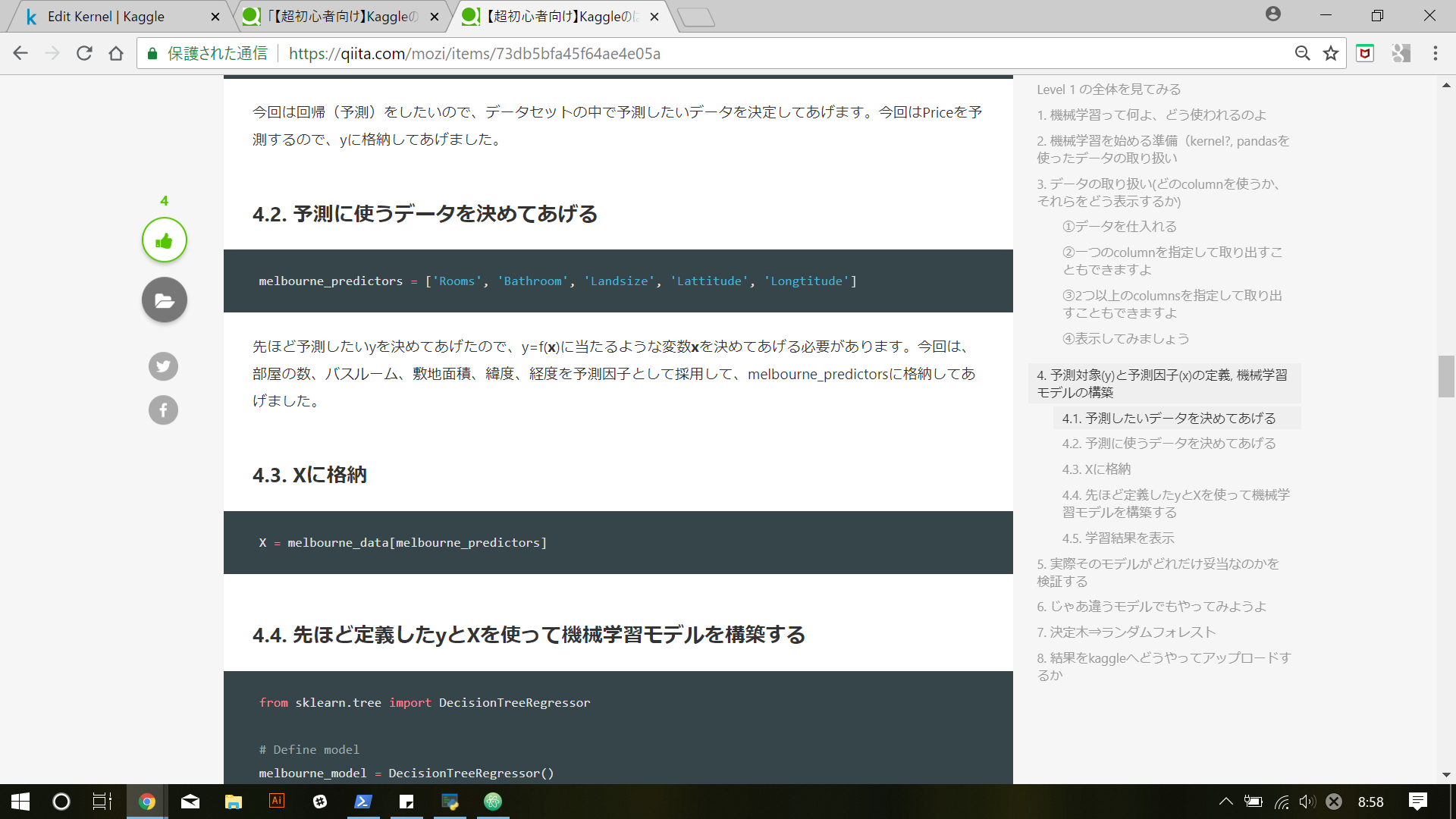
# Xとyの定義からおさらい
melbourne_predictors = ['Rooms', 'Bathroom', 'Landsize', 'Lattitude', 'Longtitude']
X = melb_data[melbourne_predictors]
y = melb_data.Price
# データ分割用のライブラリをインストール
from sklearn.model_selection import train_test_split
# 訓練データと検証データに分ける
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state = 0)
# やっと本題
# 1)でやったことの復習です
cols_with_missing = [col for col in X_train.columns
if X_train[col].isnull().any()]
reduced_X_train = X_train.drop(cols_with_missing, axis=1)
reduced_X_test = X_test.drop(cols_with_missing, axis=1)
print("Mean Absolute Error from dropping columns with Missing Values:")
print(score_dataset(reduced_X_train, reduced_X_test, y_train, y_test))
こんなかんじですね。結果は
Mean Absolute Error from dropping columns with Missing Values:
347381.741366
2) 埋め合わせ
from sklearn.preprocessing import Imputer
my_imputer = Imputer()
imputed_X_train = my_imputer.fit_transform(X_train)
imputed_X_test = my_imputer.transform(X_test)
print("Mean Absolute Error from Imputation:")
print(score_dataset(imputed_X_train, imputed_X_test, y_train, y_test))
結果
Mean Absolute Error from Imputation:
205575.689237
3)
imputed_X_train_plus = X_train.copy()
imputed_X_test_plus = X_test.copy()
cols_with_missing = (col for col in X_train.columns
if X_train[col].isnull().any())
for col in cols_with_missing:
imputed_X_train_plus[col + '_was_missing'] = imputed_X_train_plus[col].isnull()
imputed_X_test_plus[col + '_was_missing'] = imputed_X_test_plus[col].isnull()
# Imputation
my_imputer = Imputer()
imputed_X_train_plus = my_imputer.fit_transform(imputed_X_train_plus)
imputed_X_test_plus = my_imputer.transform(imputed_X_test_plus)
print("Mean Absolute Error from Imputation while Track What Was Imputed:")
print(score_dataset(imputed_X_train_plus, imputed_X_test_plus, y_train, y_test))
結果
Mean Absolute Error from Imputation while Track What Was Imputed:
204026.405381
長くなってしまったので、今回はここまで