はじめに
データ分析用のWebアプリを簡単に作成できるWebフレームワークStreamlitの学習・実践も兼ねて、Genbank形式ファイルからゲノム内の遺伝子情報を比較・可視化するWebアプリを開発してみました。
本記事では、開発したWebアプリGBKvizの紹介と利用した技術要素の解説をします。
対象読者としては、以下に興味がある人を想定しています。
- Streamlitを利用したデータ分析・可視化Webアプリ開発
- バイオインフォ分野のデータ分析プログラム開発
- ゲノム・遺伝子データの比較・可視化
GBKvizの紹介
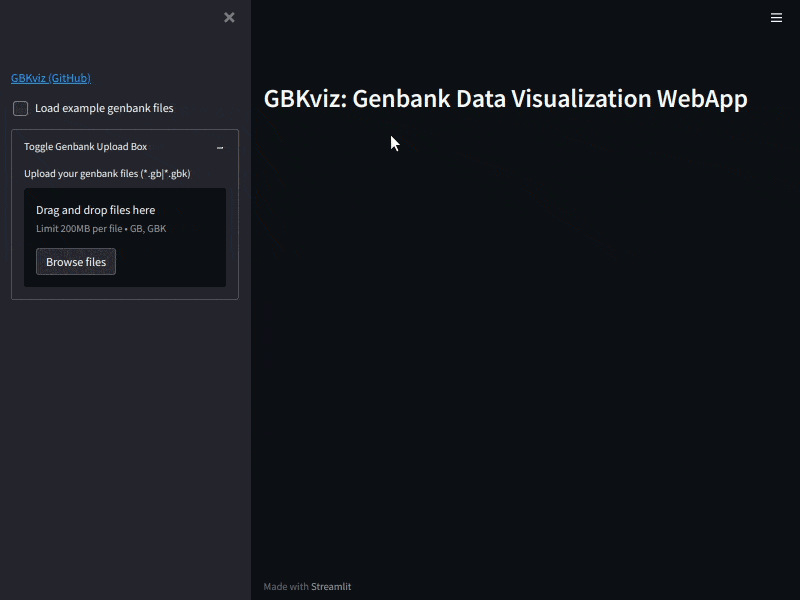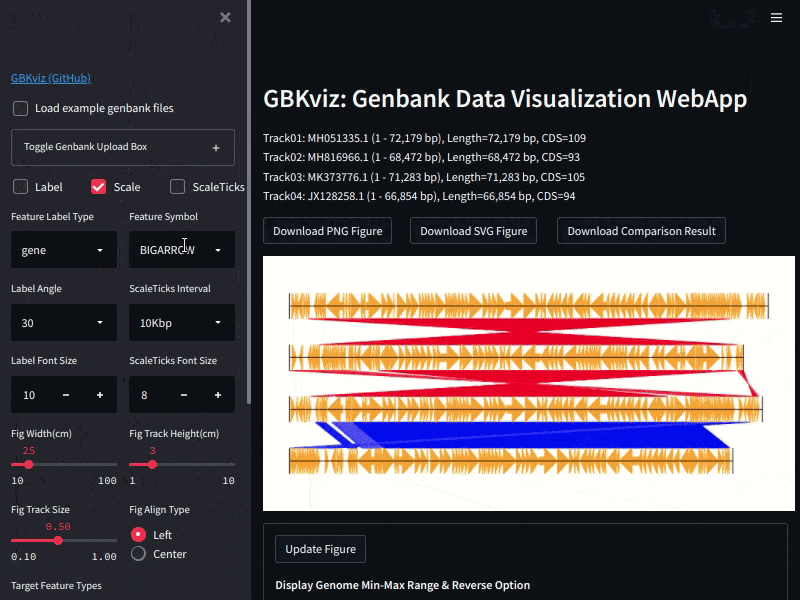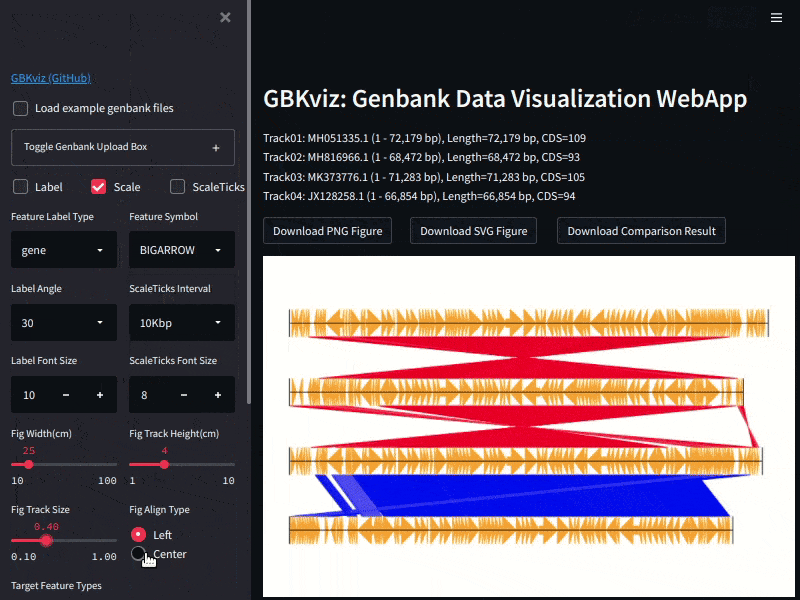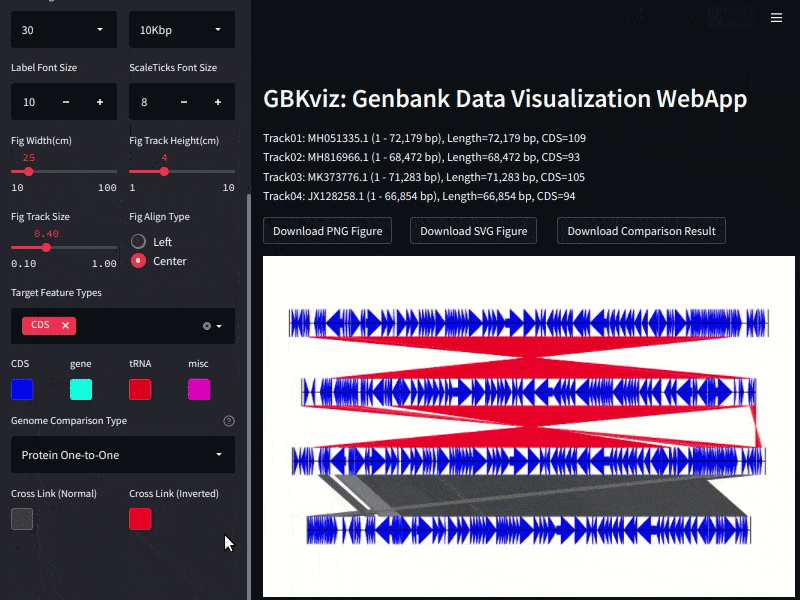
GBKvizは入力されたGenbankファイルから下記のようなゲノム内遺伝子の比較・可視化図をさくっと作成可能にすることを目的として開発したWebアプリです。
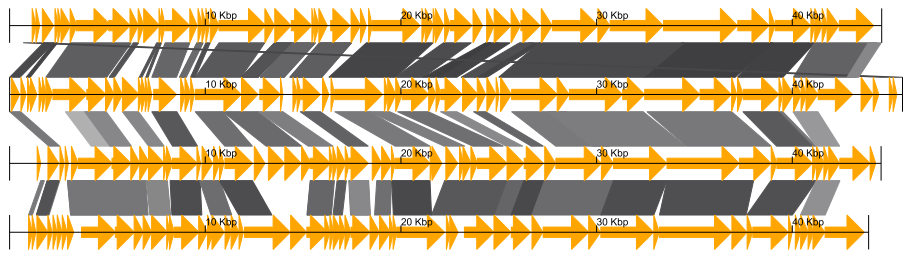
同様の操作が可能な既存プログラムとしてEasyFigがありましたが、操作性や細かい図調整に少し不満に思うところもあったので、個人的な学習も兼ねて新規に開発をしてみました。
↓開発したWebアプリの操作感はこんな感じです (こちらからお試しできます)。
開発には主に Streamlit (Webフレームワーク)、GenomeDiagram (ゲノム・遺伝子可視化ライブラリ)、MUMmer (ゲノム相同性比較ツール)を利用しています。ソースコードや詳細な仕様はGitHubリポジトリから確認できます。
インストール&実行方法
パッケージ化してPyPIに登録しているので、pipコマンドでインストールできます。
pip install gbkviz
ゲノム比較実行には、MUMmer のインストールも必要
インストール後、下記コマンドでGBKvizをブラウザ上でローカル起動できます。
gbkviz_webapp
お試し版 (Streamlit Cloud)
Streamlit Cloudを利用することで、GitHubリポジトリ上のStreamlitで開発したソースコードを無料で簡単にデプロイできます。デプロイしたお試し版のGBKvizには、こちらからアクセスできます。
Streamlit CloudはCPU・メモリリソースが少ないため、動作が不安定かもしれないです。
GBKvizの技術要素
-
Streamlit
データ分析向けWebフレームワーク -
GenomeDiagram (BioPython)
ゲノム遺伝子・比較情報の可視化ライブラリ
Streamlit
Streamlitは機械学習やデータサイエンス分野のデータ分析用のWebアプリをPythonの記述のみで開発できるWebフレームワークです。開発の手軽さから最近よく利用・紹介されているので、既に知っている人も多いかと思います。
↓各種パラメータを変更した結果をインタラクティブに確認するWebアプリと相性が良いと思います。

Streamlitの全体像を把握するには、公式ドキュメントが一番分かりやすいと思うのでおすすめです。以下の記事も分かりやすいので参考になるかと思います。
Streamlitは基本的に簡単ですが、個人的に少し躓いたところもあったので、その辺りの注意すべき点について以降に記載します。
注意点①:キャッシュの有効化
Streamlitは各ウィジェットが押下・変更される度に、都度コード全体が再実行される仕様となっています。そのため、最初のデータ読み込みのような実行内容が同一かつ重たい処理も何も対策をしないと毎回実行され、画面がしばらく固まることがあります。Streamlitでは関数単位でキャッシュを有効化することで、再実行時にキャッシュした結果を利用できるようになるので、画面が固まるようなこともなくなります。
どの機能のキャッシュを有効化するか検討して、関数化した処理のキャッシュを有効化するのはStreamlitの動作を効率化する上で重要です。GBKvizの開発では、負荷の大きいゲノム比較処理のキャッシュ有効化前後で使用感が大きく変化しました。
関数のキャッシュ有効化はデコレータ(@st.cache)で簡単にできるようになっています。
注意点②:アップデートの確認
Streamlitはほぼ毎月アップデートをしていて、機能追加・改修が継続的に行われています。そのため、ネット上のStreamlitに関する記事内容は古くなっている場合も良くあり、記事を参考にしてもうまくいかずに躓くことがありました。公式の最新のAPI ReferenceやChangeLogを参照して、自分の使っているバージョンのStreamlitの機能を最大限活用して開発できるようにするといいと思います。
GenomeDiagram (BioPython)
GBKvizではBioPythonの実装機能の一つであるGenomeDiagramを利用してゲノム遺伝子とその相同性比較結果の可視化を行っています (ゲノム相同性比較にはMUMmerを利用)。
GenomeDiagramは直線状あるいは環状にバクテリアゲノムの可視化図を描くことを目的に作成されたライブラリです。ファージ・プラスミド・ミトコンドリア等の小さなゲノムの図を描くのにも適しています。
GenomeDiagramは検索しても情報が少なく、個人的には使い方を把握するのに少し手間取ったので、参考に簡単な実装コード例やGBKvizの実装例を紹介していきます。
簡単な実装・可視化例
GenomeDiagramで3つのゲノム内の遺伝子情報を直線状に可視化するコード例
※ 実行にはBioPythonとreportlabが必要 (pip install biopython reportlab)
from typing import List, Tuple
from Bio.Graphics import GenomeDiagram
from Bio.Graphics.GenomeDiagram import FeatureSet
from Bio.SeqFeature import FeatureLocation, SeqFeature
from reportlab.lib.units import cm
# 3つのゲノム内の遺伝子位置・向きを示す3次元配列
# 通常はGenbankファイルからこうした遺伝子情報を抽出するが、今回はテスト用に適当に作成
# 各配列内の数値は、1. 遺伝子開始位置, 2. 遺伝子終端位置, 3. 向き (順方向=1, 逆方向=-1)、を示す
genomes: List[List[Tuple[int, int, int]]] = [
[(0, 200, 1), (300, 400, -1), (420, 500, -1), (700, 800, 1), (800, 1000, 1)],
[(30, 70, 1), (100, 500, 1), (500, 550, 1), (550, 700, 1), (700, 800, -1)],
[(50, 350, 1), (400, 700, -1), (800, 1100, 1)],
]
# 3つのゲノム内の各遺伝子情報を可視化用に設定
gd = GenomeDiagram.Diagram("Genome Diagram")
for features in genomes:
# Track上への遺伝子設定用オブジェクト(FeatureSet)を作成
gd_feature_set: FeatureSet = gd.new_track(
track_level=0, start=0, end=max([end for (_, end, _) in features])
).new_set()
# 遺伝子設定用オブジェクト(FeatureSet)へ各遺伝子情報(SeqFeature)・描画設定を追加
for (start, end, strand) in features:
feature = SeqFeature(FeatureLocation(start, end, strand))
gd_feature_set.add_feature(feature=feature, color="orange", sigil="BIGARROW")
# ゲノム可視化図設定&出力
gd.draw(format="linear", fragments=1, pagesize=(15 * cm, 50 * cm))
gd.write(filename="test.png", output="png")
上記コードを実行すると、以下のような遺伝子情報可視化図が出力される。
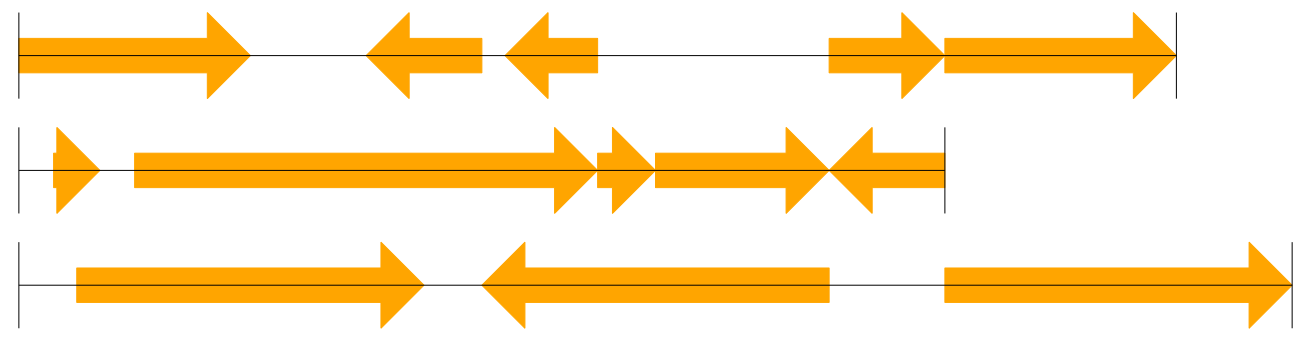
実装コード例の各種関数の引数を変更(color="blue", sigil="ARROW", format="circular")することで、以下のような環状の可視化図の作成もできます。
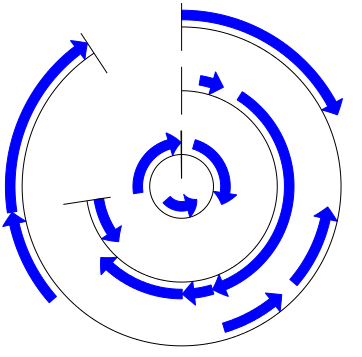
その他にも設定可能な引数パラメータは色々あるので、興味があれば BioPython(GenomeDiagram) API Reference を参照してください。
応用的な実装・可視化例 (GBKviz)
前項で挙げたコード実装例・図例はシンプルなものですが、GBKvizでは各種パラメータ調整・ゲノム相同性比較等をすることで、より詳細な可視化図を柔軟にGenbank形式ファイルから作成できるようにしています。GBKvizのGenomeDiagramを利用した実装に興味があれば、GitHubリポジトリから参照してみてください。
例として、GBKvizでは以下のような図を作成できます。

↑ 4種のファージゲノムの比較・可視化図。相同なゲノム領域をグレーで表示し、類似度の大小を色の濃淡で表現。

↑ 4種の大腸菌の部分ゲノムの比較・可視化図。逆位した相同ゲノム領域は赤色で表現。
おわりに
StreamlitはとてもシンプルなWebフレームワークなので、簡単にデータ比較・可視化Webアプリを開発することができました。GBKvizのコード総数は1000行程度ですが、Streamlit×BioPythonをフル活用することでそれなりに実用的なWebアプリになったなという印象です。Streamlitはデータ分析・可視化Webアプリの開発ハードルを下げてくれる、とても良いWebフレームワークだと思いました。
本記事では、バイオインフォ分野におけるStreamlitを利用した実践的なデータ比較・可視化Webアプリの開発事例について紹介しました。興味を持たれた方は、ぜひStreamlitを利用して開発をしてみてください!!
参考
-
Streamlit - Documentation
Streamlitドキュメンテーション -
Streamlit - Gallery (Science & technology)
科学技術分野(バイオインフォ含む)におけるStreamlitの活用例ギャラリー - BioPython(GenomeDiagram) API Reference
- BioPython Tutorial and Cookbook - Chapter17 Graphics including GenomeDiagram
-
Graphics including GenomeDiagram
GenomeDiagramのAPIリファレンス・利用例・チュートリアル -
MUMmer - GitHub repository
ゲノム比較ツール MUMmer のGitHubリポジトリ