趣旨
表題のままです。Windows で動かそうしたら Symblic Link が使えないOSなんぞ知らんとか言われたので、仕方なく(でもないけど)WSL2 で動かしました。Windows をインストールしただけのマシンから始めたらちょっと大変だったので、忘れないように手順をメモしておきます。
anomalib 2.0.0 対応版の記事も書きました。
ポイント
- Python はバージョン 3.11 でないどダメ
これにつきます。
手順
Windwis 11 Home + WSL2 + RTX 3060Ti, 4060Ti では動きました。Anaconda を使ってますがそこは必須ではないです。バージョンについては、2024.6 の時点で動く最新のセットになっていますが、時間が経過したら変わっているかもしれません。
- NVIDIA の Driver を最新版にする
- Windows 11 に Ubuntu 22.04LTS (WSL2) を入れる
- Ubuntu で cuda 12.1 を入れる
- Anaconda をインストールする
- 環境を作って python 3.11 をインストールする
- Pytorch をインストールする
- anomalib v1.1.1 をインストールする
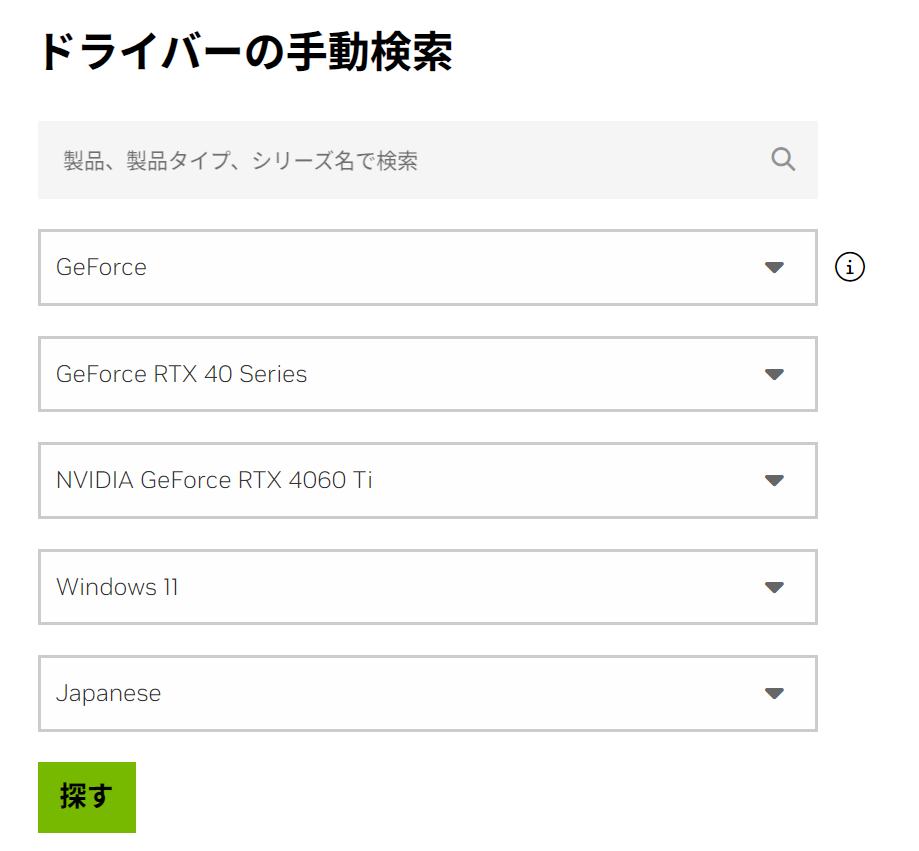
NVIDIA の Driver を最新版にする
上記リンク先で適切なOS、デバイスを選ぶと最新版のドライバをダウンロードできます。
Game Ready 販と Studio 版の選択は、どちらでも問題ありませんがゲームしないなら Studio 版でOKです。
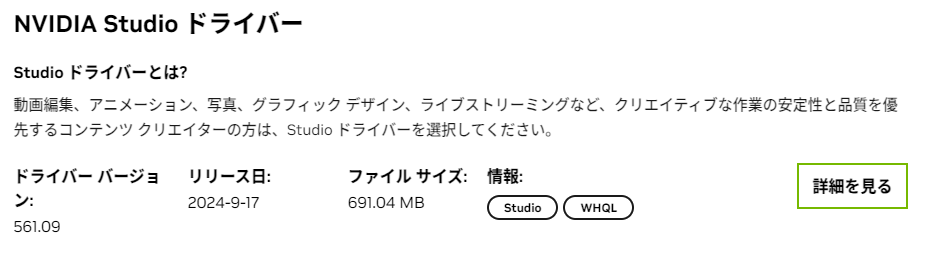
2024.09 の時点では最新版のバージョンは 561.09です。時間がたつとすぐ変わるので、最新版を入れておけばとりあえずOKでしょう。
Windows に Ubuntu 22.04LTS を入れる
Store で Ubuntu と検索して Ubuntu 22.04.3 LTS を見つけてインストールします。
Ubuntu を起動してエラーとか言われたとき
BIOS で CPU の Virtualization の機能を有効にしていないか、Windows の機能の「仮想マシン プラットフォーム」が有効化されていない可能性が高いです。下記記事を参照してください。
- Windwos11のWSLインストール時に0x80370114エラーが出る問題
- [マザーボード] BIOSにVT (Virtualization Technology) を設定しWindowsに仮想マシンをインストールする方法(ASUS)
- Windows Subsystem for Linux のトラブルシューティング
- WSL2 でUbuntuを立ち上げたら、「0x80370102」と「0x800701bc」というエラーが出た
BIOS の Virtualization についてはマザーボードごとに設定方法が違うので、マザーボードのメーカーのページを探して設定するのが間違いないです(上のリンクはASUSのもの)
Ubuntu で cuda 12.1 を入れる
Ubuntu を起動できたら、下記のページに行って書いてある通りにして cuda 12.1 をインストールする。2024/9 時点では 12.5 をインストールして 12.4 対応版の pytorch をインストールしても動くようですが、試していません。
もし過去に cuda をインストールしたことがあるなら、先に uninstall します。しておかないとハマります。
$ apt list --installed | grep cuda
上のコマンドで cuda のライブラリが何か表示されたら、何らかのバージョンがインストールされています。
$ cat /usr/local/cuda/version.json
上記コマンドでインストールされているバージョンがわかります。File not found と言われたら、cuda がインストールされている可能性が高いです。
$ sudo apt remove cuda
$ sudo apt autoremove -y
もし cuda 12.1 以外がインストールされていたら、アンインストールします。上記コマンドで大抵はアンインストールできますが、これをやって何かよからぬことが起こっても自己責任でお願いします。
ほどんどのPCは、上の選択でいけるはず(CPUが特殊なのでない限り)。上の選択でよければ、実行するコマンドは下記の通りです。
$ wget https://developer.download.nvidia.com/compute/cuda/repos/ubuntu2204/x86_64/cuda-ubuntu2204.pin
$ sudo mv cuda-ubuntu2204.pin /etc/apt/preferences.d/cuda-repository-pin-600
$ wget https://developer.download.nvidia.com/compute/cuda/12.1.0/local_installers/cuda-repo-ubuntu2204-12-1-local_12.1.0-530.30.02-1_amd64.deb
$ sudo dpkg -i cuda-repo-ubuntu2204-12-1-local_12.1.0-530.30.02-1_amd64.deb
$ sudo cp /var/cuda-repo-ubuntu2204-12-1-local/cuda-*-keyring.gpg /usr/share/keyrings/
$ sudo apt-get update
$ sudo apt-get -y install cuda-12-1
最後だけ NVIDIA のページと異なっていますが、cuda-12-1 とバージョンを強制的に指定すると間違いが少なくなります(他のバージョンを一度入れたことがあるときにバージョン指定をしていないとハマることがある)
インストールしたバージョンが 12.1.x であることを確認します。
$ cat /usr/local/cuda/version.json | head
{
"cuda" : {
"name" : "CUDA SDK",
"version" : "12.1.0"
},
"cuda_cccl" : {
"name" : "CUDA C++ Core Compute Libraries",
"version" : "12.1.55"
},
"cuda_cudart" : {
cuda 12.1 のインストールにどうしても失敗するときは、下記も参照してください。
Anaconda をインストールする
下記の通りにやります。
Anaconda のバージョンは、2024.09 時点では 2024.06-1 です。このバージョンを使用する場合は、コマンドは下記のようになります。
$ sudo apt-get install libgl1-mesa-glx libegl1-mesa libxrandr2 libxrandr2 libxss1 libxcursor1 libxcomposite1 libasound2 libxi6 libxtst6
$ curl -O https://repo.anaconda.com/archive/Anaconda3-2024.06-1-Linux-x86_64.sh
$ bash ./Anaconda3-2024.06-1-Linux-x86_64.sh
$ rm Anaconda3-2024.06-1-Linux-x86_64.sh
$ source ~/anaconda3/bin/activate
$ conda init
途中でライセンスへの同意を求められたら同意します。
これで conda コマンドが使えるようになります(はず)。
環境を作って python 3.11 をインストールする
例えば下記のようにします。途中で y/N ? とか聞かれたら y と答えます。
$ conda create -n anomalib
$ conda activate anomalib
$ conda install python=3.11
インストールされたバージョンが本当に 3.11.x かどうかを確認します。
$ python --version
Python 3.11.9
Pytorch をインストールする
下記で適切なバージョンなどを選択して、表示されるコマンドを実行します。
ここまでの手順に従ってインストールしてきた場合は、ほぼ下記の設定でいけるはず。cuda 12.5 をインストールしたときは、CUDA 12.4 を選びます。
$ conda install pytorch torchvision torchaudio pytorch-cuda=12.1 -c pytorch -c nvidia
インストールできたら、cuda が有効になっているかをチェックしておきます。
$ python
Python 3.11.9 (main, Apr 19 2024, 16:48:06) [GCC 11.2.0] on linux
Type "help", "copyright", "credits" or "license" for more information.
>>> import torch
>>> torch.cuda.is_available()
True
>>> exit()
torch.cuda.is_available() を実行して True と表示されたら成功です。False になったら、振出しに戻ってどこかで間違っていないか見直します・・・
anomalib v1.1.1 をインストールする
下記のとおりです。
$ pip install anomalib
$ anomalib install
WSL2 を使わず Windows 11 側の anaconda で install すると、途中でシンボリックリンクが作れないといわれてエラーで落ちます。
numpy でエラーが出るとき
anpmalib のインストール時に AttributeError: np.float_was removed in the NumPy 2.0 release. Usenp.float64 instead.. Did you mean: 'float16'? とかエラーが出たら、numpy のバージョンを下げます。
$ pip uninstall numpy
$ pip install "numpy<2.0.0"
$ anomalib install
実行テスト
MVTec のデータをトレーニングに使うときは、MVTec のサイトから直接 tar ファイルをダウンロードしたほうが早いです。
上記からデータ mvtec_anomaly_detection.tar. を取得した場合は、anomalib train を実行するディレクトリに datasets というフォルダを作って、その中にファイルを置くと、anomalib train のダウンロードのフェイズがスキップされます。
$ anomalib train --model Patchcore --data anomalib.data.MVTec
... 中略
2024-06-19 19:45:33,761 - lightning.pytorch.utilities.rank_zero - INFO - GPU available: True (cuda), used: True
INFO GPU available: True (cuda), used: True
... 中略
┏━━━━━━━━━━━━━━━━━━━━━━━━━━━┳━━━━━━━━━━━━━━━━━━━━━━━━━━━┓
┃ Test metric ┃ DataLoader 0 ┃
┡━━━━━━━━━━━━━━━━━━━━━━━━━━━╇━━━━━━━━━━━━━━━━━━━━━━━━━━━┩
│ image_AUROC │ 1.0 │
│ image_F1Score │ 0.9919999837875366 │
│ pixel_AUROC │ 0.9814878702163696 │
│ pixel_F1Score │ 0.730120062828064 │
└───────────────────────────┴───────────────────────────┘
Testing ━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━ 3/3 0:00:10 • 0:00:00 0.19it/s
エラーがでることなく、最後に上のような表が表示されたら成功です。ログの途中に GPU available: True (cuda) という表示がでていれば GPU をちゃんと使えてます。GPU available: False と出ていたら、driver, cuda, python, pytorch のいずれかのインストールに失敗しています。
python 3.12 だと途中で ModuleNotFoundError: No module named 'imp' というエラーが出て止まります。
学習済みモデルは ./results/Patchcore/MVTec/bottle/v*/weights/lightning/model.ckpt に作成されます。v* の * の部分は、学習を実行するたびに自動的にインクリメントされます。
カスタムデータを使って学習させる
学習方法としては、下記の三つの用意の仕方があります。
- 正常なデータだけを学習させる(異常データの学習はしない)
- 正常データを用意し、異常データを自動生成して学習させる
- 正常データと異常データを用意して両方学習させる
基本的には、下にいくほど精度がよくなります。mask データはテスト時のみ使われるようです。
正常なデータのみを使用して学習させる例
学習用データは、下記フォルダに配置してあるものとします。
datasets/mydata/
├── test # テスト用データを配置(必要に応じて)
├── ground_truth # テスト用のデータの正解(マスク)画像を配置(必要に応じて)
└── train
├── good # 学習用正常データを配置
└── bad # 学習用異常データを配置(必要に応じて)
下記の設定ファイルは 1. のケースです。2 や 3 の学習方法を使用したい場合は、必要なファイルを配置して対応する行のコメントアウトを外してください。
class_path: anomalib.data.Folder # 必ず anomalib.data.Folder を指定する
init_args:
name: mydata # データセットの名前
root: "datasets/mydata" # データを配置するフォルダ
normal_dir: "train/good" # 学習用の正常データを配置するフォルダ。datasets/custom/train/good と解釈される
#abnormal_dir: "train/bad" # 学習用の異常データを配置するときは、このコメントアウトをはずします。
normal_test_dir: "test" # テスト用のデータを配置するフォルダ。テストしない場合でも指定は必要っぽい
#mask_dir: "ground_truth" # マスク画像をテスト用の正解データとして使用する場合はこのフォルダに配置します。マスク画像は黒背景に白抜き画像とし、学習データと同サイズ同名である必要があります。
normal_split_ratio: 0
extensions: [".png"]
train_batch_size: 32
eval_batch_size: 32
num_workers: 8
task: segmentation
transform: null
train_transform: null
eval_transform: null
test_split_mode: none # テストしない場合は none にする
test_split_ratio: 0 # テストしない場合は 0 にする
val_split_mode: synthetic # synthetic にする
val_split_ratio: 0.5
seed: null
上記のファイルを作成してから、下記コマンドを実行します。
$ anomalib train --model Patchcore --data custom.yaml
実行に成功すると、./result/Patchcore/mydata/v*/weights/lightning/model.ckpt 以下に学習済みの重みデータが出力されます。v* の * 部分は、実行した回数によって自動的にインクリメントされます。
マスク画像なしでテストする場合は、例えば下記のようにします。テスト結果は、./result/Patchcore/mydata/v*/images/test/ 以下に出力されます。
class_path: anomalib.data.Folder
init_args:
name: mydata
root: "datasets/mydata"
normal_dir: "train/good"
#abnormal_dir: "train/bad"
normal_test_dir: "test"
#mask_dir: "ground_truth"
normal_split_ratio: 0
extensions: [".png"]
train_batch_size: 32
eval_batch_size: 32
num_workers: 8
task: segmentation
test_split_mode: synthetic # synthetic を指定する。
test_split_ratio: 0
val_split_mode: synthetic
val_split_ratio: 0.5
seed: null
transform: null
推論 (prediction)
重みデータを使用した推論は下記のようにします (推論データが datasets/mydata/test 以下にあり、v0 に重みデータが出力されている場合)。
$ anomalib predict --model Patchcore --data datasets/mydata/test --ckpt_path results/Patchcore/mydata/v0/weights/lightning/model.ckpt
推論結果は、results/Patchcore/latest/ 以下に出力されます。出力先は常に同じになるので、何度も推論させると同じフォルダ以下に出力結果のファイルがたまります。
出力先を指定したいときは yaml ファイルを書きます。
data: datasets/mydata/test
ckpt_path: results/Patchcore/mydata/v0/weights/lightning/model.ckpt
default_root_dir: myresult/v0
return_predictions: true
model:
class_path: anomalib.models.Patchcore
上のようなファイル pred.yaml を書いてから、下記のコマンドを実行します。
$ anomalib predict --config pred.yaml
これで myresult/v0 以下に結果が出力されます。default_root_dir を変更すれば、出力先を変更できます。
default_value を変更することで、anomaly として検出する閾値を変更できます。
学習データの変形(transformation)
学習時の transform の設定については、下記を参照してください。
CenterCrop する場合の例です。公式ドキュメントにある yaml ファイルの書き方は間違っていて、そのまま書くとエラーになります。(class_path の前に - は不要)
class_path: anomalib.data.Folder
init_args:
name: mydata
root: "datasets/sound"
normal_dir: "train/good"
#abnormal_dir: "train/bad"
normal_test_dir: "test"
#mask_dir: "ground_truth"
normal_split_ratio: 0
extensions: [".png"]
train_batch_size: 32
eval_batch_size: 32
num_workers: 8
task: segmentation
test_split_mode: none
test_split_ratio: 0
val_split_mode: synthetic
val_split_ratio: 0.5
seed: null
transform:
class_path: torchvision.transforms.v2.CenterCrop
init_args:
size: 224
torchvision V2 で使える transforms は何でも使えるようです。引数が複数必要な関数は配列形式で与えます。たとえば Resize なら以下のような感じです。
transform:
class_path: torchvision.transforms.v2.Resze
init_args:
size: [224, 224]
CLI を使わず python のコードで学習させる
python のコードで書く場合は以下のような感じです。
from anomalib.models import Patchcore
from anomalib.engine import Engine
from anomalib.data.utils import TestSplitMode, ValSplitMode
import anomalib
from torchvision.transforms.v2 import Compose, CenterCrop
datamodule = anomalib.data.image.folder.Folder(
name="mydata",
root="./datasets/mydata",
normal_dir="train/good",
#normal_test_dir="test",
test_split_mode=TestSplitMode.NONE, # テストしないとき NONE
val_split_mode=ValSplitMode.SYNTHETIC, # NONE にすると失敗する
val_split_ratio=0.5,
#test_split_mode=TestSplitMode.SYNTHETIC,
#mask_dir="ground_truth",
task="segmentation",
transform=Compose( # transform を使わない場合はコメントアウト
[
CenterCrop( 224 ),
],
)
)
datamodule.setup()
model = Patchcore()
engine = Engine()
# 学習
engine.fit(datamodule=datamodule, model=model)
# 推論テスト
predictions = engine.predict(
data_path="./datasets/mydata/test/good",
model=model,
)
python のプログラム内で prediction すると、推論結果を学習結果を出力するフォルダ以下にある images というフォルダ(上の場合は results/mydata/v*/images) 以下にまとめて出力してくれます。
テスト付きにする場合は下記のようにします。テスト結果は、results/mydata/v*/images/test 以下に出力されます。
from anomalib.models import Patchcore
from anomalib.engine import Engine
from anomalib.data.utils import TestSplitMode, ValSplitMode
import anomalib
from torchvision.transforms.v2 import Compose, CenterCrop
datamodule = anomalib.data.image.folder.Folder(
name="mydata",
root="./datasets/mydata",
normal_dir="train/good",
normal_test_dir="test",
test_split_mode=TestSplitMode.SYNTHETIC, # syntetic を指定
test_split_ratio=0.2, # 不要かも?
val_split_mode=ValSplitMode.SYNTHETIC,
val_split_ratio=0.5,
#mask_dir="ground_truth",
task="segmentation",
transform=Compose(
[
CenterCrop( 224 ),
],
)
)
datamodule.setup()
model = Patchcore()
engine = Engine()
# 学習
engine.fit(datamodule=datamodule, model=model)
推論のみを python のプログラムでする場合は例えば下記のようにします。
from anomalib.models import Patchcore
from anomalib.engine import Engine
predictions = Engine().predict(
data_path="./datasets/mydata/test", # テストデータのあるフォルダ
model=Patchcore(),
ckpt_path="./results/Patchcore/mydata/v2/weights/lightning/model.ckpt", # 学習済み重みデータのパス
)
上記の場合、結果は ./results/Patchcore/mydata/v2/images 以下に出力されます。繰り返し出力した場合、ファイルは上書きされずに、ファイル名に _1 のようなポストフィックスが付いた状態で出力されます。
推論結果を画像として取得する
results 以下への出力は、あくまでテスト出力なので推論結果以外の画像が混ざった状態で一枚の画像化されています。純粋に異常検知した部位だけを取得したい場合は、自力でデータ処理をする必要があります。
例えば、下記のようにします。
from anomalib.models import Patchcore
from anomalib.engine import Engine
from PIL import Image
import numpy as np
import os, torch
from datetime import datetime
import torchvision.transforms as transforms
# 推論処理
predictions = Engine().predict(
data_path="./datasets/mydata/test",
model=Patchcore(),
# 重みファイルへのパスを書く
ckpt_path="./results/Patchcore/mydata/v2/weights/lightning/model.ckpt",
)
# 画像を出力するルートフォルダ
outpath = './output'
# 出力パスが上書きされないように日付と時刻名でフォルダを作成する
now = datetime.now()
formatted_datetime = now.strftime("%Y%m%d_%H%M%S")
result_path = os.path.join( outpath, formatted_datetime)
# 出力フォルダを作成
os.makedirs( result_path, exist_ok=True)
i = 0
for res in predictions:
# 出力画像のファイル名を作成
filename = res['image_path'][0]
basename = os.path.basename( filename )
file, _ = os.path.splitext( basename )
original_file = os.path.join( result_path, file + "_org_" + str(i) + ".png")
anomaly_file = os.path.join( result_path, file + "_ano_" + str(i) + ".png")
# 推論に使用した画像の出力(変形の適用後)
to_pil_image = transforms.ToPILImage()
image_array = res['image'].to('cpu').squeeze(0)
pil_image = to_pil_image(image_array)
pil_image.save(original_file)
# 異常部分のみを取り出した画像(グレー画像。白に近いほど異常があると推論)
image_array = res['anomaly_maps'].to('cpu').squeeze(0)
pil_image = to_pil_image(image_array)
pil_image.save(anomaly_file)
print( f"Results: {original_file=}, {anomaly_file=}" )
i += 1
結果は tensor で戻りますので、tensor を直接操作すれば何でもありです(多分)。
異常検知画像をヒートマップ画像にする
上記プログラムで anomaly_file に出力する画像がグレー画像になります。ヒートマップ画像(カラー画像)として出力する例を示しておきます。
from anomalib.models import Patchcore
from anomalib.engine import Engine
import matplotlib.pyplot as plt
import numpy as np
import os
from datetime import datetime
import torchvision.transforms as transforms
# 推論処理
predictions = Engine().predict(
# MVTec のデータが datasets 以下に配置されている場合
data_path="datasets/MVTec/bottle/test/broken_large",
model=Patchcore(),
# 学習済みモデルへのパスを指定する
ckpt_path="./results/Patchcore/MVTec/bottle/v1/weights/lightning/model.ckpt",
)
# 画像を出力するルートフォルダ
outpath = './output'
# 出力パスが上書きされないように日付と時刻名でフォルダを作成する
now = datetime.now()
formatted_datetime = now.strftime("%Y%m%d_%H%M%S")
result_path = os.path.join( outpath, formatted_datetime)
# 出力フォルダを作成
os.makedirs( result_path, exist_ok=True)
i = 0
for res in predictions:
# 出力画像のファイル名を作成
filename = res['image_path'][0]
basename = os.path.basename(filename)
file, _ = os.path.splitext(basename)
anomaly_file = os.path.join(result_path, file + "_ano_" + str(i) + ".png")
# 推論に使用した画像の出力(変形の適用後)
to_pil_image = transforms.ToPILImage()
# 異常部分のみを取り出した画像(グレー画像。白に近いほど異常があると推論)
image_array = res['anomaly_maps'].to('cpu').squeeze(0)
pil_image = to_pil_image(image_array)
#pil_image.save(anomaly_file)
# グレー画像をヒートマップに変換
gray_array = np.array(pil_image) # PIL画像をnumpy配列に変換
plt.figure(figsize=(6, 6))
plt.axis('off') # 軸を非表示にする
plt.imshow(gray_array, cmap='jet') # ヒートマップカラーマップを適用 ('jet' は一般的なヒートマップ)
#plt.colorbar() # カラーバーを追加(オプション)
plt.savefig(anomaly_file, bbox_inches='tight', pad_inches=0) # ヒートマップ画像を保存
plt.close()
print( f"Results: {anomaly_file=}" )
i += 1
出力はたとえば下記のようになります。
| グレー画像 | ヒートマップ画像 |
|---|---|
 |
 |
閾値を指定してマスク画像を出力させる
異常と判定する閾値を指定し、その閾値以上の値の画素を白として、それ未満の値は黒とするようなマスク画像を出力する例を示します。
from anomalib.models import Patchcore
from anomalib.engine import Engine
import numpy as np
import os
from datetime import datetime
import torchvision.transforms as transforms
# 推論処理
predictions = Engine().predict(
# MVTec のデータが datasets 以下に配置されている場合
data_path="datasets/MVTec/bottle/test/broken_large",
model=Patchcore(),
# 学習済みモデルへのパスを指定する
ckpt_path="./results/Patchcore/MVTec/bottle/v1/weights/lightning/model.ckpt",
)
# 画像を出力するルートフォルダ
outpath = './output'
# 出力パスが上書きされないように日付と時刻名でフォルダを作成する
now = datetime.now()
formatted_datetime = now.strftime("%Y%m%d_%H%M%S")
result_path = os.path.join( outpath, formatted_datetime)
# 出力フォルダを作成
os.makedirs( result_path, exist_ok=True)
i = 0
for res in predictions:
# 出力画像のファイル名を作成
filename = res['image_path'][0]
basename = os.path.basename(filename)
file, _ = os.path.splitext(basename)
mask_file = os.path.join(result_path, file + "_mask_" + str(i) + ".png")
anomaly_file = os.path.join(result_path, file + "_ano_" + str(i) + ".png")
# 推論に使用した画像の出力(変形の適用後)
to_pil_image = transforms.ToPILImage()
# 異常部分のみを取り出した画像(グレー画像。白に近いほど異常があると推論)
image_array = res['anomaly_maps'].to('cpu').squeeze(0)
pil_image = to_pil_image(image_array)
pil_image.save(anomaly_file)
# 画像データのしきい値以上の領域を白く塗りつぶす(マスク)処理
# 閾値は 0 < th < 1 の範囲で指定
th = 0.5
# image_array を取得
image_array = res['anomaly_maps'].to('cpu').squeeze(0).numpy()
# チャネルを最後に移動 (C, H, W) -> (H, W, C)
image_array = np.transpose(image_array, (1, 2, 0))
# RGB値の判定と更新
binary_image = np.where(np.all(image_array <= th, axis=-1, keepdims=True),
[0, 0, 0],
[255, 255, 255]).astype(np.uint8)
pil_image = to_pil_image(binary_image)
# マスク画像の出力
pil_image.save(mask_file)
print( f"Results: {anomaly_file=}, {mask_file=}" )
i += 1
th で閾値を指定します。mask_file にマスク画像が出力されます。
MVTec の bottle_large の test データにある 000.png を anomalib pred として推論させた結果が上のようになるのに対して、上記のプログラムで th=0.5 として出力したマスク画像は下記のようになります。
anomalib コマンドで出力した画像の右から二番目の mask と一致していることがわかります。
複数閾値で異なるマスク画像を出力する
閾値を複数指定して、どの閾値が最適か見たいときは例えば下記のようにします。
from anomalib.models import Patchcore
from anomalib.engine import Engine
import numpy as np
import os
from datetime import datetime
import torchvision.transforms as transforms
# 推論処理
predictions = Engine().predict(
# MVTec のデータが datasets 以下に配置されている場合
data_path="datasets/MVTec/bottle/test/broken_large",
model=Patchcore(),
# 学習済みモデルへのパスを指定する
ckpt_path="./results/Patchcore/MVTec/bottle/v1/weights/lightning/model.ckpt",
)
# 画像を出力するルートフォルダ
outpath = './output'
# 出力パスが上書きされないように日付と時刻名でフォルダを作成する
now = datetime.now()
formatted_datetime = now.strftime("%Y%m%d_%H%M%S")
result_path = os.path.join( outpath, formatted_datetime)
# 出力フォルダを作成
os.makedirs( result_path, exist_ok=True)
i = 0
for res in predictions:
# 出力画像のファイル名を作成
filename = res['image_path'][0]
basename = os.path.basename(filename)
file, _ = os.path.splitext(basename)
anomaly_file = os.path.join(result_path, file + "_ano_" + str(i) + ".png")
# 推論に使用した画像の出力(変形の適用後)
to_pil_image = transforms.ToPILImage()
# 異常部分のみを取り出した画像(グレー画像。白に近いほど異常があると推論)
image_array = res['anomaly_maps'].to('cpu').squeeze(0)
pil_image = to_pil_image(image_array)
pil_image.save(anomaly_file)
# 画像データのしきい値以上の領域を白く塗りつぶす(マスク)処理
# 閾値は 0 < th < 1 の範囲で指定
threshoulds = [0.4, 0.5, 0.6]
for th in threshoulds:
mask_file = os.path.join(result_path, file + "_mask_" + str(i) + "_" + str(th) + ".png")
# image_array を取得
image_array = res['anomaly_maps'].to('cpu').squeeze(0).numpy()
# チャネルを最後に移動 (C, H, W) -> (H, W, C)
image_array = np.transpose(image_array, (1, 2, 0))
# RGB値の判定と更新
binary_image = np.where(np.all(image_array <= th, axis=-1, keepdims=True),
[0, 0, 0],
[255, 255, 255]).astype(np.uint8)
pil_image = to_pil_image(binary_image)
# マスク画像の出力
pil_image.save(mask_file)
print( f"Results: {anomaly_file=}, {mask_file=}" )
i += 1
デフォルトの出力(上記)に対して、threshoulds = [0.4, 0.5, 0.6] としてマスク画像を出力させると下記のようになります。
| 0.4 | 0.5 | 0.6 |
|---|---|---|
 |
 |
 |
閾値が高いほど、マスク画像の白い部分が小さくなるのが分かります。
結論
公式ドキュメント不親切だしバグってるしで、困ったもんです。バージョンアップ前のドキュメントはまだわかりやすかったんですけど。
ちなみに v0.7.0 なら WSL2 なしでも Windows の prompt で動きます。