初めに
今回の記事はSIGNATEの医学論文の自動仕分けチャレンジで使用した医学系のモデルをまとめました。
どのモデルもHuggingfaceから使用できるモデルなので簡単にすぐ使用できます。ざっくりとモデルの概要説明のみ記載しているので詳しい仕組みや学習時のパラメータ設定、モデルのベンチマークなどは論文内の情報を参照してください。
前提知識
まずは、前提として医療系モデルの事前学習に使用されるメジャーなデータセットの解説
・Pubmed
生命科学や生物医学に関する参考文献や要約を提供する検索エンジン。アメリカ国立衛生研究所のアメリカ国立医学図書館(NLM)が情報検索Entrezシステムの一部としてデータベースを運用しています。
・PMC
アメリカ合衆国の国立衛生研究所 (NIH) 内の国立医学図書館 (NLM) の部署である国立生物工学情報センター (NCBI) が運営する、生物医学・生命科学のオンライン論文アーカイブです。
・MIMIC-III
MIMIC-IIIは、2001年から2012年の間にベスイスラエルディーコネス医療センターの救命救急ユニットに滞在した4万人以上の患者に関連する匿名化された健康関連データを含む、大規模で無料で利用できるデータベースです。データベースには、人口統計、重要な情報などの情報が含まれています。
次に用語など
・BLURB(Biomedical Language Understanding and Reasoning Benchmark)
簡単に言うと生物医学に特化したベンチマークを図るためのデータセット。今回使用するモデルはこちらのサイトのベンチマークを参考にしました。
・NAACL
「NAACL」は世界中の研究者によって定期開催される国際会議で、「ACL」「EMNLP」※3と並び、自然言語処理分野(NLP)でもっとも権威ある国際会議のひとつです。
モデル紹介
BioELECTRA(2021):
概要:
NAACL2021で発表されたモデルで、pubmed,PMCの全データで事前学習済みのELECTRA。ELECTRAはBERTが事前学習としてMLMで学習を部分をGANのGeneratorとDiscriminatorで学習するように置き換わっているモデルです。
論文:https://aclanthology.org/2021.bionlp-1.16.pdf
事前学習済みモデル:https://huggingface.co/kamalkraj/bioelectra-base-discriminator-pubmed-pmc-lt
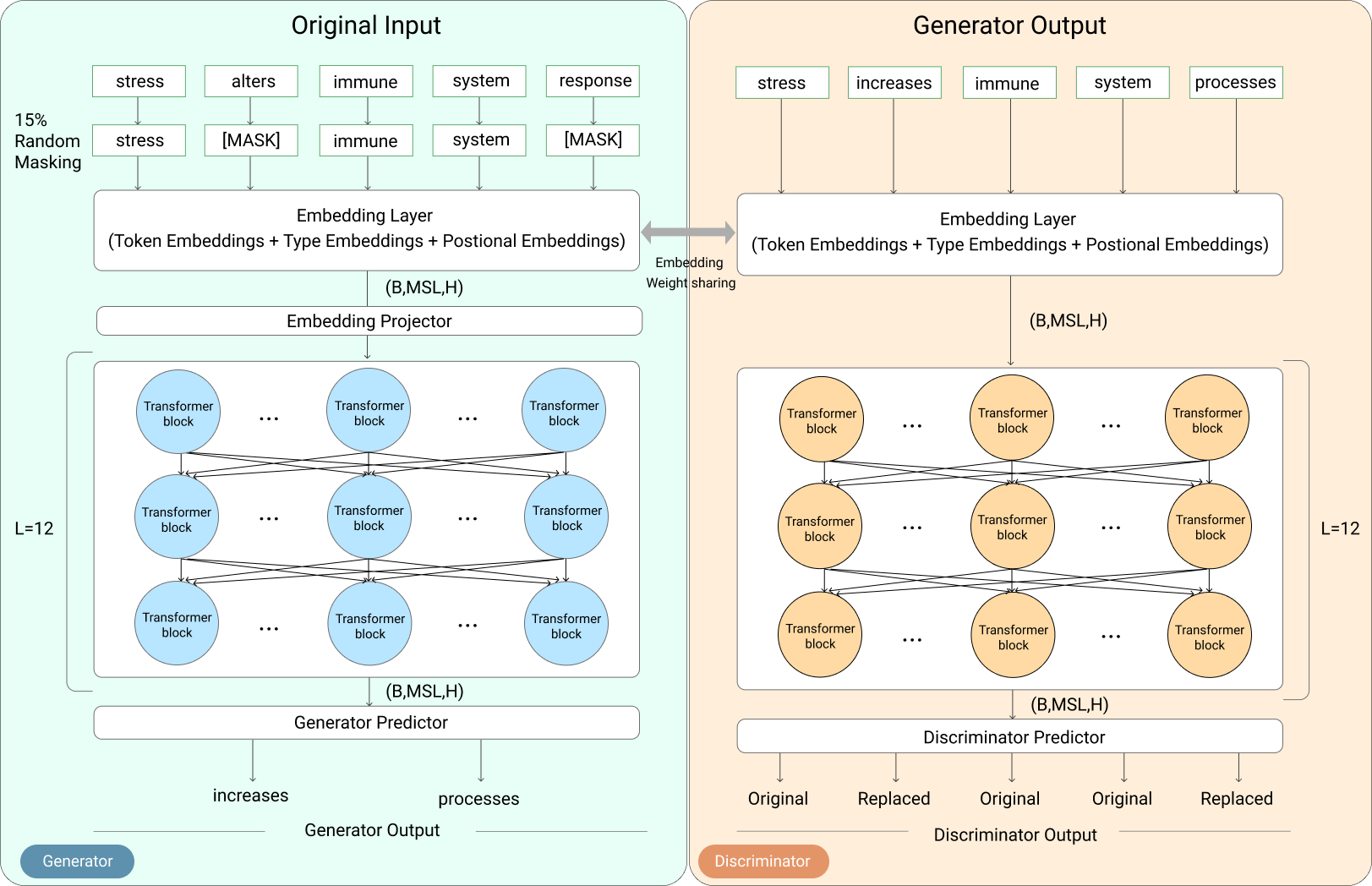
図:ERECTRAの事前学習
SapBERT(2021):
概要:
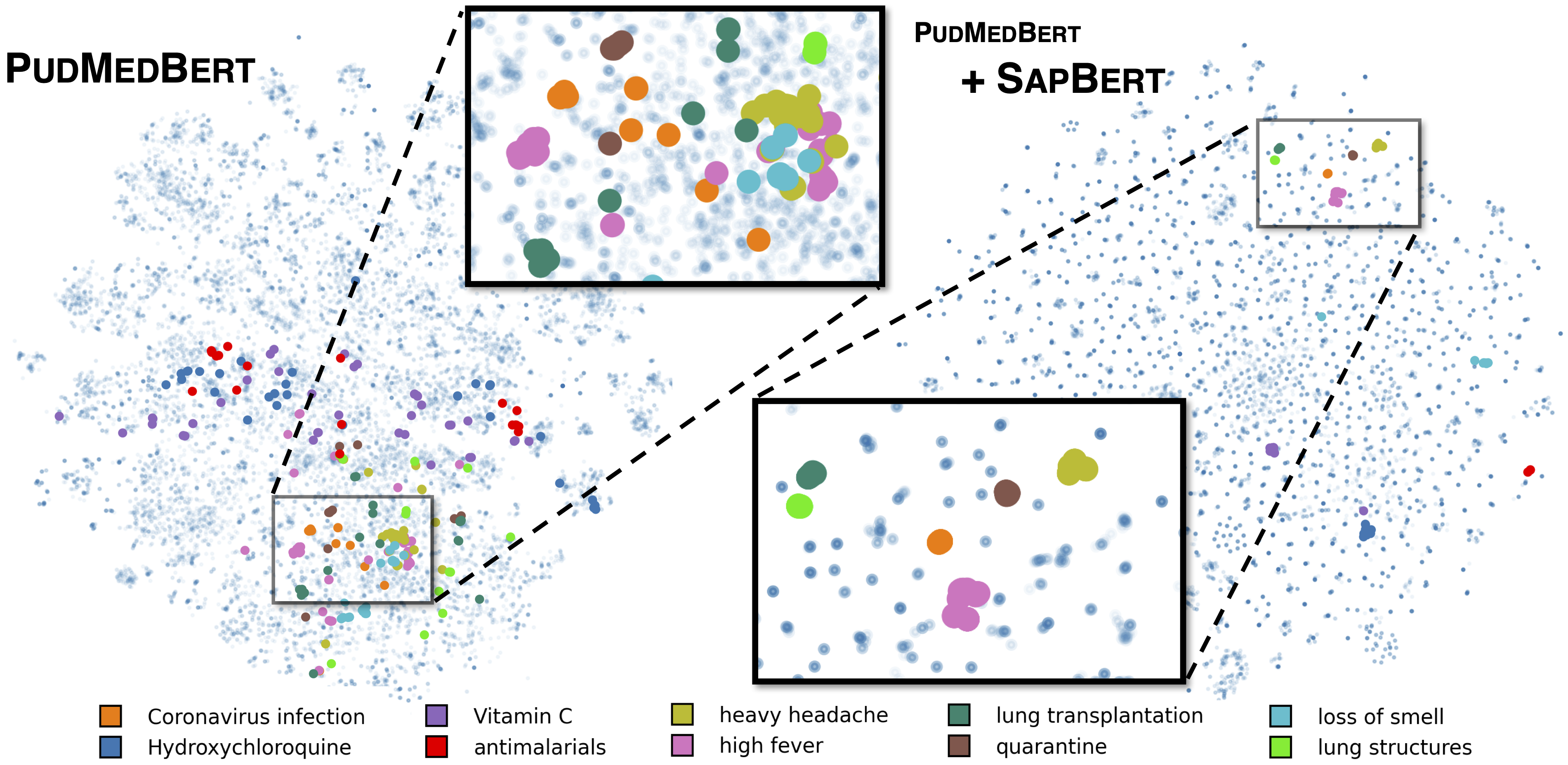
SapBERTはNAACL2021で発表された事前学習モデルの改善手法です。これは少し特殊でモデルではなくすでに事前学習済みのモデルをさらに改善するための手法が提案されています。Umlsデータセットをもとに同音異義語や単語の表記ゆれなどを改善することにより精度改善につながったようです。今回のコンペではSapBERTを適用したPubmedBERTがCV、PublicLBともにダントツで優秀でした。(LBが微妙だったため提出できなかったのが悔やまれる)
論文:https://arxiv.org/abs/2010.11784
事前学習済みモデル:https://huggingface.co/cambridgeltl/SapBERT-from-PubMedBERT-fulltext
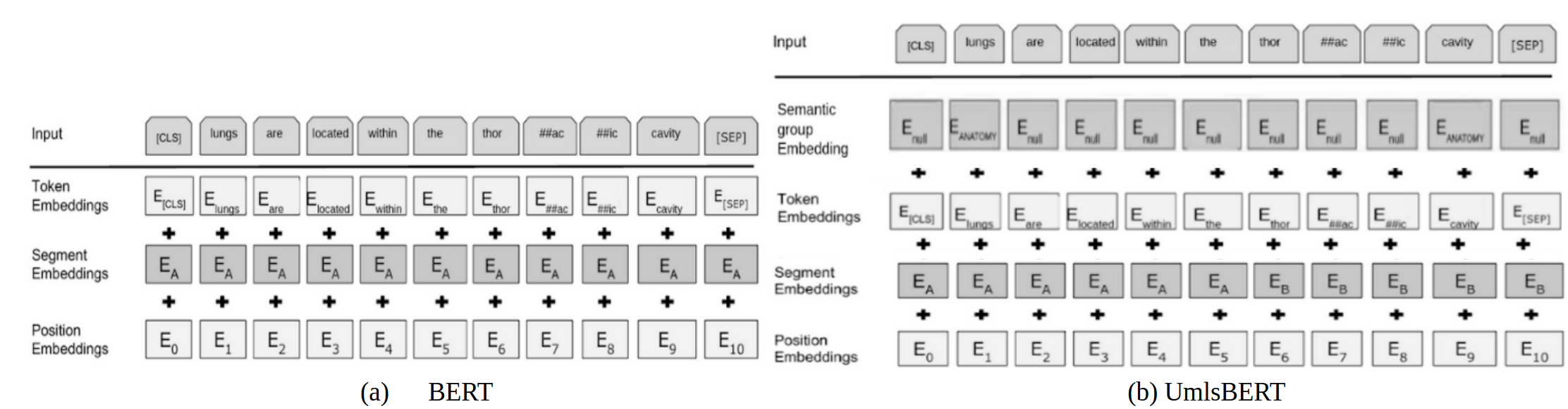
Umls BERT (2021):
概要:
NAACL2021で発表されたモデルで、BioBERT, Clinical BERTの発展形モデル。論文内既存のモデルたちは固有コーパスによって専門用語を学習することには成功したが、医者が持っているようなドメイン知識は獲得できていないと述べています。そのためUMLSを導入することにより、単語間の関係性も学習することが可能になることで、既存モデルを超える精度を実現しています。
論文:https://arxiv.org/pdf/2010.10391.pdf
事前学習済みモデル:https://github.com/gmichalo/UmlsBERT
RoBERTa-base-PM(2020)
概要:
EMNLP2020で発表されたモデルで、Pubmed,PMC,MIMIC-IIIなどのデータセットを事前学習済みのRoBERTaモデル。
Base,distil,largeの3パターンと学習データセットごとにそれぞれモデルが用意されています。
論文:https://aclanthology.org/2020.clinicalnlp-1.17.pdf
事前学習済みモデル:https://github.com/facebookresearch/bio-lm