はじめに
マテリアルズインフォマティクスの研究をしていると、jupyterlabで結晶構造を可視化したいことがよくあります。適当にグラフに原子をプロットすることも可能ですが、マウスでインタラクティブに動かしながら、様々な角度から見ることができれば便利です。
今回は結晶構造の可視化を簡単に行える、crystal-toolkitを紹介します。
インストール
pip install crystal-toolkit
jupyterlabのバージョンは3以上にしてください。2だと追加でextensionをインストールする必要があります。
結晶構造を可視化してみる
jupyterlabで以下を実行することで、簡単に可視化可能です。
from crystal_toolkit.renderables import StructureGraph
from pymatgen.analysis.local_env import MinimumDistanceNN
from pymatgen.core import Structure
struct = Structure.from_file("ZnO.cif")
env = MinimumDistanceNN()
graph = StructureGraph.with_local_env_strategy(struct, env)
graph
これを実行すると、出力部分に以下のような画像が表示されます。
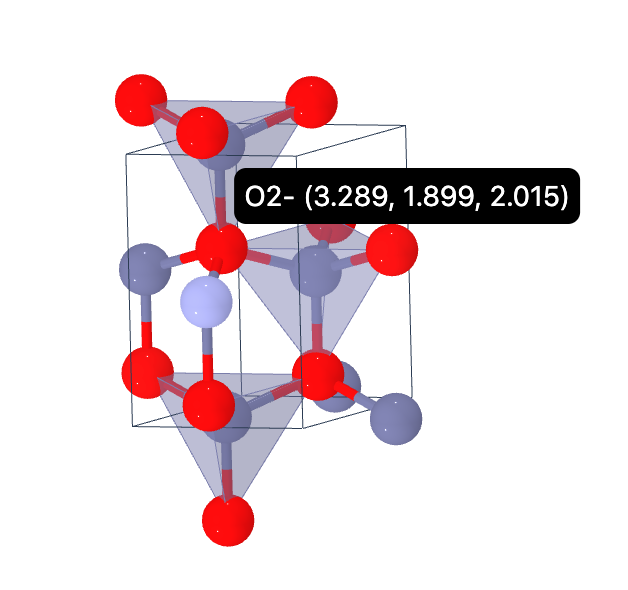
マウスで結晶を回転させることができ、原子にカーソルを合わせると座標が表示されます。
コードの説明
from crystal_toolkit.renderables import StructureGraph
この部分ではcrystal_toolkitのStructureGraphをインポートしているように見えますが、実はpymatgenのStructureGraphをインポートしています。その上で、StructureGraphにget_scene()というメソッドを追加して返しています。このget_scene()により、jupyterでインタラクティブなオブジェクトを表示することが可能になっています。
あとはこれをインスタンス化すればよく、Structureオブジェクトと、結合を判断するためのクラスMinimumDistanceNNを渡すだけです。結合判断のクラスは他のもの(CrystalNNなど)でも問題ありません。
見た目を変える
結晶の見た目を変えることも可能です。上記のように、get_scene()をハックすれば良さそうです。ソースコードを見ると、
StructureGraph.get_scene = get_structure_graph_scene
となっています。get_structure_graph_sceneを変更してみましょう。
from crystal_toolkit.core.legend import Legend
from crystal_toolkit.renderables.structuregraph import get_structure_graph_scene
StructureGraph.get_scene = lambda x: get_structure_graph_scene(
x,
bond_radius=0.2,
legend=Legend(struct, color_scheme="VESTA")
)
graph = StructureGraph.with_local_env_strategy(struct, MinimumDistanceNN())
graph
get_structure_graph_sceneの引数を変えてみました。bondを少し太くして、VESTAと同じ原子の色を用いて描画します。

見た目を変えることができました。より細かい調整をする場合はSceneオブジェクトを自分で作る必要があります。
まとめ
crystal-toolkitを使ってjupyterlabで簡単に結晶構造を可視化することができました。これの他にも分子や相図など、Materials Projectのページのような見た目で表示することが可能なようです。
これらも便利なものがあれば紹介したいと思います。