主成分分析などでのプロットを見せられて、「この2つのデータの分布は大きく違わない」という説明を受けて「ホントか?」と思う機会があったので、そうじゃないデータを作ってみました。
データの作成
x, y, z の3次元からなる2種類のデータ data1 と data2 を次のようにして作成します。
import numpy as np
x = np.random.normal(50,10, size=(500))
y = np.random.normal(50,10, size=(500))
z = np.random.normal(50, 1, size=(500))
x1 = x[np.where(z > 51, True, False)]
y1 = y[np.where(z > 51, True, False)]
z1 = z[np.where(z > 51, True, False)]
x2 = x[np.where(z < 49, True, False)]
y2 = y[np.where(z < 49, True, False)]
z2 = z[np.where(z < 49, True, False)]
data1 = np.concatenate([x1, y1, z1]).reshape(3, (len(x1))).transpose(1, 0)
data2 = np.concatenate([x2, y2, z2]).reshape(3, (len(x2))).transpose(1, 0)
data = np.concatenate([data1, data2])
colors = ["red" if i >len(data1) else "blue" for i in range(len(data1)+len(data2))]
主成分分析
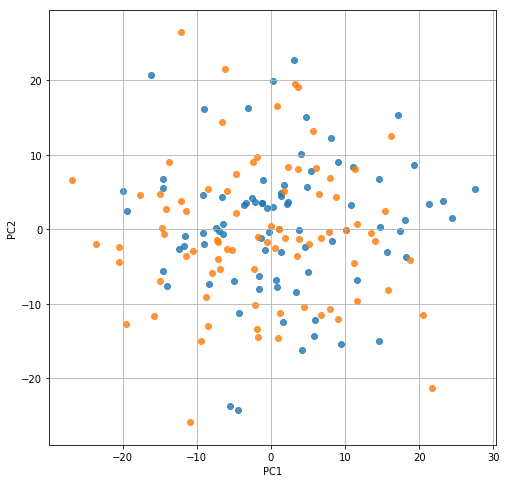
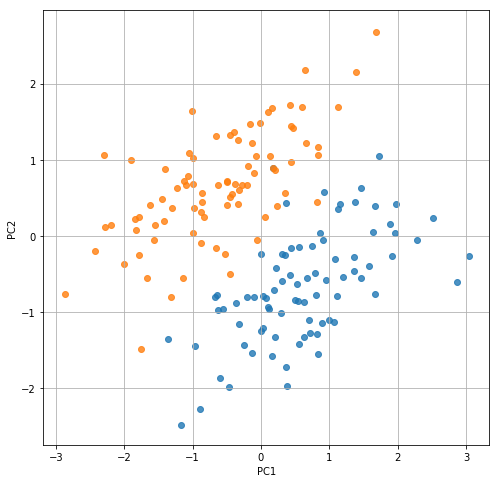
主成分分析して、「ほうら、この2つのデータセットは、分布が大きく違わないでしょう?」(ドヤ顔)
%matplotlib inline
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA #主成分分析器
# 主成分分析の実行
pca = PCA()
pca.fit(data)
# データを主成分空間に写像 = 次元圧縮
feature = pca.transform(data)
# 第一主成分と第二主成分でプロットする
plt.figure(figsize=(8, 8))
plt.scatter(feature[:len(data1), 0], feature[:len(data1), 1], alpha=0.8)
plt.scatter(feature[len(data1):, 0], feature[len(data1):, 1], alpha=0.8)
plt.xlabel('PC1')
plt.ylabel('PC2')
plt.grid()
plt.show()
累積寄与率
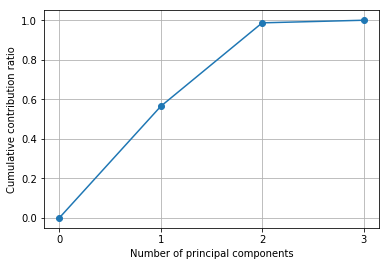
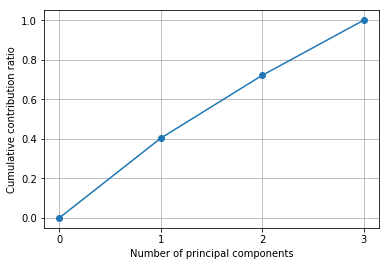
「なんなら、累積寄与率も見せちゃいますよ。第二主成分(PC2)までで累積寄与率ほぼ100%でしょう?」(ドヤ顔)
# 累積寄与率を図示する
import matplotlib.ticker as ticker
import numpy as np
plt.gca().get_xaxis().set_major_locator(ticker.MaxNLocator(integer=True))
plt.plot([0] + list( np.cumsum(pca.explained_variance_ratio_)), "-o")
plt.xlabel("Number of principal components")
plt.ylabel("Cumulative contribution ratio")
plt.grid()
plt.show()
散布図行列
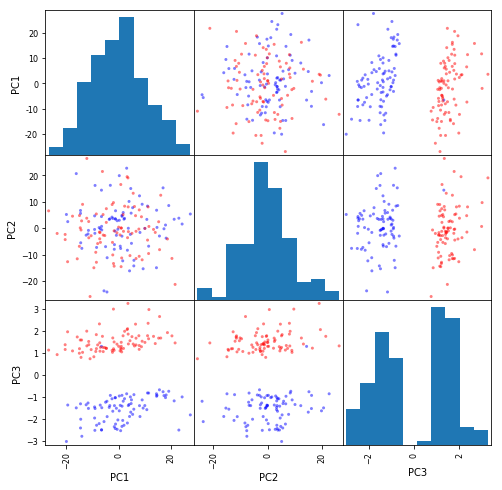
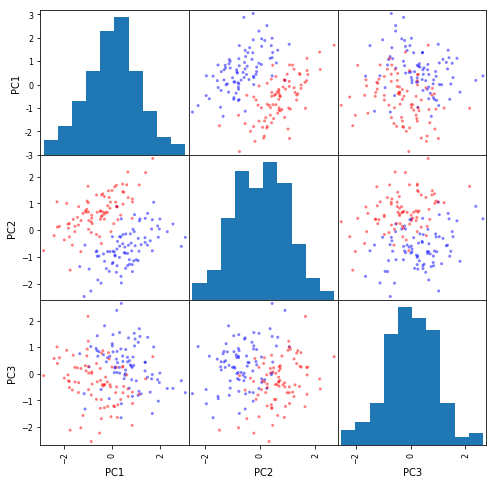
「散布図行列も見せてもらえませんか?」
(ドッキーン!!)
import pandas as pd
from pandas.tools import plotting
plotting.scatter_matrix(pd.DataFrame(feature, columns=['PC1', 'PC2', 'PC3']), figsize=(8, 8), color=colors)
plt.show()
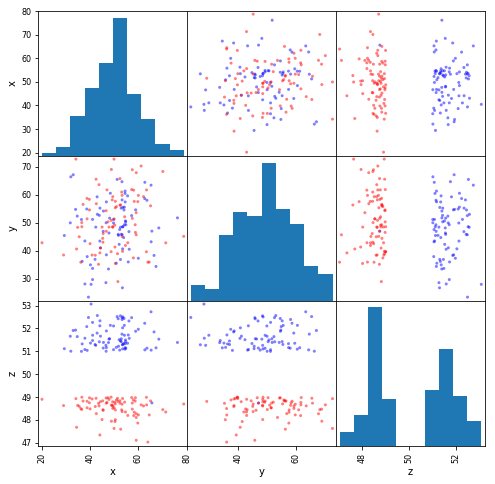
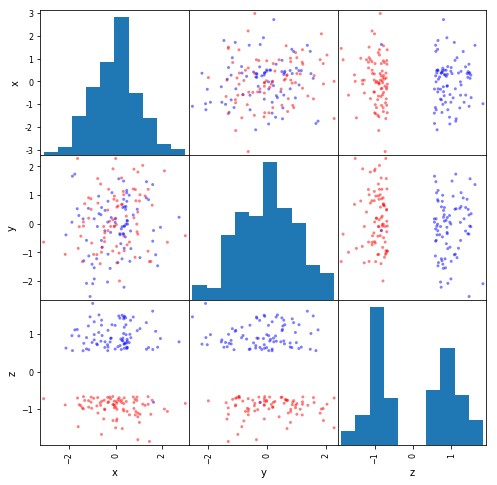
「ぜんぜん別物じゃあないかーッ!! 元データの散布図行列も見せろーッ!!」
「すすすすみませーん」
import pandas as pd
from pandas.tools import plotting
plotting.scatter_matrix(pd.DataFrame(data, columns=['x', 'y', 'z']), figsize=(8, 8), color=colors)
plt.show()
データの標準化
データは多角的に眺めなければいけませんね。その方法の一つがデータの標準化です。それをするだけでデータの見え方がだいぶ変わります。
import numpy as np
def zscore(x, axis = None):
xmean = x.mean(axis=axis, keepdims=True)
xstd = np.std(x, axis=axis, keepdims=True)
zscore = (x-xmean)/xstd
return zscore
%matplotlib inline
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA #主成分分析器
# 主成分分析の実行
pca = PCA()
pca.fit(zscore(data, axis=0))
# データを主成分空間に写像 = 次元圧縮
feature = pca.transform(zscore(data, axis=0))
# 第一主成分と第二主成分でプロットする
plt.figure(figsize=(8, 8))
plt.scatter(feature[:len(data1), 0], feature[:len(data1), 1], alpha=0.8)
plt.scatter(feature[len(data1):, 0], feature[len(data1):, 1], alpha=0.8)
plt.xlabel('PC1')
plt.ylabel('PC2')
plt.grid()
plt.show()
# 累積寄与率を図示する
import matplotlib.ticker as ticker
import numpy as np
plt.gca().get_xaxis().set_major_locator(ticker.MaxNLocator(integer=True))
plt.plot([0] + list( np.cumsum(pca.explained_variance_ratio_)), "-o")
plt.xlabel("Number of principal components")
plt.ylabel("Cumulative contribution ratio")
plt.grid()
plt.show()
import pandas as pd
from pandas.tools import plotting
plotting.scatter_matrix(pd.DataFrame(feature, columns=['PC1', 'PC2', 'PC3']), figsize=(8, 8), color=colors)
plt.show()
import pandas as pd
from pandas.tools import plotting
plotting.scatter_matrix(pd.DataFrame(zscore(data, axis=0), columns=['x', 'y', 'z']), figsize=(8, 8), color=colors)
plt.show()
/Users/kot/miniconda3/envs/py3new/lib/python3.6/site-packages/ipykernel_launcher.py:3: FutureWarning: 'pandas.tools.plotting.scatter_matrix' is deprecated, import 'pandas.plotting.scatter_matrix' instead.
This is separate from the ipykernel package so we can avoid doing imports until