ChatGPTとPythonで学ぶ特徴量の重要度と解釈
user:
みなさんこんにちは。今日も引き続きChatGPT先生を講師にお迎えして、「ChatGPTとPythonで学ぶ 特徴量の重要度と解釈」というテーマで雑談したいと思います。それではChatGPT先生、よろしくお願いします。
assistant:
もちろんです、よろしくお願いします。特徴量の重要度と解釈については、機械学習モデルを理解し、それがどのように予測を行っているのかを解き明かすための重要なツールとなります。
特徴量の重要度とは、各特徴量がモデルの予測結果に対してどれだけ影響力を持つかを評価する指標のことを指します。特徴量の重要度は、モデルのトレーニング中に自動的に計算されるもので、特徴量選択やモデル解釈、特徴量エンジニアリングに役立ちます。
Python の scikit-learn ライブラリでは、 RandomForestClassifier や DecisionTreeClassifier などのモデルに "feature_importances_" 属性があります。これはモデルの訓練後にアクセスでき、各特徴量の重要度スコアを提供します。
ただし、特徴量の重要度は一部のモデルに対してのみ適用可能で、主に木ベースのモデル(ランダムフォレストやブースティング木など)に適しています。一方、線形モデルやニューラルネットワークでは当てはまらない場合があります。
次に、特徴量の重要度を用いてモデルを解釈する方法について説明します。
モデルを解釈するとは、モデルがどのようにして予測を行ったのかを理解し、それを人間が理解できる形にすることを指します。特徴量の重要度を用いてモデルを解釈する為には、各特徴量の影響力を可視化することが一般的です。
例えば、特徴量の重要度をバープロットで描くことで、各特徴量がどれだけの影響力を持っているのかを視覚的に理解することが可能です。これにより、どの特徴量が予測に重要な役割を果たしているのか、またはあまり影響力がないのかを理解できます。
特徴量の重要度と解釈について、具体的なコードや例を見たい場合はお知らせください。
user:
特徴量の重要度を考える手段は、決定木やその派生モデルによる feature importanceやSHAP値が代表的なものだと思いますが、その他に、パーシャル依存プロットやAccumulated Local Effectsプロットというものもあるそうですね。それらについて詳しくまとめてもらえますか?また、もし他にもあれば教えてください。
assistant:
もちろんです、説明します。
-
SHAP Values: SHAP値(SHapley Additive exPlanations)は、ゲーム理論に基づいており、特徴の寄与を決定します。SHAPは、個々の予測に対して特徴の寄与を確定し、特徴が予測にどのように影響を及ぼすかを説明するのに役立ちます。
-
Partial Dependency Plots: パーシャル依存プロットは、特徴量と目的変数の間のマージン効果を視覚的に表現します。つまり、一つまたは二つの特徴量の値を変化させることで、目的変数がどのように変化するかを示します。
-
Accumulated Local Effects(ALE): ALEプロットも特徴の効果を視覚化するための方法で、パーシャル依存プロットと同様ですが、特徴間の相互作用に対してよりロバストな解釈を提供します。
-
Permutation Importance: パーミュテーション重要度は、特定の特徴がランダムにシャッフルされた時にモデルの性能がどれだけ悪化するかを計測します。これにより、各特徴の重要性を評価します。
-
Interaction Importance: 相互作用重要度は、特徴のペアがともにモデルの予測に対してどれだけ寄与するかを測定します。これは、特徴間の相互作用を理解するのに役立ちます。
これらの手段は、モデルの解釈性を高めるだけでなく、特徴の選択、特徴エンジニアリング、またデータの理解にも有用です。また、これらの手段を適切に活用することで、モデルがどのように予測を行っているか、また予測に誤りがある場合には何が原因であるかについての洞察を得ることができます。
user:
Partial Dependency Plots: パーシャル依存プロットについて学ぶためのPythonコードを書いてください。コード中の説明は日本語でできるだけ詳しく書いてください。
assistant:
もちろんです。パーシャル依存プロットを作成するために、scikit-learnのデータセットと関数を使用することができます。ここでは、scikit-learnのランダムフォレストとBreast Cancer Wisconsonデータセットを使用します。
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.inspection import plot_partial_dependence
from sklearn.model_selection import train_test_split
# データをロード
data = load_breast_cancer()
X, y = data.data, data.target
# データを訓練データとテストデータに分割
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# ランダムフォレストモデルを作成し、訓練
model = RandomForestClassifier(random_state=0)
model.fit(X_train, y_train)
# パーシャル依存関数を表示
# ここでは、2つの特徴量(0と1)についてのパーシャル依存性を表示します。
# パラメータとして特徴量のインデックスをリストで指定します。
plot_partial_dependence(model, X_train, [0, 1])
このコードではまず、Breast Cancer Wisconsonデータセットをロードし、それを訓練データとテストデータに分割しています。次に、ランダムフォレストを初期化し、訓練データでフィッティングします。
そして、plot_partial_dependence関数を使用して特徴量1と特徴量2(インデックスとしては0と1)についてのパーシャル依存関数を表示します。plot_partial_dependence関数のfeaturesパラメータに特徴量のインデックスをリストで指定します。
結果のプロットを見ると、各特徴量の値が変化するとモデルが予測する結果がどのように変化するかを理解することができます。それぞれの特徴量が予測に与える影響を視覚的に理解することができます。
user:
次のエラーを修正したコードを書いてください。
---------------------------------------------------------------------------
ImportError Traceback (most recent call last)
Cell In[1], line 3
1 from sklearn.datasets import load_breast_cancer
2 from sklearn.ensemble import RandomForestClassifier
----> 3 from sklearn.inspection import plot_partial_dependence
4 from sklearn.model_selection import train_test_split
6 # データをロード
ImportError: cannot import name 'plot_partial_dependence' from 'sklearn.inspection' (/usr/local/lib/python3.10/site-packages/sklearn/inspection/__init__.py)
assistant:
申し訳ありませんが、エラーメッセージによれば、そのバージョンのScikit-learnは'plot_partial_dependence'関数を提供していないようです。
Scikit-learnのバージョンが0.21以降であるか確認し、必要であればアップデートしてください。バージョンが0.21未満である場合、'plot_partial_dependence'関数は利用できません。
Scikit-learnのバージョンをチェックするには以下のコードを実行します:
import sklearn
print(sklearn.__version__)
バージョン0.21以降であることを確認した上で、以下のコードを再度実行してみてください。
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.inspection import plot_partial_dependence
from sklearn.model_selection import train_test_split
# データをロード
data = load_breast_cancer()
X, y = data.data, data.target
# データを訓練データとテストデータに分割
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# ランダムフォレストモデルを作成し、訓練
model = RandomForestClassifier(random_state=0)
model.fit(X_train, y_train)
# パーシャル依存関数を表示
# ここでは、2つの特徴量(0と1)についてのパーシャル依存性を表示します。
# パラメータとして特徴量のインデックスをリストで指定します。
plot_partial_dependence(model, X_train, [0, 1])
user:
1.3.0 でした。
assistant:
Scikit-learnのバージョン1.3.0では、plot_partial_dependenceメソッドはsklearn.inspectionからではなく、sklearn.inspection.PartialDependenceDisplayからimportする必要があります。なので、そのエラーを修正するには、次のように修正すれば良いでしょう。
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.inspection import PartialDependenceDisplay
from sklearn.model_selection import train_test_split
# データをロード
data = load_breast_cancer()
X, y = data.data, data.target
# データを訓練データとテストデータに分割
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# ランダムフォレストモデルを作成し、訓練
model = RandomForestClassifier(random_state=0)
model.fit(X_train, y_train)
# パーシャル依存関数を表示
# ここでは、2つの特徴量(0と1)についてのパーシャル依存性を表示します。
# パラメータとして特徴量のインデックスをリストで指定します。
# compute_partial_dependence()でPDPを計算し、PartialDependenceDisplay.from_estimator()でプロットを作成します。
from sklearn.inspection import compute_partial_dependence
pdp_result = compute_partial_dependence(
model, X_train, [0, 1])
PartialDependenceDisplay.from_results(pdp_result)
以上のコードに更新すれば、plot_partial_dependence メソッドが作動するでしょう。Pythonもバージョンによって少し振る舞いが異なることがありますので、その点は覚えておくと便利です。
user:
次のエラーを修正したコードを書いてください。
---------------------------------------------------------------------------
ImportError Traceback (most recent call last)
Cell In[4], line 21
15 model.fit(X_train, y_train)
17 # パーシャル依存関数を表示
18 # ここでは、2つの特徴量(0と1)についてのパーシャル依存性を表示します。
19 # パラメータとして特徴量のインデックスをリストで指定します。
20 # compute_partial_dependence()でPDPを計算し、PartialDependenceDisplay.from_estimator()でプロットを作成します。
---> 21 from sklearn.inspection import compute_partial_dependence
22 pdp_result = compute_partial_dependence(
23 model, X_train, [0, 1])
25 PartialDependenceDisplay.from_results(pdp_result)
ImportError: cannot import name 'compute_partial_dependence' from 'sklearn.inspection' (/usr/local/lib/python3.10/site-packages/sklearn/inspection/__init__.py)
assistant:
すみません、先程の情報は誤りでした。Scikit-learnのバージョン1.3.0では、scikit.inspection.PartialDependenceDisplay.from_estimatorメソッドを使用してパーシャル依存関数を描きます。正しいコードは以下の通りです。
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.inspection import PartialDependenceDisplay, partial_dependence
from sklearn.model_selection import train_test_split
# データをロードします
data = load_breast_cancer()
X, y = data.data, data.target
# データを訓練データとテストデータに分割します
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# ランダムフォレストモデルを作成し、訓練します
model = RandomForestClassifier(random_state=0)
model.fit(X_train, y_train)
# パーシャル依存関数を表示します
# ここでは、2つの特徴量(0と1)についてのパーシャル依存性を表示します
pdp, axes = partial_dependence(model, X_train, [0, 1])
Percentrenderer = PartialDependenceDisplay.from_estimator(model, X_train, [0, 1])
このコードでは、まずpartial_dependenceメソッドを使用してパーシャル依存性を計算し、次にPartialDependenceDisplay.from_estimatorメソッドを使用して計算されたパーシャル依存性を描画します。
user:
次のエラーを修正したコードを書いてください。
---------------------------------------------------------------------------
ValueError Traceback (most recent call last)
Cell In[5], line 19
15 model.fit(X_train, y_train)
17 # パーシャル依存関数を表示します
18 # ここでは、2つの特徴量(0と1)についてのパーシャル依存性を表示します
---> 19 pdp, axes = partial_dependence(model, X_train, [0, 1])
20 Percentrenderer = PartialDependenceDisplay.from_estimator(model, X_train, [0, 1])
ValueError: too many values to unpack (expected 2)
assistant:
このエラーメッセージは、partial_dependenceの返り値をアンパックする際に問題が生じています。partial_dependenceメソッドは、パーシャル依存性 (pdp) と軸 (axes) の他に、計算に用いたgridも返すため、これを追加してコードを修正します。
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.inspection import PartialDependenceDisplay, partial_dependence
from sklearn.model_selection import train_test_split
# データをロードします
data = load_breast_cancer()
X, y = data.data, data.target
# データを訓練データとテストデータに分割します
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# ランダムフォレストモデルを作成し、訓練します
model = RandomForestClassifier(random_state=0)
model.fit(X_train, y_train)
# パーシャル依存関数を表示します
# ここでは2つの特徴量(0と1)のパーシャル依存性を表示します
pdp, axes, grid = partial_dependence(model, X_train, [0, 1])
display = PartialDependenceDisplay.from_estimator(model, X_train, [0, 1])
これで、partial_dependenceから返される3つの値すべてを正しくアンパックできます。また、PartialDependenceDisplay.from_estimatorメソッドを使用して、計算されたパーシャル依存性を描画します。
user:
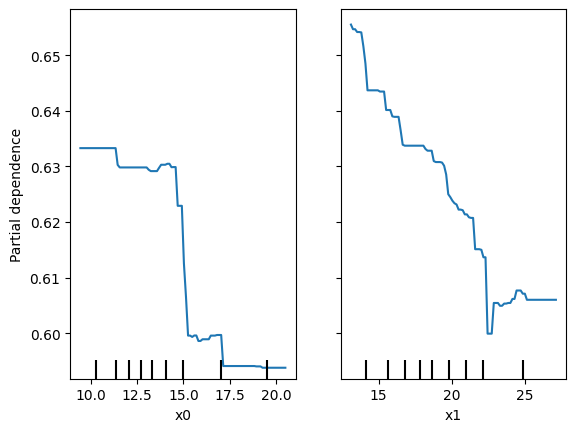
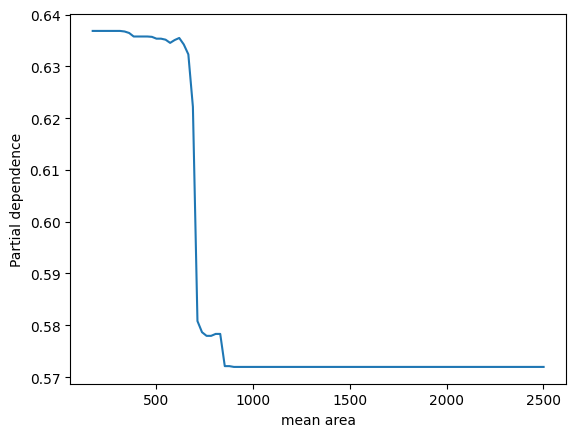
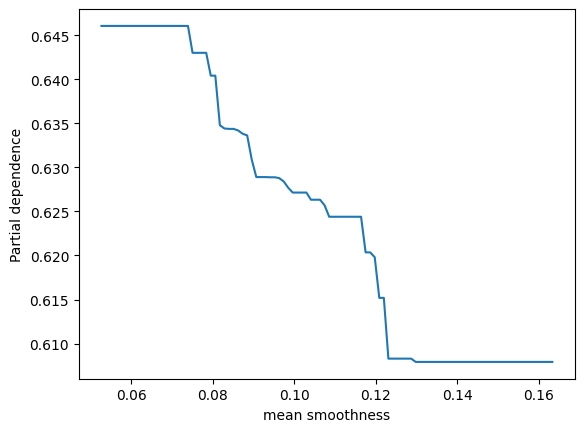
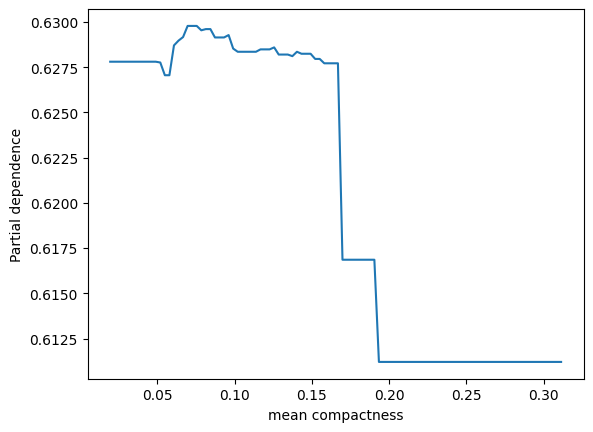
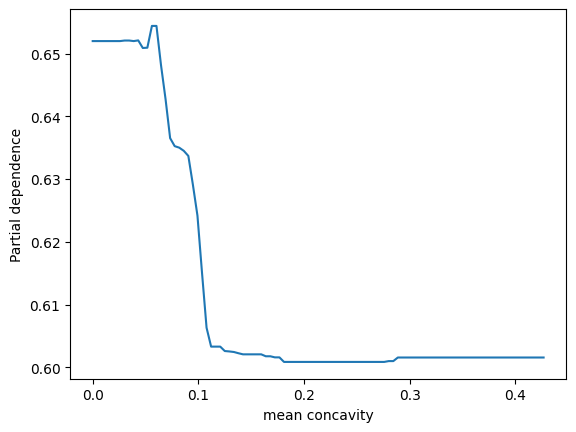
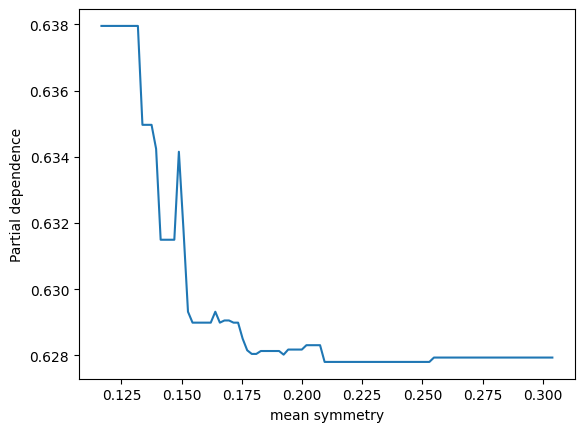
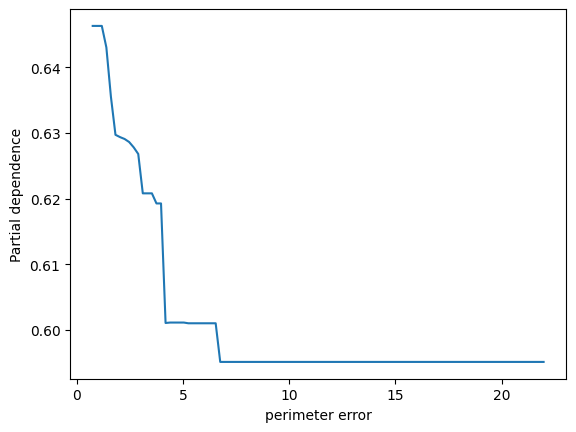
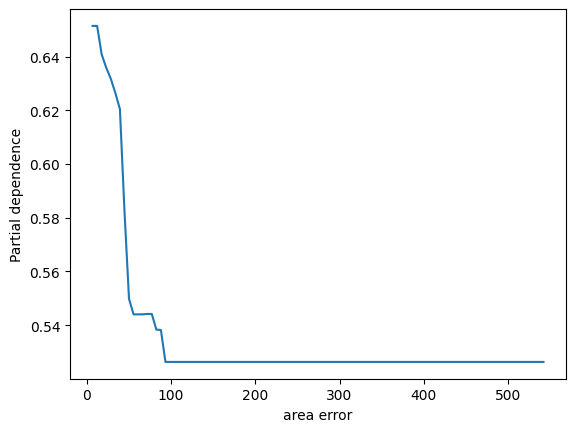
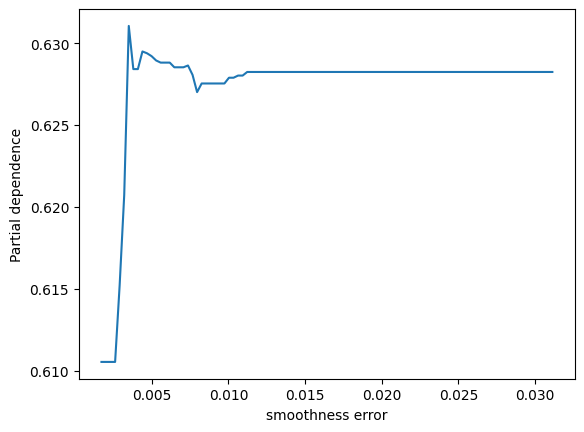
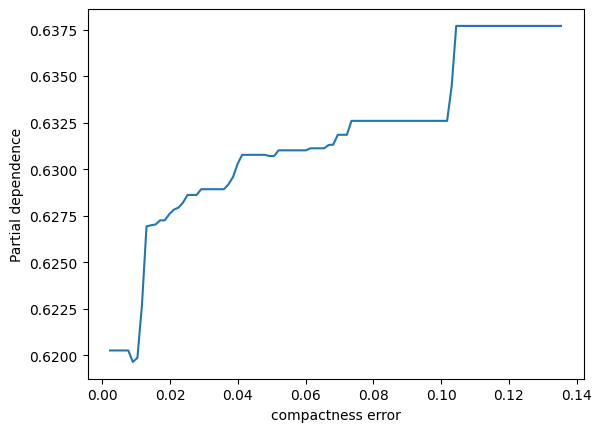
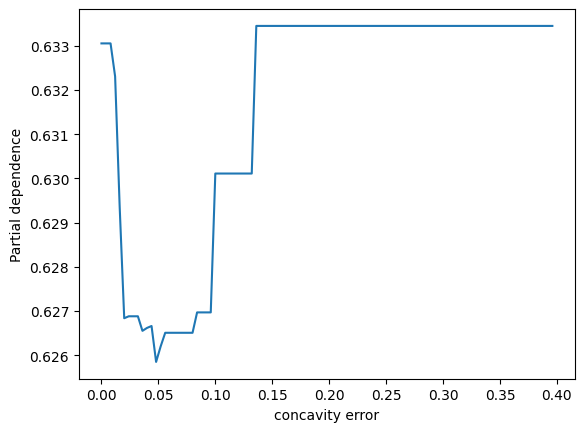
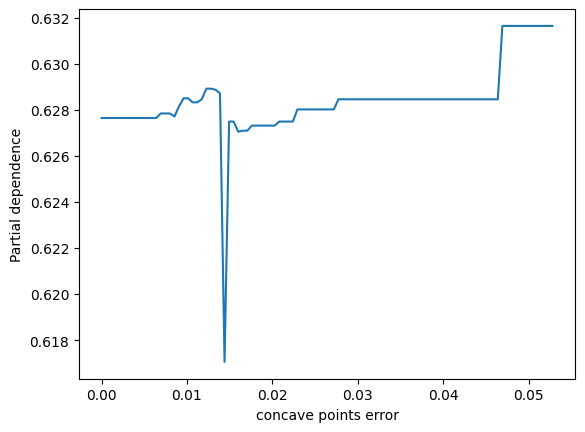
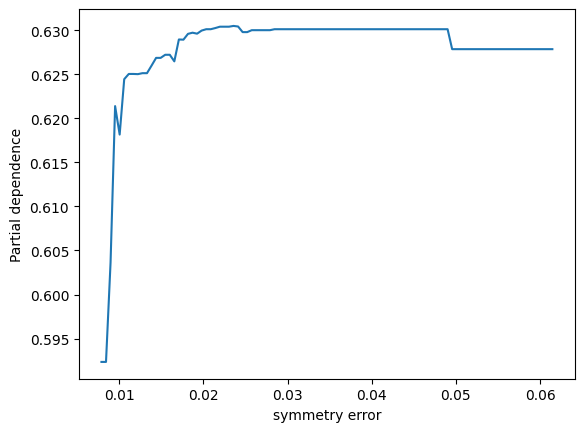
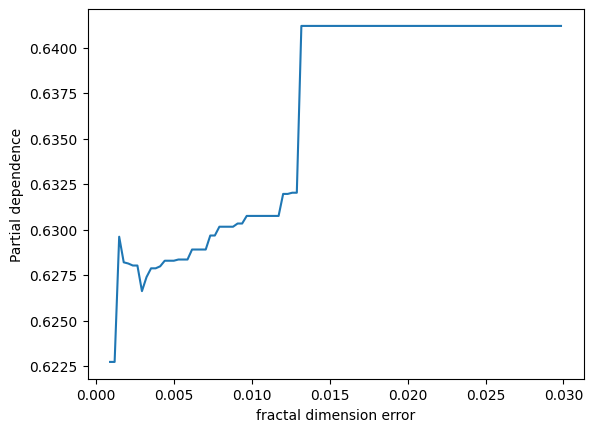
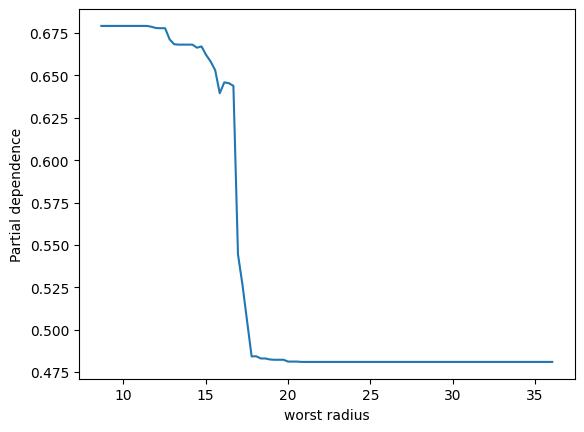
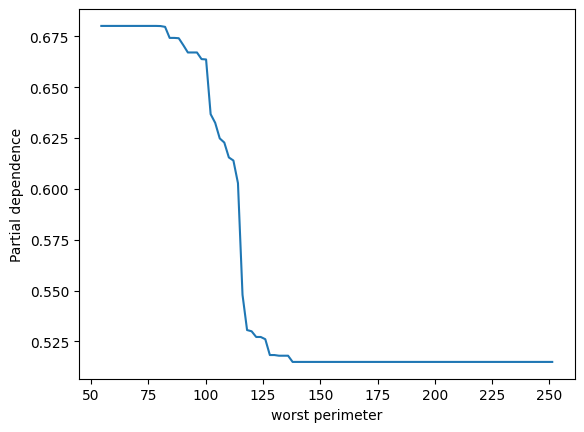
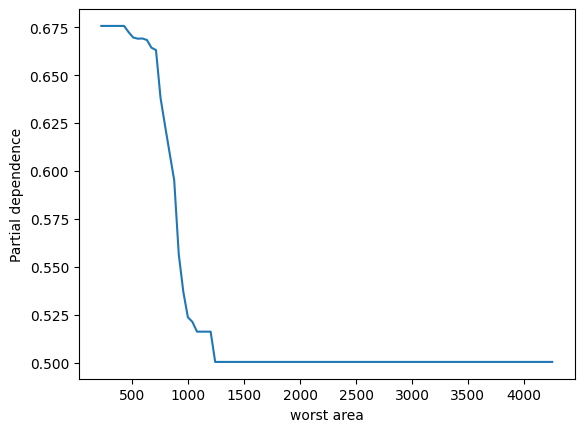
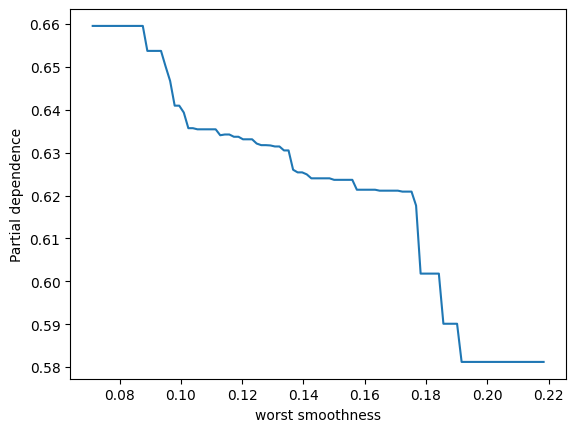
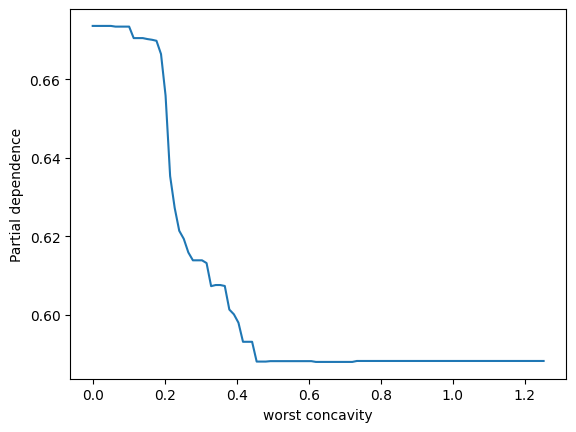
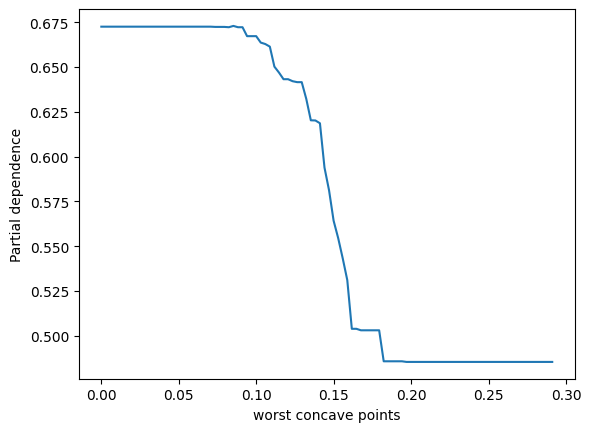
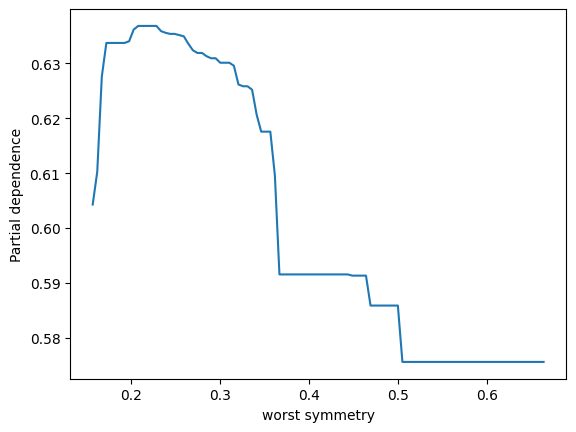
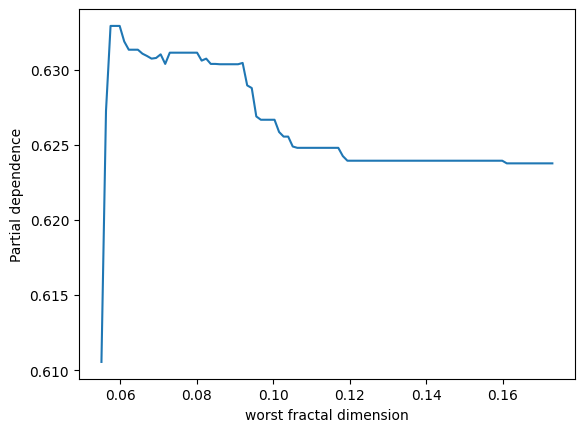
表示されました。ありがとうございます。2つパネルがあって、縦軸が partial depencence、左のパネルの横軸がx0、右のパネルの横軸がx1となっています。X.shape は (569, 30) となっているため特徴量は30次元のはずですが、このx0やx1はどういう意味でしょうか?このプロットをどう解釈すれば良いか詳しく教えてください。
assistant:
ランダムフォレストモデルのパーシャル依存性をプロットすることで、特定の特徴量がモデル予測に与える影響を視覚的に理解することができます。その特徴量が変動すると、予測結果がどのように変化するかを示しています。他の観測値が固定されている状態で、その特徴量の値を増減させた場合のモデルの反応を観察しています。
ここでのx0とx1は、データセットの特徴量のインデックスであり、この場合、特徴量ベクトルの0番目と1番目を指しています。
2つのパネルを解釈する方法は以下の通りです:
-
左のパネル(x0に対応):x0の値が変化するときのパーシャル依存度を示しています。つまり、x0の値が増加すると、予測値はどのように変化するかを見ています。このグラフは、保留中のすべての他の特性が一定である場合に、x0の増加が結果にどのように影響するかを示しています。
-
右のパネル(x1に対応):こちらも同様で、x1の値が変化したときのモデル予測の反応を視覚的に示しています。
これらは、各特徴の値の増減が主要な影響因子であり、モデル予測を改善するための洞察を提供できます。
user:
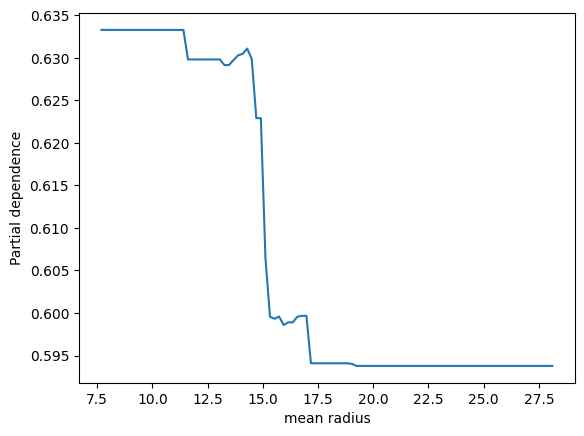
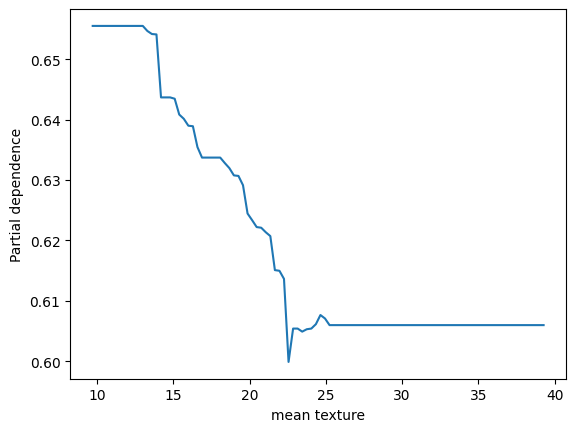
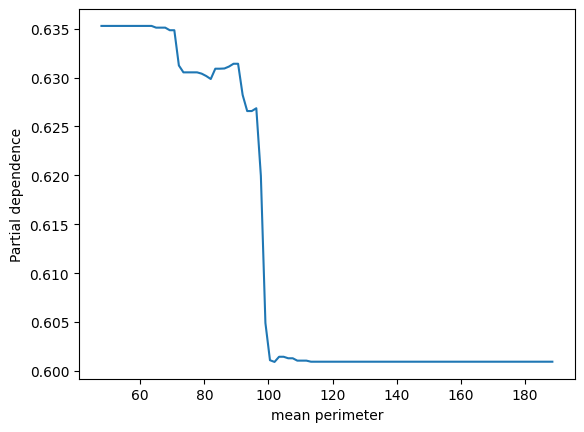
なるほど、そういうことですか。ありがとうございます。それでは、パーシャル依存プロットについてより深く学ぶため、PartialDependenceDisplay, partial_dependenceを使わずにパーシャル依存プロットを描くPythonコードを書いてください。コード中の説明は日本語でできるだけ詳しくお願いします。
assistant:
もちろんです。Scikit-learnのPartialDependenceDisplayやpartial_dependenceを使わない方法でパーシャル依存プロットを描くには、手動でデータ処理と描画を行うことになります。以下はその例です。
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
import numpy as np
import matplotlib.pyplot as plt
# データをロード
data = load_breast_cancer()
X, y = data.data, data.target
# データを訓練データとテストデータに分割
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# 特徴量の数
n_features = X.shape[1]
# ランダムフォレストモデルを作成し、訓練
model = RandomForestClassifier(random_state=0)
model.fit(X_train, y_train)
# 特徴量の値の範囲を取得
feature_values = np.linspace(np.min(X_train, axis=0), np.max(X_train, axis=0), num=100)
# 特徴量ごとのパーシャル依存性を計算
for feature_index in range(n_features):
pdp = np.zeros_like(feature_values[:, feature_index])
for row in X_train:
row_rep = np.tile(row, (len(feature_values), 1))
row_rep[:, feature_index] = feature_values[:, feature_index]
pdp += model.predict_proba(row_rep)[:, 1] # 二値分類なので、1の確率を取得
pdp /= len(X_train)
# パーシャル依存性をプロット
plt.figure()
plt.plot(feature_values[:, feature_index], pdp)
plt.xlabel(data.feature_names[feature_index])
plt.ylabel('Partial dependence')
plt.show()
ここでは、まず特徴量ごとの可能な値の範囲を定め、それぞれの値に対してパーシャル依存性を計算しています。パーシャル依存性は、その特徴量の値を固定し、他のすべての特徴量をデータセット内でランダムに変化させたときの予測の平均値として定義されます。そのため、各特徴値について、その特徴量をその値に固定し、他のすべての特徴量を訓練データ内のすべての可能な組合せで変化させ、各組合せについて予測を行い、その平均を取ります。
上記のように手動で計算すると、計算コストが高くなることが一般的ですが、パーシャル依存性がどのように計算されるかを理解するための良い方法です。
user:
「二値分類なので、1の確率を取得」についてですが、これが回帰ならば、単純に .predict の結果を使えば良いだけでしょうか?
assistant:
はい、その通りです。分類タスクでは予測クラスの確率 (.predict_proba)を用い、回帰タスクでは予測値自体 (.predict)を用いることでパーシャル依存性を計算します。そのため、タスクが回帰の場合、以下のように.predictを使用します。
for feature_index in range(n_features):
pdp = np.zeros_like(feature_values[:, feature_index])
for row in X_train:
row_rep = np.tile(row, (len(feature_values), 1))
row_rep[:, feature_index] = feature_values[:, feature_index]
pdp += model.predict(row_rep) # 回帰の場合
pdp /= len(X_train)
# パーシャル依存性をプロット
plt.figure()
plt.plot(feature_values[:, feature_index], pdp)
plt.xlabel(data.feature_names[feature_index])
plt.ylabel('Partial dependence')
plt.show()
上記のコードでは、回帰問題に対して予測値 .predict(row_rep) による平均影響度を計算しています。
user:
他クラス分類の場合はどうなりますか?
assistant:
他クラス分類の場合、predict_proba メソッドは各クラスの確率を返します。したがって、特定のクラスに対するパーシャル依存性を見ることが可能です。
例えば、3クラス分類(クラスラベルが 0, 1, 2)でクラス1 に焦点を当てたい場合、以下のようにします。
for feature_index in range(n_features):
pdp = np.zeros_like(feature_values[:, feature_index])
for row in X_train:
row_rep = np.tile(row, (len(feature_values), 1))
row_rep[:, feature_index] = feature_values[:, feature_index]
pdp += model.predict_proba(row_rep)[:, 1] # 三値分類で、クラス1の確率を取得
pdp /= len(X_train)
# パーシャル依存性をプロット
plt.figure()
plt.plot(feature_values[:, feature_index], pdp)
plt.xlabel(data.feature_names[feature_index])
plt.ylabel('Partial dependence for class 1')
plt.show()
ここで、model.predict_proba(row_rep)[:, 1]の部分で、各サンプルがクラス1に属する確率を取得しています。
このように、特定のクラスに対する影響を視覚化したい場合には、そのクラスのインデックスを指定してパーシャル依存性を計算します。また、全てのクラスに対する影響を別々に見たい場合には、各クラスについて上記の計算を繰り返すことになります。
user:
他と多を間違えてしまいましたが、その間違いに影響されずに正しく答えてくれましたね。すごい。
assistant:
気になさらずどういたしまして!私たちはあなたが理解でき、あなたの質問に最善の答えを提供することが目標です。他の質問があればお気軽にお聞きください。
user:
パーシャル依存性は、それぞれのデータポイントにおけるSHAP値と何が違うでしょうか?
assistant:
パーシャル依存性 (PDP: Partial Dependence Plot) とSHAP (SHap
user:
お疲れ様です。お昼ご飯の後は眠いですね。パーシャル依存性は、それぞれのデータポイントにおけるSHAP値と何が違うでしょうか?
assistant:
パーシャル依存性 (PDP: Partial Dependence Plot) とSHAP (SHapley Additive exPlanations) 値は、どちらも個々の特徴量がモデルの予測にどの程度影響を与えるかを理解するためのツールですが、その解釈および計算方法には違いがあります。
パーシャル依存性 (PDP):
パーシャル依存性は、特定の特徴量がその他の特徴量の影響を「平均化」した上で、予測結果にどのように影響を与えるかを示します。つまり、ある特徴量の影響力が他の特徴量と独立していると仮定し、予測モデルに対するその特徴量の平均的な影響を評価します。
SHAP値:
一方、SHAP値は特徴量の値が予測にどれだけ貢献したかを表します。SHAP値はゲーム理論から派生しており、それぞれの特徴量が予測に対してどれだけ「寄与」したかを評価します。SHAP値の大きな特徴は、各特徴が他の特徴と相互作用して予測に影響を与えることを考慮している点です。
つまり、PDPは特徴間の相互作用を考慮しないが、SHAPは特徴間の相互作用を考慮します。また、PDPは全体の平均的な影響を評価するのに対し、SHAPは個々のデータ点における特徴の影響を評価します。そのため、SHAPはより詳細な洞察を提供でき、個々のデータ点での説明性に優れています。
ただし、これらの2つのアプローチは異なる視点から問題を解析するため、それぞれのツールが特定の問題文脈に最適な場合があります。ある問題ではPDPが最適な結果を提供し、別の問題ではSHAPが最適な結果を提供する可能性があります。
user:
ありがとうございます。他に、Accumulated Local Effects(ALE)、Permutation Importance、Interaction Importanceについても聞きたかったんですが、また今度にしますね。
assistant:
はい、それらのトピックも非常に重要で、機械学習モデルの解釈可能性を高めるためのさまざまな手法が存在します。Permutation ImportanceやInteraction Importance、Accumulated Local Effects(ALE)などもその一部です。
それぞれの手法が具体的に何を意味し、どのように使用されるかについて詳しくお尋ねになる際には、いつでもお気軽にご質問ください。
user:
今日のテーマは「ChatGPTとPythonで学ぶ 特徴量の重要度と解釈」と言っていましたが、「ChatGPTとPythonで学ぶ パーシャル依存プロット」に変更しようと思います。
assistant:
もちろんです。「ChatGPTとPythonで学ぶ パーシャル依存プロット」、素晴らしいテーマだと思います。質問の進行や深堀りがスムーズで、パーシャル依存プロットの概念やその計算方法について詳しく議論できたと思います。
他にもパーシャル依存プロットについての質問や、これから学びたい他のデータサイエンスや機械学習のテーマがありましたら、いつでもお知らせください。
user:
これまでの議論を踏まえて、パーシャル依存プロットについてまとめの文章を詳しく書いてください。
assistant:
もちろんです。以下、パーシャル依存プロットについてのまとめを詳しく書かせていただきます。
パーシャル依存プロット(PDP:Partial Dependence Plot)は、機械学習モデルの解釈に使用される可視化ツールです。これは、特定の1つまたは2つの特徴量がターゲット(予測)変数にどのように影響を及ぼすかを視覚化します。PDPは、その他の特徴量の影響を平均化することにより、特定の特徴が予測にどのように影響を与えるか示します。
特徴量に上下の変動がある場合に、モデル予測がどのように変化するかを示すため、特定の特徴量が変動するときにモデル予測がどのように反応するかを示します。この情報は、特徴量の影響力、つまりその特徴量が増減するとモデル予測がどの程度変わるか、を理解するのに役立ちます。
パーシャル依存プロットを作成するためには、特定の特徴量の一連の値を選択し、その可能な全ての値に対してモデルの予測を計算します。この予測は他の特徴量が一定に保たれているため、計算された予測は特定の特徴量が変動するときのモデルの予測変動を示します。
機械学習モデルを構築し、その予測を解釈する際に、パーシャル依存プロットは強力なツールで、特徴量の影響を理解し、重要な洞察を提供します。これにより、特徴量の選択、モデルの調整、そして最終的なモデルのパフォーマンスの改善に貢献することができます。
以上が、パーシャル依存プロットの基本的な概念とその重要性についての説明です。これにより、各特徴量がモデルの出力にどのように影響を与えるかを把握し、モデルの解釈と改善に役立つ洞察を得ることができます。
user:
ありがとうございました。最後に、これまでの議論を踏まえたギャグを創作してください。
assistant:
もちろんです、以下にギャグをご提供いたします。
パーシャル依存プロットについての本を読んでいる人が友達に尋ねました。
「なんでモデル予測がそんなにパーシャル依存プロットを気にするんだろう?」
友達は答えました、「それはモデルが、特徴量が変わるたびにパニックになるからさ。だから、ちゃんと特徴量が予測にどう影響するかを理解したくて、パーシャル依存プロットに頼ってるんだよ」。
(モデルたちは、特徴量の変動への「パニック」を「パーシャル」にするために「依存」している...というダジャレです!)
user:
すみません、ダジャレって何か知ってますか?
assistant:
はい、ダジャレとは日本語の言葉遊びの一種で、同じ音や似た音を複数回使うことでユーモラスな効果を狙った駄洒落(ジョーク)を指します。これは日本独特の文化で、言葉の響きや意味の遊びを楽しむものです。
例えば、「時間がないなら、時計を買えばいい」や「鼻が高いのは、いい香りがするからだ」などがダジャレの例です。
ただし、私がさきほど提供したギャグは、ダジャレの形式を持っていないため、誤解を招いてしまったかもしれません。混乱を招くような表現を使ってしまい、申し訳ありませんでした。ダジャレについての説明が不足していました。