皆さんこんにちは。今日も引き続きChatGPT先生をお迎えして、「ChatGPTとPythonで学ぶ DBSCAN クラスタリング」というテーマで雑談したいと思います。それではChatGPT先生、よろしくお願いします。
assistant:
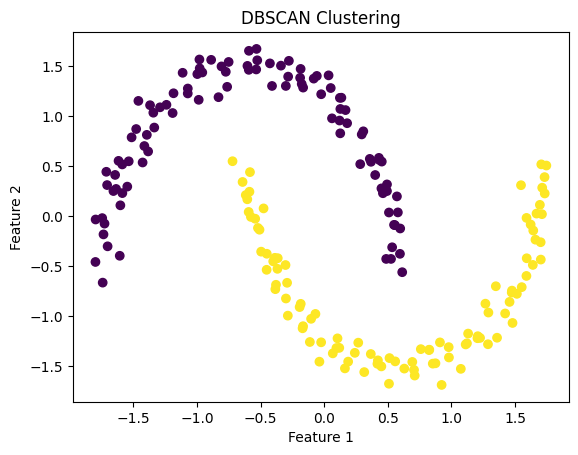
こんにちは、皆さん。今日はDBSCANというクラスタリング手法をご紹介します。DBSCANはDensity-based spatial clustering of applications with noiseの略で、高密度な空間領域を発見してそこにクラスタ(つまり群)を作る手法です。では、具体的な手順について説明しましょう。
まず、DBSCANには以下のような3つのパラメータがあります。
- ε(エプシロン): クラスタの半径というような概念です。この範囲内にある点はそのクラスタの一部と見なします。
- Minimum Points:εの範囲内に存在すべき最小限の点の数です。この数値を満たすもしくは越える点がεの範囲内にあれば、その点はコアポイント(クラスタの中心となるような点)となります。
- Noise Points:上記2つの条件を満たさない点はノイズとして扱われます。
以下にPythonで実装されたDBSCANのサンプルコードを示します。
from sklearn.datasets import make_moons
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import DBSCAN
import matplotlib.pyplot as plt
import numpy as np
# データセットを生成する
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# データを標準化する
X = StandardScaler().fit_transform(X)
# DBSCANを適用する
db = DBSCAN(eps=0.3, min_samples=5)
db.fit(X)
# ラベルを取得してプロットする
labels = db.labels_
plt.scatter(X[:, 0], X[:, 1], c=labels)
# タイトルとラベルを設定
plt.title('DBSCAN Clustering')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
# プロットを表示
plt.show()
このコードではDBSCANを用いてデータをクラスタリングし、その結果をプロットしています。
以上がDBSCANアルゴリズムの基本的な説明とPythonでの実装例です。どのように設定したεとMinimum Pointsにより、クラスタリングの結果が大きく変わることを理解してみてください。
user:
eps=0.3, min_samples=5 というハイパーパラメーターがあるようですが、それらの最適な値を決めるにはどうすれば良いですか?
assistant:
DBSCANのハイパーパラメーターε(eps)とmin_samplesを最適化するためには、実際のデータと目的によって適切に調節する必要があります。
ここで重要なのは、どのような結果を得たいかという目的と、実際のデータの性質・特性を理解することです。まず、データを可視化し、その分布を理解することから始めると良いでしょう。
次にε(eps)とmin_samplesについての基本的な考え方ですが:
-
ε(eps):
- 小さすぎる値:多くのデータ点がノイズになり、クラスタの数が増えすぎます。
- 大きすぎる値:全てのデータ点が一つのクラスタに分類される可能性があります。
-
min_samples:
- 小さすぎる値:過剰なクラスタが形成される可能性があります。
- 大きすぎる値:クラスタ数が少なくなりすぎ、あるいは全てのデータ点がノイズになる可能性があります。
これらのハイパーパラメーターをチューニングするときは、かなりの試行錯誤が必要です。様々なパラメーターの組み合わせを試すといったグリッドサーチなどの手法が一般的です。
ただし、DBSCANは不均一な密度のデータにはうまく対応できないので、それを考慮に入れる必要があります。言い換えれば、すべてのクラスタが同じ程度の密度を持っていない場合、εとmin_samplesの一つの値が全体で最適とは限らないということです。
user:
ありがとうございます。たとえばk-meansや階層的クラスタリングでは、シルエットスコアを使って最適なクラスタ数を得ることができましたが、DBSCANでは、そういうことはできないという理解で正しいですか?
assistant:
DBSCANは密度ベースのクラスタリングなので、クラスタ数を事前に決定する必要がありません。そのため、K-meansや階層クラスタリングのように、最適なクラスタ数を見つけるための評価基準(シルエットスコアなど)を使う必要はありません。
ただし、DBSCANにも評価指標は存在します。それはシルエットスコアでもありますが、DBSCANではノイズ点も考慮に入れる必要があるため、シルエットスコアの値は全体的に低くなりがちです。
DBSCANのパラメーター(εとmin_samples)を調整する際には、クラスタリングの結果が問題となる特定のタスクに対して有効であることを評価することが一般的です。
結論として、DBSCANで最適なパラメータを見つけるための最も一般的な方法は、異なるパラメータ設定で複数回実行し、その結果を直接評価することです。具体的な評価指標については、問題の性質や目的によって異なります。
user:
上記のデータに対して、DBSCANのパラメーター(εとmin_samples)をさまざまな値に変化させた時に、クラスタリングの結果がどのようになるか可視化してください。
assistant:
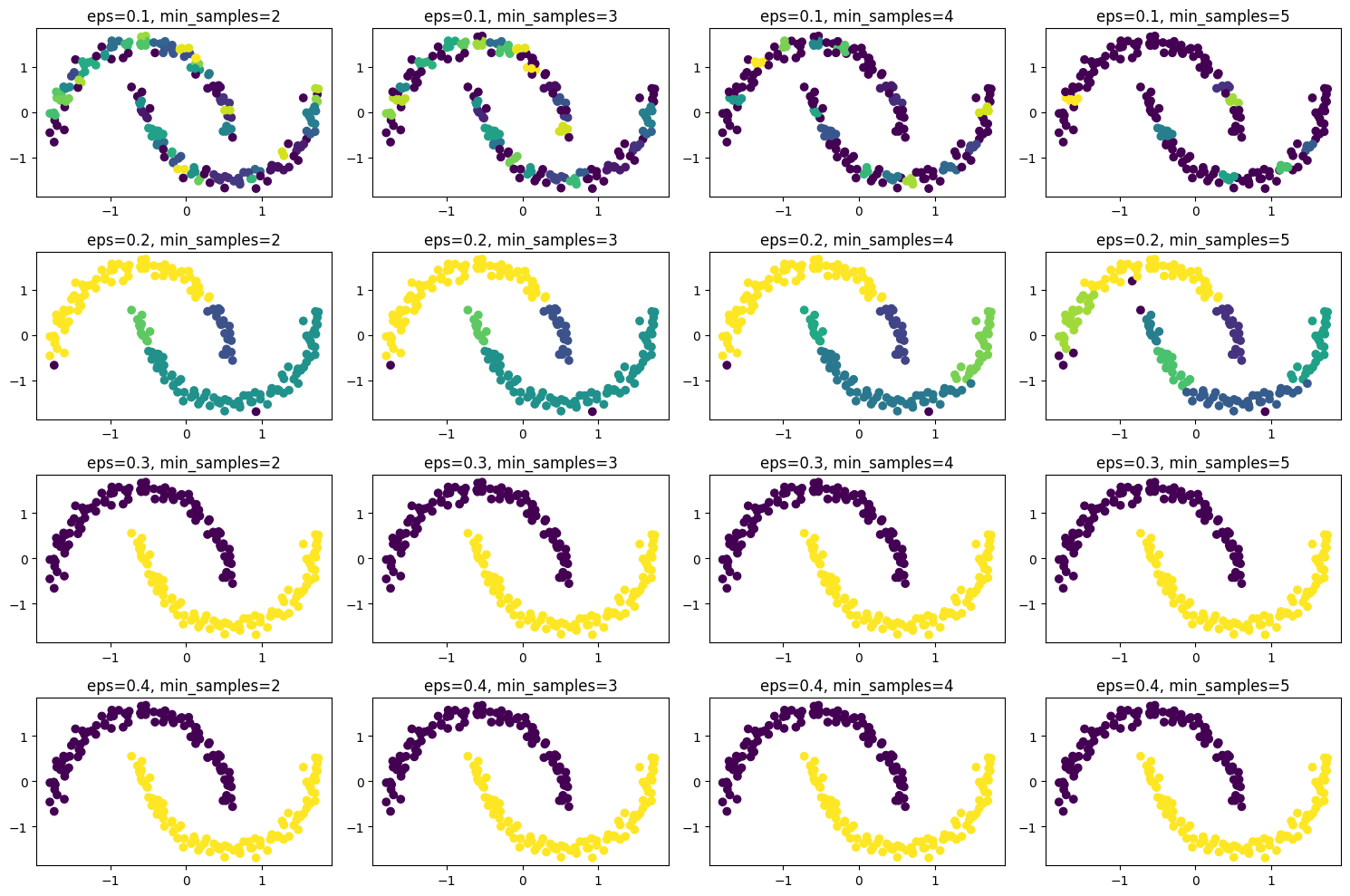
Pythonを使ってDBSCANのパラメータ(εとmin_samples)を変化させたときのクラスタリング結果の違いを視覚化しましょう。
以下のスクリプトでは、εを0.1から0.4までの間で四つの値、min_samplesを2から5までの間で四つの値にそれぞれ設定して、各組み合わせでDBSCANを適用し、その結果をプロットしています。
import matplotlib.pyplot as plt
from sklearn.datasets import make_moons
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import DBSCAN
import numpy as np
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
X = StandardScaler().fit_transform(X)
fig, axes = plt.subplots(nrows=4, ncols=4, figsize=(15, 10))
eps_values = [0.1, 0.2, 0.3, 0.4]
min_samples_values = [2, 3, 4, 5]
for i, row in enumerate(axes):
for j, cell in enumerate(row):
db = DBSCAN(eps=eps_values[i], min_samples=min_samples_values[j])
db.fit(X)
labels = db.labels_
cell.scatter(X[:, 0], X[:, 1], c=labels)
cell.set_title(f'eps={eps_values[i]}, min_samples={min_samples_values[j]}')
plt.tight_layout()
plt.show()
この結果から、εとmin_samplesの値によってクラスタリング結果がどのように変わるかを視覚的に理解することができます。例えば、εが大きすぎるとほとんどのデータが一つのクラスタにまとまってしまうこと、min_samplesが大きすぎると多くの点がノイズ(クラスタに所属しない)になることが観察できるはずです。
user:
epsが0.3以上の時は、min_samplesの値に関わらず、良好な結果が得られているようですね。epsの値が小さい時は、クラスタ数が多くなってしまって、min_samplesの値が変わると分かれ方も変わるようですね。でも、min_samplesがどのような影響を与えているかは、この図を見た限りではよく分かりません。
assistant:
min_samplesの値は、クラスタの「密度」を制御します。具体的には、min_samplesの値が高いほど「高密度」のクラスタが形成され、その結果、低密度のデータポイントはノイズとして扱われやすくなります。
この結果、min_samplesの値が大きい場合、クラスタの数が少なくなることが多くなります。また、クラスタが非常に密接している場合や、データセットが非常に大きい場合(つまり、データポイントの数が多い場合)などにおいては、min_samplesの値を大きく設定することが有効となる場合があります。
逆に、min_samplesの値が小さい場合には、より「低密度」のクラスタが許容されるため、クラスタ数が増えることが多いです。データセットが小さい場合や、クラスタが適度に分散している場合には、min_samplesの値を小さく設定することが有効です。
前述の通り、これらのパラメータの最適な値はデータの性質に大きく依存しますが、実践的には様々な値で試行錯誤することになるでしょう。その際に、min_samplesが密度の観点からクラスタ形成に影響を与えると認識しておくと良いでしょう。
user:
クラスタリングがうまくいったかどうか、可視化すれば分かる気はしますが、可視化しないで客観的に判断する方法はありませんか?
assistant:
クラスタリングの評価には、内部評価指標と外部評価指標という2つのカテゴリが存在します。
-
内部評価指標:これはクラスタリング自体の品質を評価します。真のクラスター情報が不明あるいは存在しない場合に適しています。シルエットスコアは内部評価指標の一例です。これは各点のクラスター内の一貫性(同一クラスター内の他の点との距離)とクラスター間の差異性(最も近い他のクラスターとの距離)を使用してクラスタリングの品質を評価します。
-
外部評価指標:これは何らかの「真の」クラスターラベルが存在していて、それを元にクラスタリングの結果を評価します。一般的な外部評価指標は、調整ランド指数、ジャッカード係数、相互情報量などがあります。
可視化することが難しい(たとえば、次元数が多すぎる)場合や、真のクラスターラベルが存在しない(つまり、教師なし学習の場合)場合は、内部評価指標を用いてクラスタリングの良し悪しを評価することも可能です。ただし、これらの評価指標も必ずしも「最適な」クラスタリングを求めるための完全なガイドラインを提供するわけではなく、各指標には一長一短があります。したがって、最終的な判断はそれぞれのユースケース、問題設定、そして知識によって異なる場合があります。
user:
DBSCANを使って2次元データをクラスタリングし、内部評価指標を用いて、最適なクラスタリングを行うパラメータを発見するPythonコードを書いてください。
assistant:
DBSCANのパラメータ選択に影響を与える二つの主要なパラメータは、eps(近傍距離)とmin_samples(コア点とするために必要な最小限の近傍点数)です。さまざまなパラメータ設定を試し、各設定についてシルエットスコアを計算し、最も良いスコアを出すパラメータを選びます。
以下に、そのためのコード例を示します。
from sklearn.datasets import make_moons
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import DBSCAN
from sklearn.metrics import silhouette_score
import numpy as np
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
X = StandardScaler().fit_transform(X)
# パラメータ範囲
eps_values = np.arange(0.1, 0.5, 0.1)
min_samples_values = np.arange(2, 6, 1)
best_eps = None
best_min_samples = None
best_score = -1
# 各パラメータ値の組み合わせについてDBSCANを実行し、シルエットスコアを計算
for eps in eps_values:
for min_samples in min_samples_values:
db = DBSCAN(eps=eps, min_samples=min_samples)
labels = db.fit_predict(X)
if len(set(labels)) > 1:
score = silhouette_score(X, labels)
if score > best_score:
best_eps = eps
best_min_samples = min_samples
best_score = score
print(f'Best parameters: eps = {best_eps}, min_samples = {best_min_samples}, with silhouette score = {best_score}')
Best parameters: eps = 0.2, min_samples = 5, with silhouette score = 0.4468495377426796
このコードでは、DBSCANの各パラメータの値の組み合わせについてシルエットスコアを計算し、最も高いスコアを出すパラメータの組み合わせを探します。
なお、シルエットスコアはシルエット係数の平均で、シルエット係数は-1から1までの値で、1に近いほど良いクラスタリング結果を表します。しかし、クラスタの数が2より少ない場合(すなわち、すべてのサンプルが一つのクラスタに属するか、まったくクラスタリングされていない場合)、シルエットスコアを計算することはできないので注意が必要です。そのため、クラスタの数が2以上の場合にのみスコアを計算しています。
user:
シルエットスコアについて理解を深めるために、シルエットスコアを計算する関数を、sklearn.metrics の silhouette_score を使わずにスクラッチで実装してください。
assistant:
シルエットスコアは、クラスタ間距離(クラスタ間の分離度)とクラスタ内距離(クラスタ内の凝集度)のバランスを測定します。クラスタ内距離が小さく、クラスタ間距離が大きいほど、シルエットスコアは大きくなります。全てのサンプルのシルエット係数の平均がシルエットスコアとなります。
1つのサンプルについてシルエット係数 s を求める手順は以下の通りです:
- a:同じクラスタ内の全ての他のサンプルとの平均距離を計算します。これは、そのサンプルが所属するクラスタの凝集度を測定します。
- b:次に最も近いクラスタ内の全てのサンプルとの平均距離を計算します。これは、そのサンプルが隣のクラスタとどの程度離れているかを測定します。
- s = (b - a) / max(a, b)
以下に、シルエットスコアを求めるPythonの関数を示します:
import numpy as np
from scipy.spatial import distance
def silhouette_score_scratch(X, labels):
all_silhouette_scores = []
# 各データについてシルエット係数を計算
for i in range(X.shape[0]):
distances = distance.cdist([X[i]], X, metric='euclidean') # 与えられた点から他の全ての点への距離
cluster_distances = np.delete(distances, i) # 自分自身への距離0を除く
# クラスタ内距離 a
in_cluster_data = cluster_distances[labels == labels[i]]
a = np.mean(in_cluster_data)
# 最近傍クラスタ内距離 b
nearest_cluster_distances = []
for j in set(labels):
if j != labels[i]: # 自分自身のクラスタ除外
other_cluster_data = cluster_distances[labels == j]
nearest_cluster_distances.append(np.mean(other_cluster_data))
b = min(nearest_cluster_distances)
# シルエット係数
s = (b - a) / max(a, b)
all_silhouette_scores.append(s)
# 全てのシルエット係数の平均を取ってシルエットスコアとする
return np.mean(all_silhouette_scores)
この関数は、データ行列Xとその対応するラベルを入力とし、全データのシルエット係数の平均を返します。注意点として、この関数は計算量が大きいため、大量のデータに対してはかなり時間がかかる可能性があります。また、このシルエットスコアは -1 から 1 までの範囲の値を取り、1に近いほど良いクラスタリングを示します。
user:
シルエットスコアはユークリッド距離に基づいているのですよね?ということは、今回のようなmake_moonsのデータセットをクラスタリングした時の指標には適さないのではないでしょうか?
assistant:
その通りです。シルエットスコアはユークリッド距離に基づいているため、"make_moons"のようなクラスタ構造が複雑なデータセットの場合、必ずしも最適なクラスタリングを評価できるわけではありません。
シルエットスコアが良い結果を示すのは、クラスタが凸形状で、クラスタ間の距離がクラスタ内距離より十分に大きいときです。また、すべてのクラスタが同じ大きさと密度を持っている場合も、シルエットスコアは有用です。
しかし、"make_moons"のようなデータセットでは、特定のクラスタが他のクラスタに包含されている、あるいはクラスタが複雑な形状をしているため、シルエットスコアは必ずしも最適な結果を示さない可能性があります。
そのため、こうしたデータセットに対してクラスタリングを行う際には、他の評価指標を検討するか、クラスタリングアルゴリズムがデータの形状や構造を適切に捉えられているかを視覚的に確認することが重要です。
user:
make_moonsデータセットのクラスタリングに使えるような「他の評価指標」として、何がありますか?
assistant:
"make_moons"のようなデータセットに対するクラスタリングの評価には、「Davies-Bouldin Index」、「Calinski-Harabasz Index」のような内部クラスタリング指標が有効です。これらの指標は、クラスタの分散や距離を考慮することで、クラスタリングの性能を評価します。
-
Davies-Bouldin Index:
- この指標は、クラスタ内の分散(クラスタのサンプルがどれだけ広がっているか)とクラスタ間の分散(クラスタがどれだけ離れているか)を考慮して評価します。
- 低い値が良い性能を示します。
- 計算のために、各クラスタ内の全ての点と、そのクラスタの重心との平均平方距離(つまり、分散)を使用します。
-
Calinski-Harabasz Index:
- この指標はクラスタ間の分散(クラスタがどれだけ離れているか)とクラスタ内の分散(クラスタのサンプルがどれだけ近いか)を考慮して評価します。
- 高い値が良い性能を示します。
- 各クラスタ間の距離(具体的には、クラスタ間の平方距離の合計)と、クラスタ内の距離(具体的には、各クラスタ内の平方距離の合計)の比率で計算します。
これらの指標の利点は、各サンプルのラベル情報だけで計算でき、非凸形状のクラスタや異なる大きさ・密度のクラスタに対しても使用できることです。それぞれsklearn.metricsモジュール内のdavies_bouldin_score、calinski_harabasz_score関数で計算することができます。
ただし、これらの指標も一長一短があり、最適なクラスタリングを必ずしも導くわけではありません。評価指標の選択は、問題の性質や目的、データの特性によります。したがって、様々な指標を試し、それぞれの結果を比較することが重要です。
user:
それでは、DBSCANを使ってmake_moonsデータをクラスタリングし、シルエットスコアを用いて、最適なクラスタリングを行うパラメータを発見し、そのベストパラメータでクラスタリングした結果を可視化するPythonコードを書いてください。
assistant:
DBSCANのパラメータを調整してmake_moonsデータをクラスタリングし、シルエットスコアに基づいて結果を評価し、最良のクラスタリング結果を見つけ、可視化するためのPythonコードを以下に示します。
import matplotlib.pyplot as plt
from sklearn.datasets import make_moons
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import DBSCAN
from sklearn.metrics import silhouette_score
import numpy as np
# データセットの生成
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
X = StandardScaler().fit_transform(X)
# パラメータ範囲
eps_values = np.arange(0.1, 0.5, 0.1)
min_samples_values = np.arange(2, 6, 1)
best_eps = None
best_min_samples = None
best_score = -1
# 各パラメータ値の組み合わせについてDBSCANを実行し、シルエットスコアを計算
for eps in eps_values:
for min_samples in min_samples_values:
db = DBSCAN(eps=eps, min_samples=min_samples)
labels = db.fit_predict(X)
# ノイズデータの除外
no_noise_labels = labels[labels != -1]
no_noise_X = X[labels != -1]
# クラスタ数が2以上ある場合のみスコアを計算
if len(set(no_noise_labels)) > 1:
score = silhouette_score(no_noise_X, no_noise_labels)
if score > best_score:
best_eps = eps
best_min_samples = min_samples
best_score = score
print(f'Best parameters: eps = {best_eps}, min_samples = {best_min_samples}, with silhouette score = {best_score}')
# ベストパラメータでクラスタリングし、結果を可視化
db = DBSCAN(eps=best_eps, min_samples=best_min_samples)
labels = db.fit_predict(X)
plt.scatter(X[labels == 0, 0], X[labels == 0, 1], s=50, color='red')
plt.scatter(X[labels == 1, 0], X[labels == 1, 1], s=50, color='blue')
plt.show()
Best parameters: eps = 0.1, min_samples = 5, with silhouette score = 0.8436292171021341
このコードは、各パラメータの組み合わせでDBSCANを適用し、シルエットスコアを計算します。ベストスコアを返すパラメータを選択し、それを用いて最終的なクラスタリングを行い、結果を出力します。結果は、異なる色で表示される二つの明瞭なクラスタを視覚的に示します。
user:
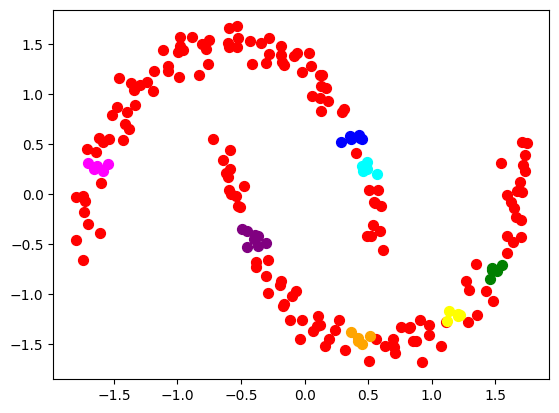
2つを超える数のクラスターに分割されたため、可視化がうまく行かないようです。全てのクラスターを可視化するように、上記のコードを改変してください。
assistant:
そうですね、2つ超える数のクラスタを含む結果を可視化するように上記のコードを修正してみましょう。ここではmatplotlibの色のリストを使うことで、各クラスタにユニークな色を割り当て、可視化します。
import matplotlib.pyplot as plt
from sklearn.datasets import make_moons
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import DBSCAN
from sklearn.metrics import silhouette_score
import numpy as np
# データセットの生成
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
X = StandardScaler().fit_transform(X)
# パラメータ範囲
eps_values = np.arange(0.1, 0.5, 0.1)
min_samples_values = np.arange(2, 6, 1)
best_eps = None
best_min_samples = None
best_score = -1
# 各パラメータ値の組み合わせについてDBSCANを実行し、シルエットスコアを計算
for eps in eps_values:
for min_samples in min_samples_values:
db = DBSCAN(eps=eps, min_samples=min_samples)
labels = db.fit_predict(X)
# ノイズデータの除外
no_noise_labels = labels[labels != -1]
no_noise_X = X[labels != -1]
# クラスタ数が2以上ある場合のみスコアを計算
if len(set(no_noise_labels)) > 1:
score = silhouette_score(no_noise_X, no_noise_labels)
if score > best_score:
best_eps = eps
best_min_samples = min_samples
best_score = score
print(f'Best parameters: eps = {best_eps}, min_samples = {best_min_samples}, with silhouette score = {best_score}')
# ベストパラメータでクラスタリングし、結果を可視化
db = DBSCAN(eps=best_eps, min_samples=best_min_samples)
labels = db.fit_predict(X)
# 使用する色のリスト
colors = ['red', 'blue', 'green', 'purple', 'orange', 'yellow', 'cyan', 'magenta', 'black']
# ユニークなクラスタラベル
unique_labels = np.unique(labels)
for i in range(len(unique_labels)):
plt.scatter(X[labels == unique_labels[i], 0], X[labels == unique_labels[i], 1], s=50, color=colors[i % len(colors)])
plt.show()
Best parameters: eps = 0.1, min_samples = 5, with silhouette score = 0.8436292171021341
これにより、2以上のクラスタ数の場合でも、各クラスタが異なる色によって可視化されます。ただし、色の数がクラスタ数を上回るように、足りなければ色のリストを追加してください。
user:
DBSCANを使ってmake_moonsデータをクラスタリングし、Davies-Bouldin Indexを用いて、最適なクラスタリングを行うパラメータを発見し、そのベストパラメータでクラスタリングした結果を可視化するPythonコードを書いてください。2以上のクラスタ数の場合でも、各クラスタを異なる色によって可視化してください。
assistant:
DBSCANのパラメータを調整してmake_moonsデータをクラスタリングし、Davies-Bouldin Indexに基づいて結果を評価し、最良のクラスタリング結果を見つけ、可視化するためのPythonコードを以下に示します。
import matplotlib.pyplot as plt
from sklearn.datasets import make_moons
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import DBSCAN
from sklearn.metrics import davies_bouldin_score
import numpy as np
# データセットの生成
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
X = StandardScaler().fit_transform(X)
# パラメータ範囲
eps_values = np.arange(0.1, 0.5, 0.1)
min_samples_values = np.arange(2, 6, 1)
best_eps = None
best_min_samples = None
best_score = float('inf') # 最小化問題なので初期値は無限大
# 各パラメータ値の組み合わせについてDBSCANを実行し、Davies-Bouldin Indexを計算
for eps in eps_values:
for min_samples in min_samples_values:
db = DBSCAN(eps=eps, min_samples=min_samples)
labels = db.fit_predict(X)
# ノイズデータ及びクラスタ数1の場合を除く
no_noise_labels = labels[labels != -1]
no_noise_X = X[labels != -1]
if len(set(no_noise_labels)) > 1:
score = davies_bouldin_score(no_noise_X, no_noise_labels)
if score < best_score:
best_eps = eps
best_min_samples = min_samples
best_score = score
print(f'Best parameters: eps = {best_eps}, min_samples = {best_min_samples}, with Davies-Bouldin Index = {best_score}')
# ベストパラメータでクラスタリングし、結果を可視化
db = DBSCAN(eps=best_eps, min_samples=best_min_samples)
labels = db.fit_predict(X)
# 使用する色のリスト
colors = ['red', 'blue', 'green', 'purple', 'orange', 'yellow', 'cyan', 'magenta', 'black']
# ユニークなクラスタラベル
unique_labels = np.unique(labels)
for i in range(len(unique_labels)):
plt.scatter(X[labels == unique_labels[i], 0], X[labels == unique_labels[i], 1], s=50, color=colors[i % len(colors)])
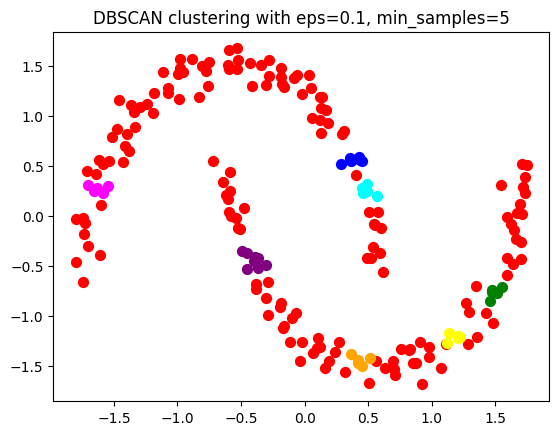
plt.title(f"DBSCAN clustering with eps={best_eps}, min_samples={best_min_samples}")
plt.show()
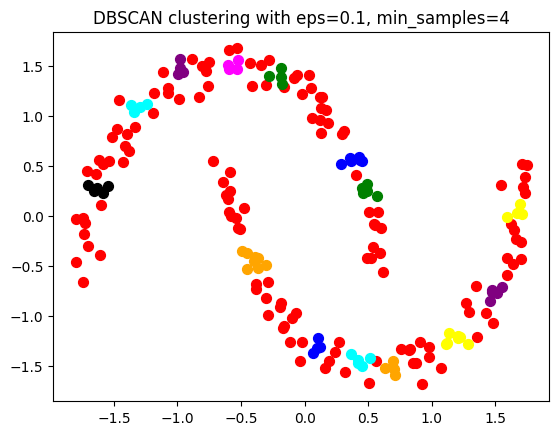
Best parameters: eps = 0.1, min_samples = 5, with Davies-Bouldin Index = 0.19588367788331634
このコードは、各パラメータの組み合わせでDBSCANを適用し、Davies-Bouldin Indexを計算します。ベストスコア(最小値)を返すパラメータを選択し、それを用いて最終的なクラスタリングを行い、結果を出力します。結果は、異なる色で表示されるクラスタを視覚的に示します。
user:
DBSCANを使ってmake_moonsデータをクラスタリングし、Calinski-Harabasz Indexを用いて、最適なクラスタリングを行うパラメータを発見し、そのベストパラメータでクラスタリングした結果を可視化するPythonコードを書いてください。2以上のクラスタ数の場合でも、各クラスタを異なる色によって可視化してください。
assistant:
DBSCANのパラメータを調整してmake_moonsデータをクラスタリングし、Calinski-Harabasz Indexに基づいて結果を評価し、最良のクラスタリング結果を見つけ、可視化するためのPythonコードを以下に示します。
import matplotlib.pyplot as plt
from sklearn.datasets import make_moons
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import DBSCAN
from sklearn.metrics import calinski_harabasz_score
import numpy as np
# データセットの生成
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
X = StandardScaler().fit_transform(X)
# パラメータ範囲
eps_values = np.arange(0.1, 0.5, 0.1)
min_samples_values = np.arange(2, 6, 1)
best_eps = None
best_min_samples = None
best_score = -1
# 各パラメータ値の組み合わせについてDBSCANを実行し、Calinski-Harabasz Indexを計算
for eps in eps_values:
for min_samples in min_samples_values:
db = DBSCAN(eps=eps, min_samples=min_samples)
labels = db.fit_predict(X)
# ノイズデータ及びクラスタ数1の場合を除く
no_noise_labels = labels[labels != -1]
no_noise_X = X[labels != -1]
if len(set(no_noise_labels)) > 1:
score = calinski_harabasz_score(no_noise_X, no_noise_labels)
if score > best_score:
best_eps = eps
best_min_samples = min_samples
best_score = score
print(f'Best parameters: eps = {best_eps}, min_samples = {best_min_samples}, with Calinski-Harabasz Index = {best_score}')
# ベストパラメータでクラスタリングし、結果を可視化
db = DBSCAN(eps=best_eps, min_samples=best_min_samples)
labels = db.fit_predict(X)
# 使用する色のリスト
colors = ['red', 'blue', 'green', 'purple', 'orange', 'yellow', 'cyan', 'magenta', 'black']
# ユニークなクラスタラベル
unique_labels = np.unique(labels)
for i in range(len(unique_labels)):
plt.scatter(X[labels == unique_labels[i], 0], X[labels == unique_labels[i], 1], s=50, color=colors[i % len(colors)])
plt.title(f"DBSCAN clustering with eps={best_eps}, min_samples={best_min_samples}")
plt.show()
Best parameters: eps = 0.1, min_samples = 4, with Calinski-Harabasz Index = 1847.7315875547401
このコードは、各パラメータの組み合わせでDBSCANを適用し、Calinski-Harabasz Indexを計算します。ベストスコア(最大値)を返すパラメータを選択し、それを用いて最終的なクラスタリングを行い、結果を出力します。結果は、異なる色で表示されるクラスタを視覚的に示します。
user:
うーん、シルエットスコアでうまく行かないのは理解できますが、Davies-Bouldin IndexでもCalinski-Harabasz Indexでも、うまく行かないですね。
assistant:
DBSCANは密度ベースのアルゴリズムで、パラメータで密度が定義されます(epsとmin_samples)。したがって、それらのパラメータ設定が重要となります。何か他のインデックスを試して見るか、アルゴリズムを変えてみるのも良いかもしれません。
また、多くのクラスタリング評価指標は、クラスタが凸形状(つまり、それらが球状または楕円状であることが期待される)かつクラスタ間に空間的な分離があるときに最もうまく機能します。しかし、DBSCANはこのような割り当てを必ずしも行わないため、これらの指標がDBSCANでのパフォーマンスを適切に反映しない場合があります。
さらに、これらの評価指標のいずれもノイズ(DBSCANによってラベル-1が割り当てられるデータポイント)を適切に処理する方法が明確に定義されていません。したがって、これらの指標がDBSCANのノイズを考慮する方法によって大幅に影響を受ける可能性があります。
データ分布と目的により適したクラスタリングアルゴリズムと評価指標を検討すると良いです。実際のデータサイエンスのプロジェクトでは、一部のデータを使用して数種類のアルゴリズムとパラメータを試し、未知のデータに対して一番良い結果をもたらすモデルを探します。また、領域知識と直感も重要な役割を果たします。
user:
Davies-Bouldin Indexについて理解を深めるために、Davies-Bouldin Indexを計算する関数を、sklearn.metrics の davies_bouldin_score を使わずにスクラッチで実装してください。コード中にて日本語でできるだけ詳しく説明してください。
assistant:
Davies-Bouldin Index (DBI) は、クラスタリングの評価指標の一つで、クラスタ内のデータのばらつき(内部散布:データがクラスタの中心からどれだけ離れているか)とクラスタ間のデータのばらつき(クラスタの中心がどれだけ離れているか)のバランスを評価します。具体的な計算式は以下の通りです:
- 各クラスタ内の全ての要素とクラスタ中心との距離の平均を計算(内部散布)
- 2つのクラスタ間のクラスタ中心間距離を計算
- 1と2の比(分散比)を計算
- 最大の分散比を選択(全てのクラスタ対に対して)
from scipy.spatial.distance import pdist, squareform
def davies_bouldin_index(X, labels):
# 各クラスタの中心を計算
n_cluster = len(np.bincount(labels))
cluster_k = [X[labels == k] for k in range(n_cluster)]
centroids = [np.mean(k, axis = 0) for k in cluster_k]
# クラスタ内の全ての要素とクラスタ中心との距離の平均を計算
variances = [np.mean([np.linalg.norm(p-c) for p in k]) for k, c in zip(cluster_k, centroids)]
db = []
for i in range(n_cluster):
for j in range(n_cluster):
if j != i:
db.append((variances[i] + variances[j]) / np.linalg.norm(centroids[i] - centroids[j]))
# return maximum value of DB list
return(np.max(db))
データの距離計算には、Numpyの線形代数ライブラリ np.linalg の norm関数を使い、ベクトル間のユークリッド距離(L2ノルム)を計算しています。一方、クラスタ中心は各クラスタの座標値の平均値で求めています。
Davies-Bouldin Indexの値が小さいほど良いクラスタリング結果と評価されます。なぜなら、各クラスタ内のデータ点がクラスタ中心に近く、かつ異なるクラスタの中心が離れているほど、Indexの値は小さくなるからです。
user:
計算内容がシルエットスコアとほとんど変わらない気がしますね
assistant:
実際に、シルエットスコアとDavies-Bouldin Index(DBI)は、クラスタリングの品質を評価するための指標として類似点があります。どちらもクラスタ内の一貫性(データが同じクラスタ内の他のデータにどれだけ近いか)とクラスタ間の差異(データが他のクラスタのデータからどれだけ遠いか)を評価します。
しかし、それぞれの計算方式には重要な違いがあります:
-
シルエットスコアは、各データ点のシルエット係数(そのデータ点が所属するクラスタ内の他の点からの平均距離と、最も近い他のクラスタのデータ点からの平均距離の差と比率)の平均値を計算します。すなわち、データ点ごとの評価を行います。
-
一方、DBIは、各クラスタの「内部散布」と「他のクラスタとの分離」を計算し、それらの比の最大値を指標とします。すなわち、クラスタごとの評価を行います。
つまり、同じくらい良いクラスタリング結果でも、各データ点の一貫性(シルエットスコア)とクラスタ全体の一貫性(DBI)を重視するかによって、両者の値は異なる場合があります。そのため、どちらの指標を使用するかは問題設定やデータの性質によると言えます。
user:
Calinski-Harabasz Indexについて理解を深めるために、Calinski-Harabasz Indexを計算する関数を、sklearn.metrics の calinski_harabasz_score を使わずにスクラッチで実装してください。コード中にて日本語でできるだけ詳しく説明してください。
assistant:
Calinski-Harabaszスコア(CHスコア)は、クラスタリングの評価指標の一つで、クラスタ間・クラスタ内分散比を評価します。具体的な計算式は以下の通りです:
- クラスタ全体の負荷(各クラスタ重心から全体の重心までの距離の二乗和)を計算(クラスタ間散布)
- 各クラスタ内の全ての要素とクラスタ重心との距離の二乗和を計算(クラスタ内散布)
- クラスタ間散布をクラスタ内散布で割り、更にデータ件数nとクラスタ数kで正規化
import numpy as np
def calinski_harabasz_index(X, labels):
n_samples, _ = X.shape
n_labels = len(np.bincount(labels))
# Check that we got at least two clusters, otherwise return negative infinity
if n_labels < 2:
return -np.inf
# Compute the sum of squares between and within each cluster
cluster_k = [X[labels == k] for k in range(n_labels)]
cluster_k_means = [np.mean(k, axis = 0) for k in cluster_k]
centroid = np.mean(X, axis=0)
ssw = 0 # Sum of squares within clusters
ssb = 0 # Sum of squares between clusters
for k in range(n_labels):
ssw += np.sum([np.square(x - cluster_k_means[k]).sum() for x in cluster_k[k]])
ssb += len(cluster_k[k]) * np.square(np.linalg.norm(cluster_k_means[k]-centroid))
# Compute the CH score
score = (ssb/ssw) * (n_samples-n_labels) / (n_labels-1.)
return score
まず、クラスタ内の平方和とクラスタ間の平方和を計算します。それらの比を求め、データ件数nとクラスタ数kで正規化します。ここで、クラスタ内の平方和は、クラスタ内の全データ点がクラスタ中心からどれだけばらついているかを示し、クラスタ間の平方和は、各クラスタ中心が全クラスタの重心からどれだけ離れているか(つまり、クラスタが互いにどれだけ離れているか)を示します。
Calinski-Harabasz Indexの値が大きいほど良いクラスタリング結果と評価されます。すなわち、クラスタ内のデータ点がクラスタ中心に近く、かつ異なるクラスタの中心が離れているほど、Indexの値は大きくなります。
user:
なるほど、そうすると、シルエットスコアはもちろん、Davies-Bouldin IndexでもCalinski-Harabasz Indexでも、make_moonsデータセットのクラスタリングを正しく評価できないのは当たり前ですね。
assistant:
その通りです。make_moonsデータセットは非凸形で、2つの半月状のクラスタが強く重なっていて、それらは一見して一つのクラスタに見えるかもしれません。シルエットスコア、Davies-Bouldin Index、そしてCalinski-Harabasz Indexなどの指標は、一般的には凸形のクラスタを想定しています。つまり、それらの指標は、クラスタが球形または楕円形で、クラスタ間には明確な空間的な分離があるという前提を基に計算されます。
しかし、非凸形や非線形構造のクラスタ(make_moonsデータセットのような)を識別するためにデザインされている密度ベースのクラスタリングアルゴリズム(例如、DBSCAN)でクラスタリングを行うと、これらの指標ではその優れた性能をうまく評価できません。
そのため、非凸や複雑な形状のクラスタを扱う場合には、適切な評価指標やクラスタリングアルゴリズムを選択することが重要となります。具体的な選択は、クラスタリングを行いたい具体的なデータセットや、それに伴う問題設定に大きく依存します。また、一部のデータを使用して様々なアルゴリズムを試し、未知のデータに対する一番良い結果を持つモデルを見つけることも有効です。
user:
DBSCANについて理解を深めるために、DBSCANをする関数を、sklearn.cluster の DBSCANを使わずにスクラッチで実装してください。コード中にて日本語でできるだけ詳しく説明してください。
assistant:
DBSCAN(Density-Based Spatial Clustering of Applications with Noise)は、密度ベースのクラスタリング法の一つで、特定の範囲内にある最低数のデータポイントに基づいてクラスタを形成します。以下にそのスクラッチ実装を記述します。
実装は次のようなフローになります。
- まず全てのデータ点を未訪問(UNVISITED)とします。
- あるデータ点をランダムに選び、その点がまだ訪問されていなければ(UNVISITEDならば)、新たなクラスタを作成し、その点をクラスタに追加します。そしてその点の状態を訪問済み(VISITED)に変更します。
- 選んだ点から一定の距離内(eps)に存在する点を探し、その数がある閾値(minPts)を超えていればその点を新たなクラスタのメンバーとします。そしてそれらの点を再帰的に探索し、距離eps内にminPts以上存在する点を新たにクラスタのメンバーとします。これを、新たにクラスタのメンバーとなる点が見つからなくなるまで続けます。
- それ以上新たなクラスタのメンバーが見つからなければ、2.に戻り新たなクラスタを探し始めます。これを全てのデータ点が訪問済みになるまで繰り返します。
なお、距離の計算にはユークリッド距離を用いています。
以下にPythonでのスクラッチ実装を示します。
import numpy as np
from scipy.spatial import distance
def DBSCAN(X, eps, minPts):
labels = [0]*len(X)
C = 0
# 距離行列を作成
D = distance.cdist(X, X, 'euclidean')
for p in range(0, len(X)):
if not (labels[p] == 0):
continue
# 点pから距離eps以内の点を探す
neighbors = []
for i in range(len(X)):
if D[p][i] <= eps:
neighbors.append(i)
# minPts未満ならノイズ(-1)にする
if len(neighbors) < minPts:
labels[p] = -1
else:
# 新しいクラスタを形成
C += 1
labels[p] = C
# 隣接点の集合に基づくクラスタの成長
i = 0
while i < len(neighbors):
Pn = neighbors[i]
if labels[Pn] == -1:
labels[Pn] = C
elif labels[Pn] == 0:
labels[Pn] = C
Pnneighbors = []
for _i in range(len(X)):
if D[Pn][_i] <= eps:
Pnneighbors.append(_i)
if len(Pnneighbors) >= minPts:
neighbors = neighbors + Pnneighbors
i += 1
return labels
このスクラッチ実装はあくまで理解の参考であり、実際の使用時には最適化されたライブラリの利用を推奨します。また、スクラッチ実装の理解を深めるために、ユークリッド距離以外の距離指標を用いてみたり、パラメータを変更して結果がどのように変わるか試してみると良いでしょう。
user:
それでは、上記のスクラッチDBSCANを用いてmake_moonsデータセットをクラスタリングし、その結果を可視化するPythonコードを書いてください。
assistant:
まず、make_moonsデータセットを作成し、上記で定義したDBSCAN関数を使ってクラスタリングを行い、最後に結果をプロットします。
以下にそのPythonコードを示します。
from sklearn.datasets import make_moons
import matplotlib.pyplot as plt
# データの生成
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# DBSCANでクラスタリング
labels = DBSCAN(X, eps=0.3, minPts=5)
# データをプロット
plt.scatter(X[:,0], X[:,1], c=labels)
plt.show()
このコードにより、make_moonsデータセットをDBSCANを用いてクラスタリングし、その結果を色分けして2次元プロットした結果が表示されます。DBSCANはクラスタ数を事前に決める必要がなく、また凸でない形状のクラスタを検出することができますので、make_moonsデータセットのような複雑な形状のデータをうまくクラスタリングできるはずです。epsとminPtsの値は適当に設定したものですので、必要に応じて調節してください。
user:
できました!
assistant:
素晴らしいですね!うまくクラスタリングが行えたようで、とても良かったです。
クラスタリングは幅広い応用がありますが、データの特性や目的によって適切なアルゴリズムやパラメータを選ぶことが重要です。実際にデータに手を触れてみることで、その理解が深まると思います。
これからもデータ解析に関する何か質問がありましたら、いつでもお気軽にお問い合わせください。どんな質問でもお答えできるよう、全力でサポートいたします。
user:
上記の簡易型DBSCANでは実装できてなくて、scikit-learnのDBSCANでは実装できていることはありますか?詳しく教えてください。
assistant:
スクラッチによる簡易型のDBSCANとscikit-learnのDBSCANの間には、いくつかの違いがあります。その主な違いは以下の通りです:
-
最適化: scikit-learnのDBSCANは、効率化と最適化が行われており、高次元データや大規模データに対処するためのアルゴリズムが採用されています。対照的に、スクラッチの実装は基本的なアルゴリズムのみで構成されており、これらの最適化手法は採用されていません。
-
メモリ効率: scikit-learnのDBSCANは、距離計算を効率的に行うために特定のデータ構造(例えば、KD-treeやBall-tree)を使用しています。これにより、メモリ使用量を抑えつつ、ネイバー検索の計算時間を短縮しています。一方、スクラッチ実装ではこれらのデータ構造を使用していません。
-
パラメータ設定: scikit-learnのDBSCANでは
eps(探索範囲の半径)やmin_samples(クラスタを形成するための最小点数)など、いくつかの重要なパラメータを調整することが可能です。これにより、データセットの特性に合わせて細かくモデルを調整することができます。対してスクラッチ実装では多くのパラメータが固定されています。 -
距離指標: scikit-learnのDBSCANでは、ユークリッド距離だけでなく、マンハッタン距離やマハラノビス距離など他の距離指標も使用することができます。一方、スクラッチ実装ではユークリッド距離のみを使用しています。
-
計算時間: 最適化や効率化が行われているため、scikit-learnのDBSCANはスクラッチ実装と比べて一般に計算が早く終わることが多いです。
以上のように、scikit-learnのDBSCANとスクラッチ実装のDBSCANとではいくつかの違いがあります。しかし、概念を理解する初期段階では、スクラッチ実装も有益です。そして、より高度な機能や効率化が必要になった場合には、専用のライブラリを使用することをお勧めします。
user:
ありがとうございます。DBSCANは密度ベースのクラスタリング法の一つとのことですが、密度ベースのクラスタリング法は他に何がありますか?詳しく教えてください。
assistant:
はい、DBSCANは密度ベースのクラスタリング手法の一つで、密度ベースとはデータポイントが密集している領域をクラスタとみなす手法のことを言います。
DBSCAN以外にもいくつかの密度ベースのクラスタリング手法が存在します。以下にその例を挙げます:
-
Mean-Shift: Mean-Shiftは滑らかな密度分布を勾配上昇法で登っていき、密度のピークを見つけることでクラスタを形成します。初期値を変えて何度も適用し、最終的にどれだけのピークを見つけられたかでクラスタ数を決めます。初期値をどう選ぶかという問題や計算コストの高さが課題となりますが、クラスタ数を事前に設定する必要が無い点や形状の制約が無いことが強みです。
-
OPTICS (Ordering Points To Identify the Clustering Structure): OPTICSはDBSCANの改良版とも言える手法で、DBSCANと同様に密度ベースでクラスタを作る一方で、パラメータepsによる制約を緩和しています。そのため、DBSCANに比べて密度が大きく異なるクラスタが混在する場合などに有効です。
-
HDBSCAN (Hierarchical Density-Based Spatial Clustering of Applications with Noise): HDBSCANもDBSCANの発展形で、階層的クラスタリングの手法を取り入れることで、より一層複雑な構造のデータに対応しています。群集に属さないノイズポイントも無理にクラスタに含まれることはないため、クラスタの品質が向上しやすいとされています。
各手法にはそれぞれ長所と短所がありますので、使用するデータや課題によって適した手法を選択することが重要です。
なお、これらの手法もPythonのライブラリであるScikit-learnを用いて実装することが可能です。
user:
そういえば、DBSCANって、階層的クラスタリングにおける最短距離法や、グラフ理論における連結成分の切り出しとよく似てますね。
assistant:
その通りです。DBSCAN、階層的クラスタリングの最短距離法(single linkage method)、そしてグラフ理論における連結成分の切り出しは、どれも「近傍を考える」という共通する思考方法を持っています。
-
DBSCAN :ある基準(距離と最小の点の数)に基づいて近傍領域を特定し、それらを元にクラスタを生成します。DBSCANは凸形状でないクラスタを識別することができますが、密度(epsとminPtsパラメータ)でクラスタを定義しているため、密度の異なるクラスタへの対応が難しい場合があります。
-
階層的クラスタリング - 最短距離法:二つのクラスタ間で最も近い点の距離をクラスタ間の距離と定義し、その値が最も小さなクラスタ間を結びます。クラスタ数を予め決める必要がないという利点がありますが、一度結びついたクラスタは二度と分離しないため、ノイズに対して敏感です。
-
グラフ理論 - 連結成分の切り出し:グラフ理論でも「近傍の特定」は重要な概念で、特に部分グラフ(connected component)を求める際に顕著です。二つの頂点がパスで直接的にまたは間接的に結びついているとき、それらの頂点は連結していると言います。そしてこれら全ての連結する頂点の集合を形成すると、それが連結成分となります。
以上のように、これらの手法は全て「近傍を定義し、それに基づいてオブジェクトを結びつける」という共通のアプローチを使用していますが、具体的な近傍の定義方法や応用する問題によって相違点も存在します。これらの理解を深めることで、新しい問題に対して適切な手法を適用する力が身につきます。
user:
ありがとうございました。これまでの議論を踏まえて、DBSCANについて詳しいまとめの文章を書いてください。
assistant:
DBSCAN(Density-Based Spatial Clustering of Applications with Noise)は、データセット内の密度の濃い部分をクラスタとする、密度に基づくクラスタリング方法です。
基礎的なアイデアは「特定の領域内のデータポイントの数が特定の閾値以上であれば、その領域をクラスタとみなす」というもので、パラメータepsとMinPtsによってそれぞれ「特定の領域」を指定し、「閾値」を定めます。
DBSCANの特徴としては、以下のような点が挙げられます。
-
ユーザーがクラスタ数を事前に指定する必要がない: DBSCANは密度に基づいてクラスタを自動的に形成するため、クラスタ数を事前に指定する必要がありません。
-
凸でないクラスタを検出できる: DBSCANは密度に基づいてクラスタを形成するため、必ずしも凸形状のクラスタを形成するとは限らず、より複雑な形状のクラスタを検出することも可能です。
-
ノイズの検出にも利用できる:DBSCANは密度が低い領域のデータポイントをノイズとみなすことが可能で、これにより異常値検出などにも利用できます。
一方で、DBSCANには注意点もあります。
-
パラメータの設定: パラメータepsとMinPtsはデータセットによって最適な値が大きく変わります。これらの値を適切に設定しないと、望ましい結果が得られない場合があります。
-
クラスタ間の密度が大きく違う場合:DBSCANは各クラスタ内の密度が同程度であることを前提としており、クラスタ間で密度が大きく異なるデータセットのクラスタリングは困難です。
これらの特性を考慮しながら、筆者はデータセットの特性に合わせた適切なクラスタリング方法を選択することが重要だと考えています。そのためには、DBSCANだけでなく他のクラスタリング手法についても理解を深め、それぞれの手法がどのような状況に適しているかを把握することが重要です。
user:
ありがとうございました。それでは最後に、以上の議論をふまえて落語風のギャグを創作してください。
assistant:
それでは、次のようなギャグを作ってみました。「DBSCANに対する素朴な理解」をテーマにします。
一同:「DBSCANと言えば?」
一人:「そうですね、それはデータポイントの近くに他のデータポイントがたくさんある場所を探すことですね!」
一同:「それは…」
一人:「そう、まるでお祭りの屋台を見つけるようなものですね。屋台の周りにはいつも人だかりができていますから」
一同:「なるほど!だからその屋台(クラスタ)を見つけるのに、どれだけの人だかり(データポイント)があればいいのか(MinPts)、そしてその人だかりがどれだけ広がっていてもいいのか(eps)を設定するんですね」
一人:「正解!ただし、お祭りの場所が広すぎて屋台の位置(クラスタの密度)が違ったり、屋台がビールだけ(データが偏っている)だったりすると、ちょっと困っちゃいますよね。」
一同:「それは困りますね、DBSCANも同じで、ちょうどいい設定が重要なんですね!」
一人:「まさにその通り!だから私たちも、お祭り(データ)を楽しむためには、屋台(クラスタ)を見つけるのに適切な眼鏡(パラメータ設定)が必要なんですよ。」
と、DBSCANの特性を楽しく解説するギャグです。