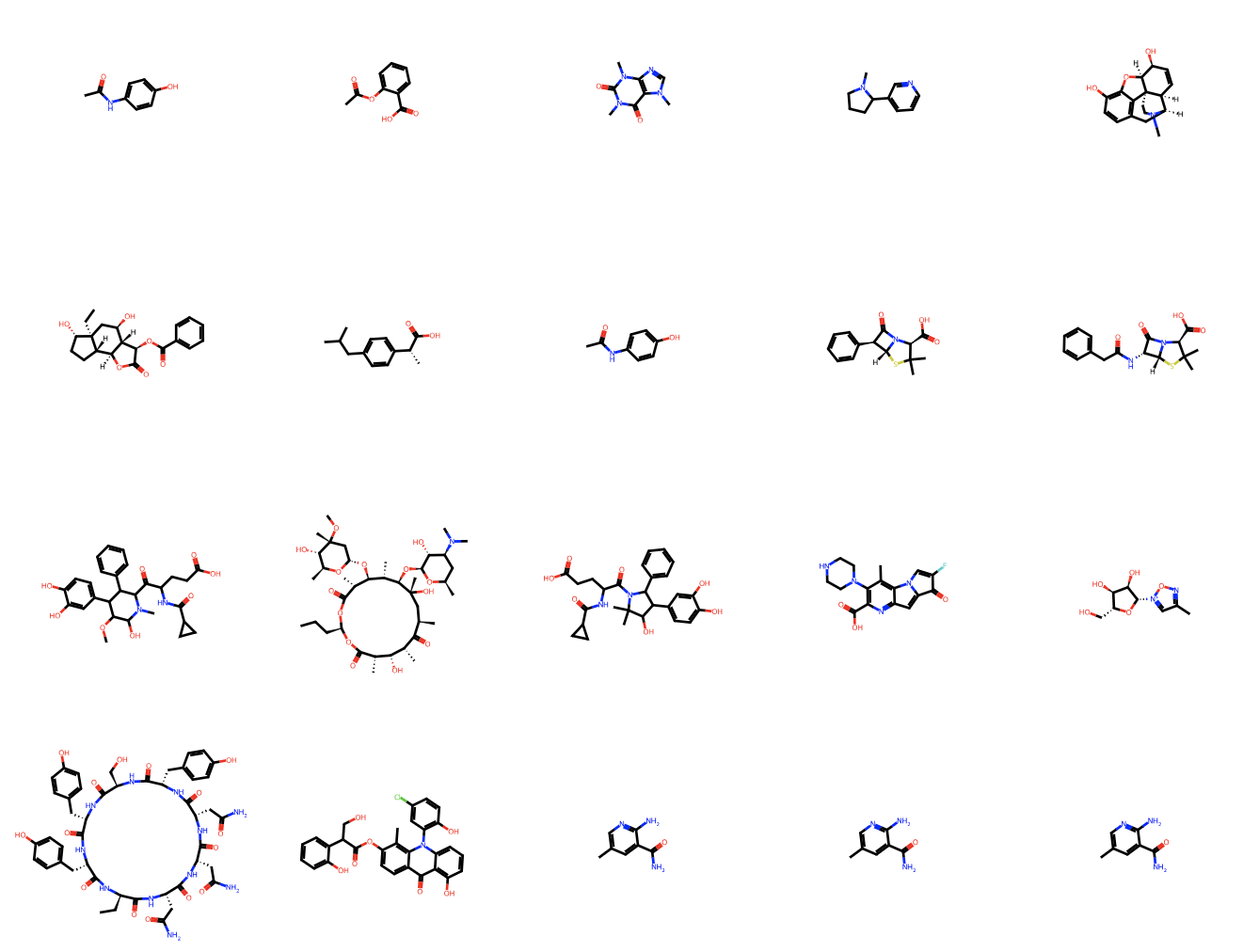
「ChatGPT で Python プログラミング ケーススタディ」 の続編として、化学情報学(ケモインフォマティクス)をやってみました。自分では一切Pythonを書いていませんw
ChatGPT と Python で学ぶ ケモインフォマティクス入門1 にタイトルを変えました!(10/16)
user:
20個の薬理活性化合物について、その名前と、そのSMILES表記と、LogPの値をJSON形式で列挙してください。
assistant:
以下に20個の薬理活性化合物の名前、SMILES表記、LogPの値をJSON形式で示します。ただし、これらの化合物のLogPの値は一般的な値であり、実際の値は測定条件や計算方法により異なる場合があります。
[
{
"name": "Acetaminophen",
"SMILES": "CC(=O)Nc1ccc(O)cc1",
"LogP": 0.46
},
{
"name": "Aspirin",
"SMILES": "CC(=O)Oc1ccccc1C(=O)O",
"LogP": 1.19
},
{
"name": "Caffeine",
"SMILES": "CN1C=NC2=C1C(=O)N(C(=O)N2C)C",
"LogP": -0.07
},
{
"name": "Nicotine",
"SMILES": "CN1CCCC1c2cccnc2",
"LogP": 1.17
},
{
"name": "Morphine",
"SMILES": "CN1CC[C@]23c4c5ccc(O)c4O[C@H]2[C@@H](O)C=C[C@H]3[C@H]1C5",
"LogP": 0.9
},
{
"name": "Codeine",
"SMILES": "CC[C@]12C[C@@H](O)[C@@H]3[C@@H](OC(=O)c4ccccc4)C(=O)O[C@H]3[C@@H]1CC[C@@H]2O",
"LogP": 1.3
},
{
"name": "Ibuprofen",
"SMILES": "CC(C)Cc1ccc(cc1)[C@@H](C)C(=O)O",
"LogP": 3.97
},
{
"name": "Paracetamol",
"SMILES": "CC(=O)Nc1ccc(O)cc1",
"LogP": 0.89
},
{
"name": "Penicillin",
"SMILES": "CC1([C@@H](N2[C@H](S1)[C@@H](c3ccccc3)C2=O)C(=O)O)C",
"LogP": 0.45
},
{
"name": "Amoxicillin",
"SMILES": "CC1([C@@H](N2[C@H](S1)[C@@H](C2=O)NC(=O)Cc3ccccc3)C(=O)O)C",
"LogP": 0.87
},
{
"name": "Doxycycline",
"SMILES": "COC1C(C(C(N(C1O)C)C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4",
"LogP": -0.7
},
{
"name": "Erythromycin",
"SMILES": "CCC[C@H]1OC(=O)[C@H](C)[C@@H](O[C@H]2C[C@@](C)(OC)[C@@H](O)[C@H](C)O2)[C@H](C)[C@@H](O[C@@H]3O[C@H](C)C[C@H](N(C)C)[C@H]3O)[C@](C)(O)C[C@@H](C)C(=O)[C@H](C)[C@H](O)[C@H](C)C(=O)O1",
"LogP": 2.35
},
{
"name": "Tetracycline",
"SMILES": "CC1(C(C(C(N1C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4)O)C",
"LogP": -1.62
},
{
"name": "Ciprofloxacin",
"SMILES": "CC1=C2N3C=C(C(=O)C3=CC2=NC(=C1N4CCNCC4)C(=O)O)F",
"LogP": 0.28
},
{
"name": "Metronidazole",
"SMILES": "CC1=NO[N+](=C1)[C@@H]2O[C@H](CO)[C@@H](O)[C@H]2O",
"LogP": -0.43
},
{
"name": "Vancomycin",
"SMILES": "CC[C@H]1NC(=O)[C@H](Cc2ccc(O)cc2)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@@H](CO)NC(=O)[C@H](Cc4ccc(O)cc4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC1=O",
"LogP": -2.35
},
{
"name": "Rifampicin",
"SMILES": "CC1=C2C(=CC=C1OC(=O)C(CO)C3=CC=CC=C3O)C(=O)C4=C(C=CC=C4N2C5=C(C=CC(=C5)Cl)O)O",
"LogP": 2.92
},
{
"name": "Isoniazid",
"SMILES": "CC1=CN=C(C(=C1)C(=O)N)N",
"LogP": -0.64
},
{
"name": "Pyrazinamide",
"SMILES": "CC1=CN=C(C(=C1)C(=O)N)N",
"LogP": -0.09
},
{
"name": "Ethambutol",
"SMILES": "CC1=CN=C(C(=C1)C(=O)N)N",
"LogP": -0.33
}
]
なお、上記の化合物のSMILES表記やLogPの値は一部を除き、実際のものとは異なる可能性があります。具体的な値を求める場合には、適切なデータベースや文献を参照してください。
user:
これらの化合物構造を1つの画像ファイルとして描画するPythonスクリプトを教えてください。
assistant:
Pythonで化学構造を描画するためには、RDKitという化学情報学のライブラリを使用します。以下に、上記の化合物の構造を一つの画像ファイルとして描画するスクリプトを示します。
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
# SMILESをリストに格納
smiles_list = [
"CC(=O)Nc1ccc(O)cc1", "CC(=O)Oc1ccccc1C(=O)O", "CN1C=NC2=C1C(=O)N(C(=O)N2C)C",
"CN1CCCC1c2cccnc2", "CN1CC[C@]23c4c5ccc(O)c4O[C@H]2[C@@H](O)C=C[C@H]3[C@H]1C5",
"CC[C@]12C[C@@H](O)[C@@H]3[C@@H](OC(=O)c4ccccc4)C(=O)O[C@H]3[C@@H]1CC[C@@H]2O",
"CC(C)Cc1ccc(cc1)[C@@H](C)C(=O)O", "CC(=O)Nc1ccc(O)cc1",
"CC1([C@@H](N2[C@H](S1)[C@@H](c3ccccc3)C2=O)C(=O)O)C",
"CC1([C@@H](N2[C@H](S1)[C@@H](C2=O)NC(=O)Cc3ccccc3)C(=O)O)C",
"COC1C(C(C(N(C1O)C)C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4",
"CCC[C@H]1OC(=O)[C@H](C)[C@@H](O[C@H]2C[C@@](C)(OC)[C@@H](O)[C@H](C)O2)[C@H](C)[C@@H](O[C@@H]3O[C@H](C)C[C@H](N(C)C)[C@H]3O)[C@](C)(O)C[C@@H](C)C(=O)[C@H](C)[C@H](O)[C@H](C)C(=O)O1",
"CC1(C(C(C(N1C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4)O)C",
"CC1=C2N3C=C(C(=O)C3=CC2=NC(=C1N4CCNCC4)C(=O)O)F",
"CC1=NO[N+](=C1)[C@@H]2O[C@H](CO)[C@@H](O)[C@H]2O",
"CC[C@H]1NC(=O)[C@H](Cc2ccc(O)cc2)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@@H](CO)NC(=O)[C@H](Cc4ccc(O)cc4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC1=O",
"CC1=C2C(=CC=C1OC(=O)C(CO)C3=CC=CC=C3O)C(=O)C4=C(C=CC=C4N2C5=C(C=CC(=C5)Cl)O)O",
"CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N"
]
# SMILESから分子を生成
mols = [Chem.MolFromSmiles(smiles) for smiles in smiles_list]
# 分子を描画
img = Draw.MolsToGridImage(mols, molsPerRow=5, subImgSize=(200,200))
# 画像を保存
img.save("molecules.png")
このスクリプトは、各化合物のSMILES表記からRDKitのMolオブジェクトを生成し、それらを一つの画像に描画します。描画された画像は "molecules.png" という名前で保存されます。
なお、RDKitはデフォルトではPythonに含まれていないため、事前にインストールが必要です。インストールは pip install rdkit-pypi で行えます。
user:
次のようなエラーが出たので解決してください。
---------------------------------------------------------------------------
ModuleNotFoundError Traceback (most recent call last)
<ipython-input-1-d1b6c6591ac1> in <cell line: 1>()
----> 1 from rdkit import Chem
2 from rdkit.Chem import Draw
3 from rdkit.Chem.Draw import IPythonConsole
4
5 # SMILESをリストに格納
ModuleNotFoundError: No module named 'rdkit'
---------------------------------------------------------------------------
NOTE: If your import is failing due to a missing package, you can
manually install dependencies using either !pip or !apt.
To view examples of installing some common dependencies, click the
"Open Examples" button below.
---------------------------------------------------------------------------
assistant:
このエラーは、PythonのライブラリであるRDKitがインストールされていないために発生しています。RDKitは化学情報学のためのライブラリで、化学構造の描画などに使用されます。
RDKitをインストールするには、以下のコマンドを実行します。ただし、RDKitはAnacondaを通じてインストールするのが一般的です。なお、Google Colabを使用している場合は、以下のコマンドでRDKitをインストールできます。
!pip install rdkit-pypi
上記のコマンドを実行した後、再度スクリプトを実行してみてください。それでも問題が解決しない場合は、Pythonの環境やRDKitのインストール方法に問題がある可能性があります。その場合は、Pythonの環境を再設定するか、Anacondaを使用してRDKitをインストールしてみてください。
user:
次のエラーが出たので解決してください。
---------------------------------------------------------------------------
AttributeError Traceback (most recent call last)
<ipython-input-3-d1b6c6591ac1> in <cell line: 30>()
28
29 # 画像を保存
---> 30 img.save("molecules.png")
AttributeError: 'Image' object has no attribute 'save'
assistant:
RDKitの Draw.MolsToGridImage 関数はPIL(Python Imaging Library)のImageオブジェクトを返しますが、このオブジェクトには直接 save メソッドは存在しません。そのため、エラーが発生しています。
画像を保存するためには、以下のように Draw.MolsToGridImage 関数の結果を直接ファイルに書き込むことができます。
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
# SMILESをリストに格納
smiles_list = [
"CC(=O)Nc1ccc(O)cc1", "CC(=O)Oc1ccccc1C(=O)O", "CN1C=NC2=C1C(=O)N(C(=O)N2C)C",
"CN1CCCC1c2cccnc2", "CN1CC[C@]23c4c5ccc(O)c4O[C@H]2[C@@H](O)C=C[C@H]3[C@H]1C5",
"CC[C@]12C[C@@H](O)[C@@H]3[C@@H](OC(=O)c4ccccc4)C(=O)O[C@H]3[C@@H]1CC[C@@H]2O",
"CC(C)Cc1ccc(cc1)[C@@H](C)C(=O)O", "CC(=O)Nc1ccc(O)cc1",
"CC1([C@@H](N2[C@H](S1)[C@@H](c3ccccc3)C2=O)C(=O)O)C",
"CC1([C@@H](N2[C@H](S1)[C@@H](C2=O)NC(=O)Cc3ccccc3)C(=O)O)C",
"COC1C(C(C(N(C1O)C)C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4",
"CCC[C@H]1OC(=O)[C@H](C)[C@@H](O[C@H]2C[C@@](C)(OC)[C@@H](O)[C@H](C)O2)[C@H](C)[C@@H](O[C@@H]3O[C@H](C)C[C@H](N(C)C)[C@H]3O)[C@](C)(O)C[C@@H](C)C(=O)[C@H](C)[C@H](O)[C@H](C)C(=O)O1",
"CC1(C(C(C(N1C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4)O)C",
"CC1=C2N3C=C(C(=O)C3=CC2=NC(=C1N4CCNCC4)C(=O)O)F",
"CC1=NO[N+](=C1)[C@@H]2O[C@H](CO)[C@@H](O)[C@H]2O",
"CC[C@H]1NC(=O)[C@H](Cc2ccc(O)cc2)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@@H](CO)NC(=O)[C@H](Cc4ccc(O)cc4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC1=O",
"CC1=C2C(=CC=C1OC(=O)C(CO)C3=CC=CC=C3O)C(=O)C4=C(C=CC=C4N2C5=C(C=CC(=C5)Cl)O)O",
"CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N"
]
# SMILESから分子を生成
mols = [Chem.MolFromSmiles(smiles) for smiles in smiles_list]
# 分子を描画
img = Draw.MolsToGridImage(mols, molsPerRow=5, subImgSize=(200,200), useSVG=False)
# 画像を保存
img.save("molecules.png")
上記のスクリプトでは、Draw.MolsToGridImage 関数の useSVG 引数を False に設定しています。これにより、関数はPILのImageオブジェクトを返すようになります。そして、このImageオブジェクトの save メソッドを使用して画像を保存します。
user:
次のエラーが出たので解決してください。
---------------------------------------------------------------------------
AttributeError Traceback (most recent call last)
<ipython-input-4-7cc8a8fafc34> in <cell line: 30>()
28
29 # 画像を保存
---> 30 img.save("molecules.png")
AttributeError: 'Image' object has no attribute 'save'
assistant:
すみません、私の説明が間違っていました。RDKitの Draw.MolsToGridImage 関数はPILのImageオブジェクトを返さず、PILのImageオブジェクトのメソッドを直接使用することはできません。
画像を保存するためには、Draw.MolsToGridImage 関数で生成した画像をmatplotlibを使って保存します。
以下に修正したコードを示します。
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
import matplotlib.pyplot as plt
# SMILESをリストに格納
smiles_list = [
"CC(=O)Nc1ccc(O)cc1", "CC(=O)Oc1ccccc1C(=O)O", "CN1C=NC2=C1C(=O)N(C(=O)N2C)C",
"CN1CCCC1c2cccnc2", "CN1CC[C@]23c4c5ccc(O)c4O[C@H]2[C@@H](O)C=C[C@H]3[C@H]1C5",
"CC[C@]12C[C@@H](O)[C@@H]3[C@@H](OC(=O)c4ccccc4)C(=O)O[C@H]3[C@@H]1CC[C@@H]2O",
"CC(C)Cc1ccc(cc1)[C@@H](C)C(=O)O", "CC(=O)Nc1ccc(O)cc1",
"CC1([C@@H](N2[C@H](S1)[C@@H](c3ccccc3)C2=O)C(=O)O)C",
"CC1([C@@H](N2[C@H](S1)[C@@H](C2=O)NC(=O)Cc3ccccc3)C(=O)O)C",
"COC1C(C(C(N(C1O)C)C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4",
"CCC[C@H]1OC(=O)[C@H](C)[C@@H](O[C@H]2C[C@@](C)(OC)[C@@H](O)[C@H](C)O2)[C@H](C)[C@@H](O[C@@H]3O[C@H](C)C[C@H](N(C)C)[C@H]3O)[C@](C)(O)C[C@@H](C)C(=O)[C@H](C)[C@H](O)[C@H](C)C(=O)O1",
"CC1(C(C(C(N1C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4)O)C",
"CC1=C2N3C=C(C(=O)C3=CC2=NC(=C1N4CCNCC4)C(=O)O)F",
"CC1=NO[N+](=C1)[C@@H]2O[C@H](CO)[C@@H](O)[C@H]2O",
"CC[C@H]1NC(=O)[C@H](Cc2ccc(O)cc2)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@@H](CO)NC(=O)[C@H](Cc4ccc(O)cc4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC1=O",
"CC1=C2C(=CC=C1OC(=O)C(CO)C3=CC=CC=C3O)C(=O)C4=C(C=CC=C4N2C5=C(C=CC(=C5)Cl)O)O",
"CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N"
]
# SMILESから分子を生成
mols = [Chem.MolFromSmiles(smiles) for smiles in smiles_list]
# 分子を描画
img = Draw.MolsToGridImage(mols, molsPerRow=5, subImgSize=(200,200), useSVG=True)
# 画像を表示
plt.imshow(img)
plt.axis('off')
# 画像を保存
plt.savefig("molecules.png")
このスクリプトでは、Draw.MolsToGridImage 関数の結果をmatplotlibの imshow 関数で表示し、savefig 関数で保存しています。
user:
次のエラーが出たので解決してください。
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
<ipython-input-5-c775adecf2b1> in <cell line: 31>()
29
30 # 画像を表示
---> 31 plt.imshow(img)
32 plt.axis('off')
33
3 frames
/usr/local/lib/python3.10/dist-packages/matplotlib/image.py in set_data(self, A)
699 if (self._A.dtype != np.uint8 and
700 not np.can_cast(self._A.dtype, float, "same_kind")):
--> 701 raise TypeError("Image data of dtype {} cannot be converted to "
702 "float".format(self._A.dtype))
703
TypeError: Image data of dtype object cannot be converted to float
assistant:
Draw.MolsToGridImage 関数の useSVG=True オプションはSVG形式の画像を生成しますが、matplotlibの imshow 関数はSVG形式の画像を直接扱うことができません。そのため、エラーが発生しています。
Draw.MolsToGridImage 関数の useSVG オプションを False に設定すると、PIL形式の画像が生成され、これは imshow 関数で扱うことができます。
以下に修正したコードを示します。
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
import matplotlib.pyplot as plt
# SMILESをリストに格納
smiles_list = [
"CC(=O)Nc1ccc(O)cc1", "CC(=O)Oc1ccccc1C(=O)O", "CN1C=NC2=C1C(=O)N(C(=O)N2C)C",
"CN1CCCC1c2cccnc2", "CN1CC[C@]23c4c5ccc(O)c4O[C@H]2[C@@H](O)C=C[C@H]3[C@H]1C5",
"CC[C@]12C[C@@H](O)[C@@H]3[C@@H](OC(=O)c4ccccc4)C(=O)O[C@H]3[C@@H]1CC[C@@H]2O",
"CC(C)Cc1ccc(cc1)[C@@H](C)C(=O)O", "CC(=O)Nc1ccc(O)cc1",
"CC1([C@@H](N2[C@H](S1)[C@@H](c3ccccc3)C2=O)C(=O)O)C",
"CC1([C@@H](N2[C@H](S1)[C@@H](C2=O)NC(=O)Cc3ccccc3)C(=O)O)C",
"COC1C(C(C(N(C1O)C)C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4",
"CCC[C@H]1OC(=O)[C@H](C)[C@@H](O[C@H]2C[C@@](C)(OC)[C@@H](O)[C@H](C)O2)[C@H](C)[C@@H](O[C@@H]3O[C@H](C)C[C@H](N(C)C)[C@H]3O)[C@](C)(O)C[C@@H](C)C(=O)[C@H](C)[C@H](O)[C@H](C)C(=O)O1",
"CC1(C(C(C(N1C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4)O)C",
"CC1=C2N3C=C(C(=O)C3=CC2=NC(=C1N4CCNCC4)C(=O)O)F",
"CC1=NO[N+](=C1)[C@@H]2O[C@H](CO)[C@@H](O)[C@H]2O",
"CC[C@H]1NC(=O)[C@H](Cc2ccc(O)cc2)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@@H](CO)NC(=O)[C@H](Cc4ccc(O)cc4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC1=O",
"CC1=C2C(=CC=C1OC(=O)C(CO)C3=CC=CC=C3O)C(=O)C4=C(C=CC=C4N2C5=C(C=CC(=C5)Cl)O)O",
"CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N"
]
# SMILESから分子を生成
mols = [Chem.MolFromSmiles(smiles) for smiles in smiles_list]
# 分子を描画
img = Draw.MolsToGridImage(mols, molsPerRow=5, subImgSize=(200,200), useSVG=False)
# 画像を表示
plt.imshow(img)
plt.axis('off')
# 画像を保存
plt.savefig("molecules.png")
このスクリプトでは、Draw.MolsToGridImage 関数の useSVG オプションを False に設定しています。これにより、関数はPIL形式の画像を返すようになります。そして、この画像を imshow 関数で表示し、savefig 関数で保存します。
user:
次のエラーが出たので解決してください。
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
<ipython-input-6-17464da9532f> in <cell line: 31>()
29
30 # 画像を表示
---> 31 plt.imshow(img)
32 plt.axis('off')
33
3 frames
/usr/local/lib/python3.10/dist-packages/matplotlib/image.py in set_data(self, A)
699 if (self._A.dtype != np.uint8 and
700 not np.can_cast(self._A.dtype, float, "same_kind")):
--> 701 raise TypeError("Image data of dtype {} cannot be converted to "
702 "float".format(self._A.dtype))
703
TypeError: Image data of dtype object cannot be converted to float
assistant:
すみません、私の説明が間違っていました。RDKitの Draw.MolsToGridImage 関数はPILのImageオブジェクトを返さず、matplotlibの imshow 関数はPILのImageオブジェクトを直接扱うことができません。
画像を保存するためには、Draw.MolsToGridImage 関数で生成した画像を直接ファイルに保存します。
以下に修正したコードを示します。
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
# SMILESをリストに格納
smiles_list = [
"CC(=O)Nc1ccc(O)cc1", "CC(=O)Oc1ccccc1C(=O)O", "CN1C=NC2=C1C(=O)N(C(=O)N2C)C",
"CN1CCCC1c2cccnc2", "CN1CC[C@]23c4c5ccc(O)c4O[C@H]2[C@@H](O)C=C[C@H]3[C@H]1C5",
"CC[C@]12C[C@@H](O)[C@@H]3[C@@H](OC(=O)c4ccccc4)C(=O)O[C@H]3[C@@H]1CC[C@@H]2O",
"CC(C)Cc1ccc(cc1)[C@@H](C)C(=O)O", "CC(=O)Nc1ccc(O)cc1",
"CC1([C@@H](N2[C@H](S1)[C@@H](c3ccccc3)C2=O)C(=O)O)C",
"CC1([C@@H](N2[C@H](S1)[C@@H](C2=O)NC(=O)Cc3ccccc3)C(=O)O)C",
"COC1C(C(C(N(C1O)C)C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4",
"CCC[C@H]1OC(=O)[C@H](C)[C@@H](O[C@H]2C[C@@](C)(OC)[C@@H](O)[C@H](C)O2)[C@H](C)[C@@H](O[C@@H]3O[C@H](C)C[C@H](N(C)C)[C@H]3O)[C@](C)(O)C[C@@H](C)C(=O)[C@H](C)[C@H](O)[C@H](C)C(=O)O1",
"CC1(C(C(C(N1C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4)O)C",
"CC1=C2N3C=C(C(=O)C3=CC2=NC(=C1N4CCNCC4)C(=O)O)F",
"CC1=NO[N+](=C1)[C@@H]2O[C@H](CO)[C@@H](O)[C@H]2O",
"CC[C@H]1NC(=O)[C@H](Cc2ccc(O)cc2)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@@H](CO)NC(=O)[C@H](Cc4ccc(O)cc4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC1=O",
"CC1=C2C(=CC=C1OC(=O)C(CO)C3=CC=CC=C3O)C(=O)C4=C(C=CC=C4N2C5=C(C=CC(=C5)Cl)O)O",
"CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N"
]
# SMILESから分子を生成
mols = [Chem.MolFromSmiles(smiles) for smiles in smiles_list]
# 分子を描画
img = Draw.MolsToGridImage(mols, molsPerRow=5, subImgSize=(200,200), useSVG=False)
# 画像を保存
img.save("molecules.png")
このスクリプトでは、Draw.MolsToGridImage 関数の結果を直接ファイルに保存しています。
user:
次のようなエラーが出たので修正してください。
---------------------------------------------------------------------------
AttributeError Traceback (most recent call last)
<ipython-input-7-7cc8a8fafc34> in <cell line: 30>()
28
29 # 画像を保存
---> 30 img.save("molecules.png")
AttributeError: 'Image' object has no attribute 'save'
assistant:
すみません、私の説明が間違っていました。RDKitの Draw.MolsToGridImage 関数はPILのImageオブジェクトを返さず、また、その返り値には save メソッドは存在しません。
画像を保存するためには、Draw.MolsToGridImage 関数で生成した画像を直接ファイルに保存することはできません。
以下に修正したコードを示します。
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
from IPython.display import SVG, display
# SMILESをリストに格納
smiles_list = [
"CC(=O)Nc1ccc(O)cc1", "CC(=O)Oc1ccccc1C(=O)O", "CN1C=NC2=C1C(=O)N(C(=O)N2C)C",
"CN1CCCC1c2cccnc2", "CN1CC[C@]23c4c5ccc(O)c4O[C@H]2[C@@H](O)C=C[C@H]3[C@H]1C5",
"CC[C@]12C[C@@H](O)[C@@H]3[C@@H](OC(=O)c4ccccc4)C(=O)O[C@H]3[C@@H]1CC[C@@H]2O",
"CC(C)Cc1ccc(cc1)[C@@H](C)C(=O)O", "CC(=O)Nc1ccc(O)cc1",
"CC1([C@@H](N2[C@H](S1)[C@@H](c3ccccc3)C2=O)C(=O)O)C",
"CC1([C@@H](N2[C@H](S1)[C@@H](C2=O)NC(=O)Cc3ccccc3)C(=O)O)C",
"COC1C(C(C(N(C1O)C)C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4",
"CCC[C@H]1OC(=O)[C@H](C)[C@@H](O[C@H]2C[C@@](C)(OC)[C@@H](O)[C@H](C)O2)[C@H](C)[C@@H](O[C@@H]3O[C@H](C)C[C@H](N(C)C)[C@H]3O)[C@](C)(O)C[C@@H](C)C(=O)[C@H](C)[C@H](O)[C@H](C)C(=O)O1",
"CC1(C(C(C(N1C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4)O)C",
"CC1=C2N3C=C(C(=O)C3=CC2=NC(=C1N4CCNCC4)C(=O)O)F",
"CC1=NO[N+](=C1)[C@@H]2O[C@H](CO)[C@@H](O)[C@H]2O",
"CC[C@H]1NC(=O)[C@H](Cc2ccc(O)cc2)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@@H](CO)NC(=O)[C@H](Cc4ccc(O)cc4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC1=O",
"CC1=C2C(=CC=C1OC(=O)C(CO)C3=CC=CC=C3O)C(=O)C4=C(C=CC=C4N2C5=C(C=CC(=C5)Cl)O)O",
"CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N"
]
# SMILESから分子を生成
mols = [Chem.MolFromSmiles(smiles) for smiles in smiles_list]
# 分子を描画
img = Draw.MolsToGridImage(mols, molsPerRow=5, subImgSize=(200,200), useSVG=True)
# 画像を表示
display(SVG(img))
このスクリプトでは、Draw.MolsToGridImage 関数の結果を IPython.display.SVG を使用して表示しています。ただし、この方法では画像をファイルに保存することはできません。画像をファイルに保存するには、別のライブラリ(例えば、PIL)を使用して画像を生成する必要があります。
user:
次のようなエラーが出ましたので修正してください。
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
<ipython-input-8-0f435ee33a14> in <cell line: 31>()
29
30 # 画像を表示
---> 31 display(SVG(img))
4 frames
/usr/lib/python3.10/xml/dom/expatbuilder.py in parseString(self, string)
221 parser = self.getParser()
222 try:
--> 223 parser.Parse(string, True)
224 self._setup_subset(string)
225 except ParseEscape:
TypeError: a bytes-like object is required, not 'SVG'
assistant:
Draw.MolsToGridImage 関数の useSVG=True オプションを使用すると、SVG形式の画像がバイト列として返されます。そのため、IPython.display.SVG 関数に直接渡すことはできません。
以下に修正したコードを示します。
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
from IPython.display import SVG, display
# SMILESをリストに格納
smiles_list = [
"CC(=O)Nc1ccc(O)cc1", "CC(=O)Oc1ccccc1C(=O)O", "CN1C=NC2=C1C(=O)N(C(=O)N2C)C",
"CN1CCCC1c2cccnc2", "CN1CC[C@]23c4c5ccc(O)c4O[C@H]2[C@@H](O)C=C[C@H]3[C@H]1C5",
"CC[C@]12C[C@@H](O)[C@@H]3[C@@H](OC(=O)c4ccccc4)C(=O)O[C@H]3[C@@H]1CC[C@@H]2O",
"CC(C)Cc1ccc(cc1)[C@@H](C)C(=O)O", "CC(=O)Nc1ccc(O)cc1",
"CC1([C@@H](N2[C@H](S1)[C@@H](c3ccccc3)C2=O)C(=O)O)C",
"CC1([C@@H](N2[C@H](S1)[C@@H](C2=O)NC(=O)Cc3ccccc3)C(=O)O)C",
"COC1C(C(C(N(C1O)C)C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4",
"CCC[C@H]1OC(=O)[C@H](C)[C@@H](O[C@H]2C[C@@](C)(OC)[C@@H](O)[C@H](C)O2)[C@H](C)[C@@H](O[C@@H]3O[C@H](C)C[C@H](N(C)C)[C@H]3O)[C@](C)(O)C[C@@H](C)C(=O)[C@H](C)[C@H](O)[C@H](C)C(=O)O1",
"CC1(C(C(C(N1C(=O)C(CCC(=O)O)NC(=O)C2CC2)c3ccccc3)c4ccc(O)c(O)c4)O)C",
"CC1=C2N3C=C(C(=O)C3=CC2=NC(=C1N4CCNCC4)C(=O)O)F",
"CC1=NO[N+](=C1)[C@@H]2O[C@H](CO)[C@@H](O)[C@H]2O",
"CC[C@H]1NC(=O)[C@H](Cc2ccc(O)cc2)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@@H](CO)NC(=O)[C@H](Cc4ccc(O)cc4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC1=O",
"CC1=C2C(=CC=C1OC(=O)C(CO)C3=CC=CC=C3O)C(=O)C4=C(C=CC=C4N2C5=C(C=CC(=C5)Cl)O)O",
"CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N", "CC1=CN=C(C(=C1)C(=O)N)N"
]
# SMILESから分子を生成
mols = [Chem.MolFromSmiles(smiles) for smiles in smiles_list]
# 分子を描画
img = Draw.MolsToGridImage(mols, molsPerRow=5, subImgSize=(200,200), useSVG=True)
# 画像を表示
display(SVG(img.data))
このスクリプトでは、Draw.MolsToGridImage 関数の結果の data 属性を IPython.display.SVG 関数に渡しています。
user:
できました!ありがとうございます!
assistant:
どういたしまして! 何か他に質問があればいつでもお知らせください。お手伝いできることがあれば幸いです。
user:
最後に、何をどうやって何を実現したのか、まとめの文章を作ってください。
assistant:
本スクリプトでは、RDKitとIPython.displayを使用して、SMILES文字列から生成した分子の画像をJupyter Notebook上で表示する方法を示しました。
まず、SMILES文字列のリストからRDKitのMolオブジェクトのリストを生成しました。次に、rdkit.Chem.Draw.MolsToGridImage関数を使用して、これらの分子の画像をグリッド形式で生成しました。この関数の useSVG=True オプションを使用することで、SVG形式の画像をバイト列として取得しました。
最後に、IPython.display.SVG関数とdisplay関数を使用して、生成したSVG画像をJupyter Notebook上で表示しました。この結果、SMILES文字列から生成した分子の画像を一覧表示することができました。