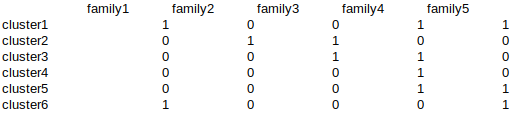はじめに
共起ネットワークとは、文章中で単語同士が共起する(文章中にともに存在する)関係を表した図である。
テキストマイニングにおいて主に用いられる手法だが、これを
文章 → バクテリアのゲノム
単語 → 糖質関連酵素
に置き換えて行っている論文1を見つけ興味を持った。
この論文では、糖質関連酵素データベース(CAZy databse)で公開されているテーブルを入力データとしており、ざっくり結果は、似た活性を持っているファミリーをクラスタリングできました、という事だった。
しかし、糖質関連酵素は非常に多様に分化しており、同じファミリーに分類される酵素であっても活性が異なる場合が多いし、ゲノム単位を一つのまとまりにしているのも大雑把すぎるのではないかと感じた。
論文の手法を改良する
細菌のゲノム上では、オペロンのように、協働して働く遺伝子がクラスターを作っていることが多い。
機能未知な標的遺伝子と同じクラスターに存在している既知遺伝子を調べることで、機能未知遺伝子の機能推定に役立てることができる。
(『バクテリアの CAZyme 遺伝子クラスターを網羅的に解析したい』)
そのため、
文章 → 遺伝子クラスター
単語 → 糖質関連酵素(ファミリー、サブファミリー単位)
とすれば、より高い解像度で似た機能を持つ酵素をクラスタリングできるのではないだろうか。
データベース上には、基質が分からないため、酵素活性の評価ができていない機能未知酵素が多く存在している。共起ネットワーク解析によって、既知酵素と機能未知タンパク質との関連性を可視化できれば、その基質の推定ができ、ひいては新規酵素の発見にも繋がるのではないかと考えた。
データの準備
配列データ
糖質関連酵素(CAZyme)は、その種類が多様化しており、非常に多くのファミリーに分類されている。
そのように細分化、複雑化した分類に対応するためのツールとして、CAZyme ファミリーのアノテーションに特化した「dbCAN」というプログラムがある。
dbCAN はアミノ酸配列を入力としてファミリーのアノテーションを行うだけでなく、ゲノムの配列データを入力としてCAZymeを含む遺伝子クラスターの予測を行う機能もついている。
また、dbCAN を開発しているチームが、DNA データをdbCANで処理した結果を dbCAN-seq2 というデータベースとして公開している。
dbCAN-seqからはまとめて配列データをダウンロードでき、今回の目的にぴったりなので、これを用いることにした。
Download タブから、COW RUMEN、 HUMAN GUT、 HUMAN ORAL、 MARINE の4つのデータセットがダウンロードできる。
それぞれの場所でサンプリングされたシークエンスの結果を使っているらしい。
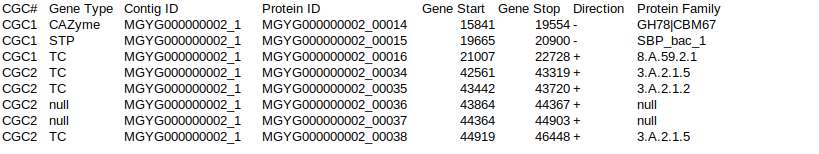
遺伝子クラスターのデータはこのようになっていた。
サブファミリー分類
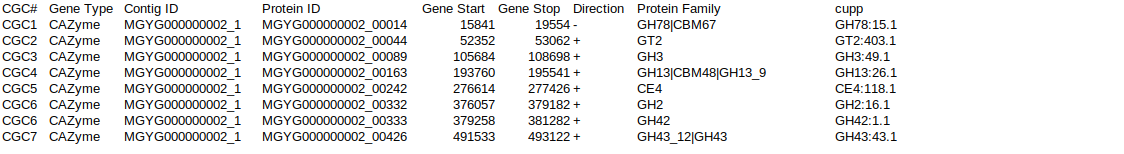
糖質関連酵素は、同じファミリー内でも多様化が進み、同じファミリーの酵素であっても基質特異性が異なることが多い。そのため、ファミリーを基質特異性が同じグループ単位まで分けられるような下位分類が欲しい。これには、CUPP プログラムによるサブファミリー分類を用いた。
糖質関連酵素以外の不要な行を除き、CUPPのサブファミリーの列を加えて、以下のような表が準備できた。
コサイン類似度
タンパク質(単語に相当)同士の関連性の高さの指標として、コサイン類似度を用いた。
下の表のように、クラスター(文に相当)とタンパク質ファミリーの総当り表を作って、各クラスター中に出現しないものは0、出現するものを1とカウントしていく。
こうすることで、各タンパク質ファミリーを(0,1,0,0,0,0,1,…)のようなベクトルとして扱え、ベクトル同士のコサイン類似度(cosθ)を以下の式で計算できる。
cosθ = \frac{\vec{x}*\vec{y}}{|\vec{x}||\vec{y}|}
遺伝子クラスターはよく行われている自然言語の共起ネット解析とは入力データが異なり、どう扱えばいいのか分からなかったので、共起ネットワークを作るライブラリを使わず、地道に pandas でデータを変形していった。
この部分は結構大変だったが、長くなるので割愛……。
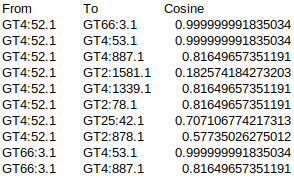
総当たり表を作って、各ファミリー同士のコサイン類似度を計算し、最終的にこのようなテーブルができた。
このグラフは無向グラフだが、便宜的に列ラベルを<From>、<To> としている。
ネットワークの可視化
ネットワークの可視化には Cytoscape を使った。
csv ファイルをドロップして開き、
Layout > Prefuse Force Directed Layout > Cosine を選択した。
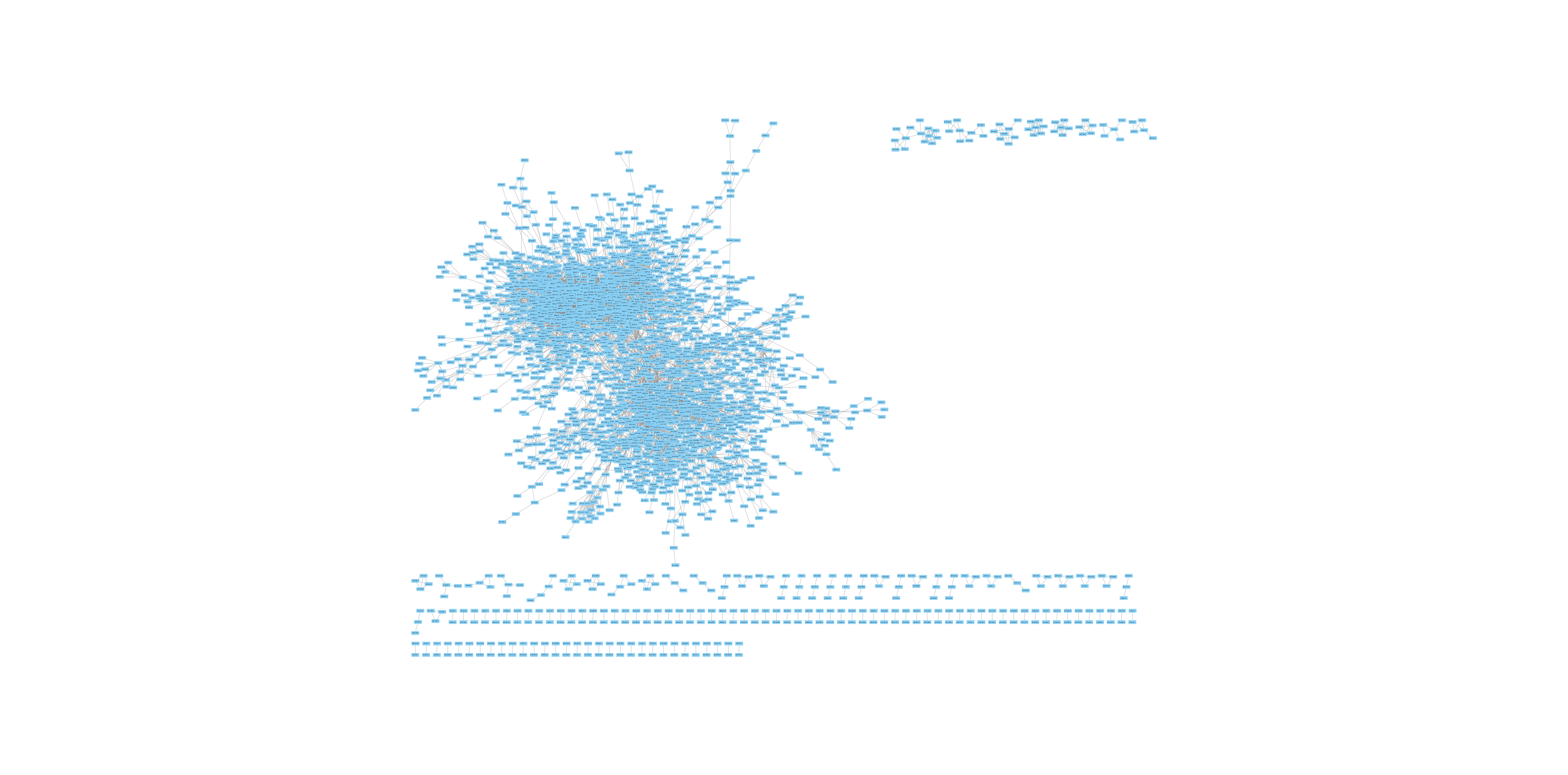
ちゃんとネットワーク図になっている!
が、ほとんどのノードがくっついてしまったので、コサイン類似度に閾値を設けてデータを減らすことにした。
いい感じにばらけた気がする。
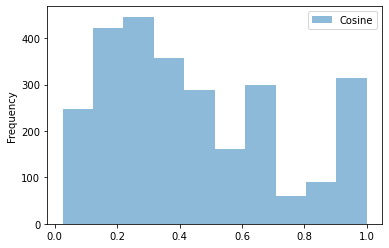
コサイン類似度 > 0.5 とすると、固まっていた部分がさらにほぐれた一方、ノードの数がかなり減った。
色々な閾値で出力しておいて、見たいものに合わせてグラフを選ぶのがよさそう。
結果の解釈
上に示したのは Marine のデータなので、海藻に含まれるアガロースの分解酵素(アガラーゼ)を見てみる。アガラーゼは複数のファミリーにおいて報告がある(収斂進化?)。ここでは、分かりやすいところで、これまでに β-アガラーゼのみが報告されている GH50 ファミリーに着目した。
ターゲットの検索
最終産物の csv ファイルから GH50 を含むサブファミリーを検索し、ID のリストを .txt ファイルにした。
GH50.txt
GH50:1.1
GH50:4.1
GH50:3.1
GH50:2.1
Select > Nodes > From ID List File
でこのファイルを読み込むと、リストにあるノードが選択されて黄色くなるので、その部分を拡大して見ていく。
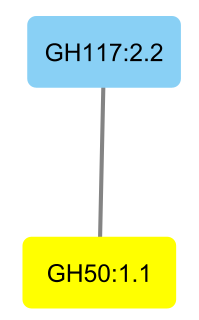
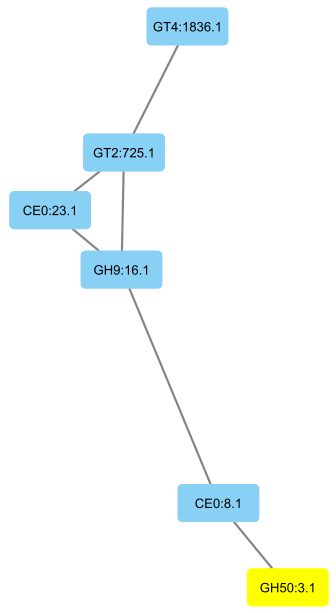
GH50 の β-アガラーゼとアガロオリゴ糖分解酵素の GH117 が繋がっている!
おそらく、アガロース(多糖)を GH50 の β-アガラーゼが分解して、生じたオリゴ糖をアガロオリゴ糖分解酵素がさらに単糖まで分解しているのだろう。
一方で、これまでに明らかになっている情報からは説明が付けられない共起関係もあった。
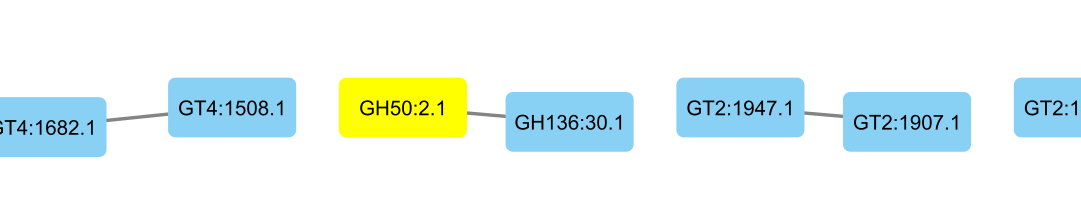 (コサイン類似度 > 0.3)
(コサイン類似度 > 0.3)
上図で GH50:2.1 と繋がっている GH136 ファミリーからは、これまでにラクト-N-ビオシダーゼという、ミルクオリゴ糖を分解する酵素の報告しかない。 元データの偏りによって偶然生じたものなのではないのか、などは検討の余地があるが、GH136:30.1 は機能解析済みの酵素が存在しない CUPP サブファミリーなので、こいつは新規な活性を持つ酵素の可能性がある……かもしれない。
さらに、ファミリー未分類の推定カルボキシエステラーゼ(加水分解ではなく、脱離反応によって糖質を分解する酵素)とGH50 の共起も見られた。
CE0:8.1 はアガロースかそのオリゴ糖を分解するのかも……?
おわりに
-
バクテリアゲノムの共起ネットワーク図からは、まだいろいろ読み取れることがありそう。
-
Cytoscape にはいろいろな機能があるようなので、ノードの色分けなどによってグラフを見やすく改良できそう。
-
Geng A, Jin M, Li N, Zhu D, Xie R, Wang Q, Lin H, Sun J. New Insights into the Co-Occurrences of Glycoside Hydrolase Genes among Prokaryotic Genomes through Network Analysis. Microorganisms. 2021 Feb 19;9(2):427. doi: 10.3390/microorganisms9020427. PMID: 33669523; PMCID: PMC7922503.(オープンアクセス誌なので誰でも読めます) ↩
-
Jinfang Zheng and others, dbCAN-seq update: CAZyme gene clusters and substrates in microbiomes, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D557–D563, https://doi.org/10.1093/nar/gkac1068 ↩