RStudio v1.0から正式サポートされた機能の中に、profvisを使ったパフォーマンスプロファイリングがあります。
ここでは、このprofileに関してさっくり触れてみたいと思います。
何ができるの?
解析対象のソースに対して、行単位or呼び出される関数単位でメモリの消費と処理時間を計測することができます。
行単位の結果はFlame Graphタブ、関数の呼び出し毎の結果はDataタブで見ることができます。
※ちなみに、ほぼ10msec程度で処理が終わるようなものは、プロファイラが処理を拾えないので何もできないので、ご注意を。。。
実際にプロファイリングしてみましょう
ということで、実際にやりつつ詳しく追ってみましょう。
今回はテストで次のようなスクリプトをまずは走らせてみましょう。
この例は、Rで処理が遅くなるのでやっちゃダメな典型例としておなじみの
ベクター処理が可能なものをfor分で処理したケースです。
n <- 7
# パターン1
# Cなんかでやってると普通だけど、
# Rの言語特徴的にダメダメなやり方
x <- 1:(10^n)
for(i in 1:length(x)){
x[i] <- x[i] + 1
}
こんな感じで、ソースの行を選択してProfile Selected Linesを選択すると、プロファイリングが開始します。
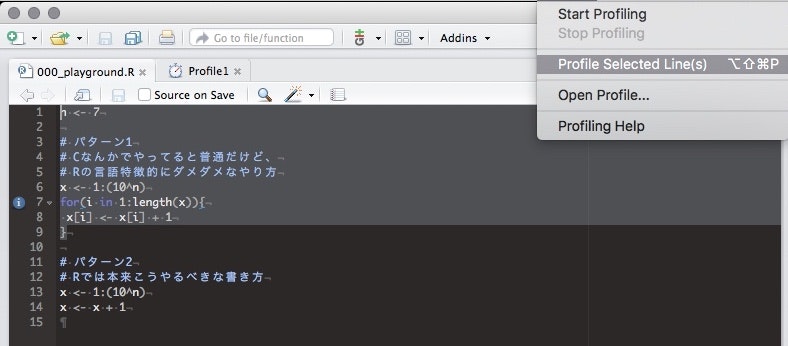
選択した行の処理が全て終わると次のように、プロファイリング結果表示画面が自動的に出てきます。
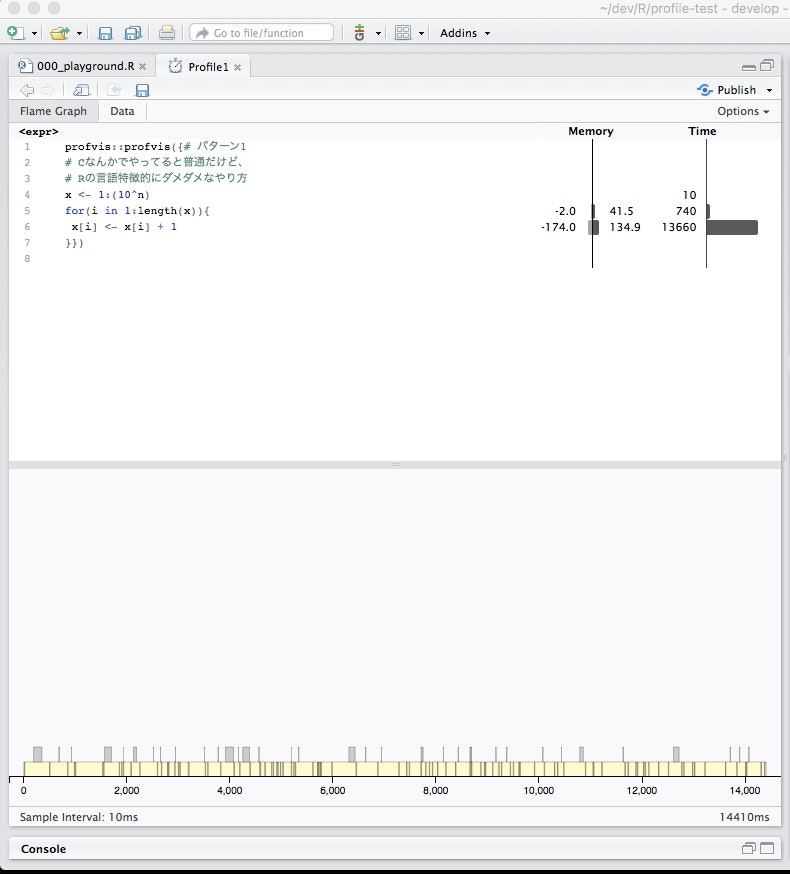
上のパネルではFlame GraphとDataという2つのタブがあります。
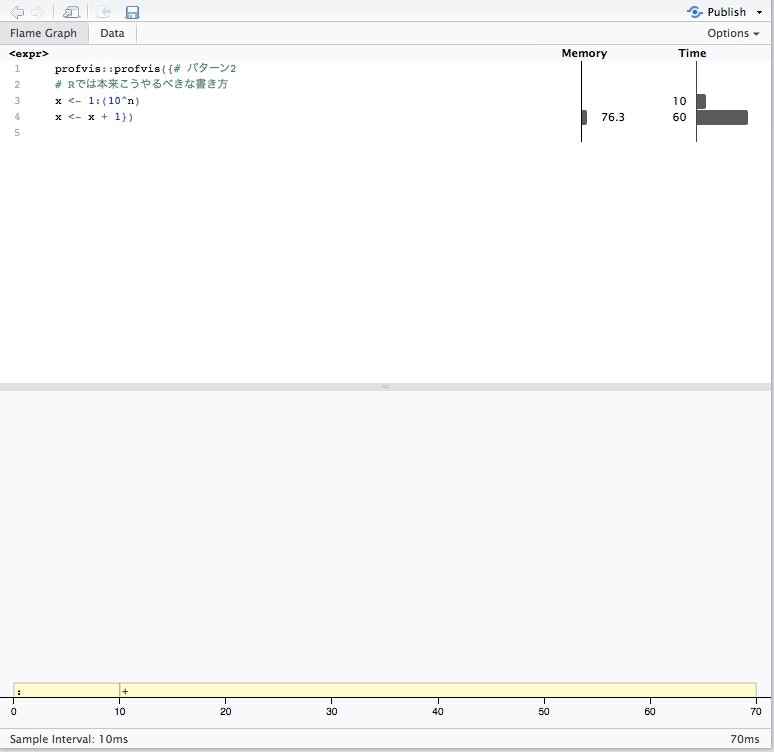
自動的に開かているはずのFrame Graphでは、行単位でのメモリ・処理時間の分析結果が表示されてますね。
下の部分には10ms単位でどんな処理が走っていたかが表示されます。
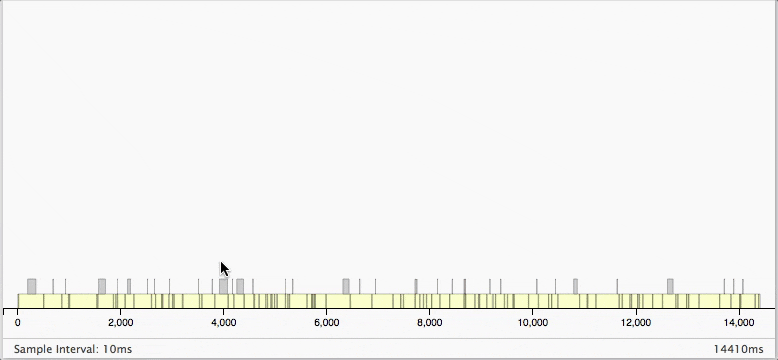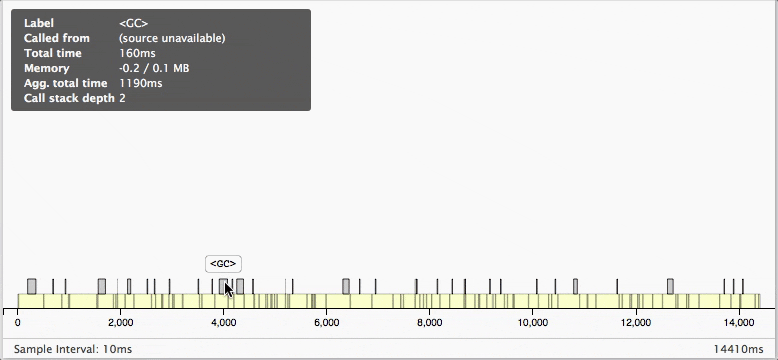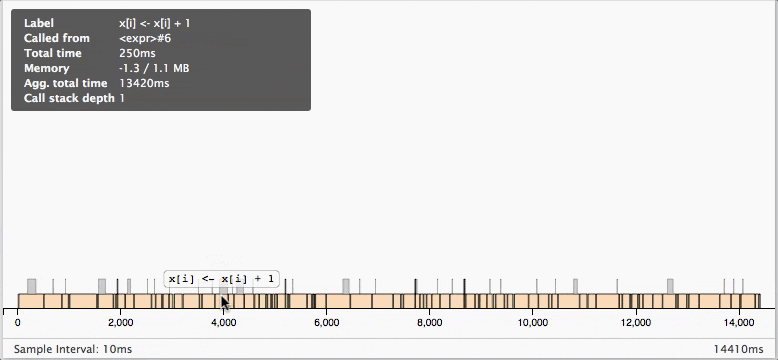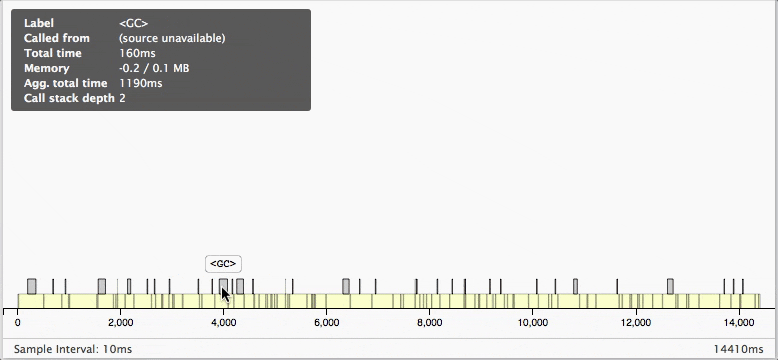
マウスオーバーすると、より詳細が見ることができます。
処理のバー部分をダブルクリックすると、その部分をズームして見ることができます。
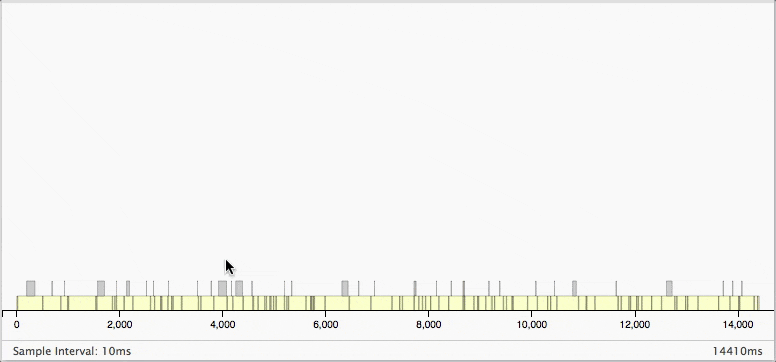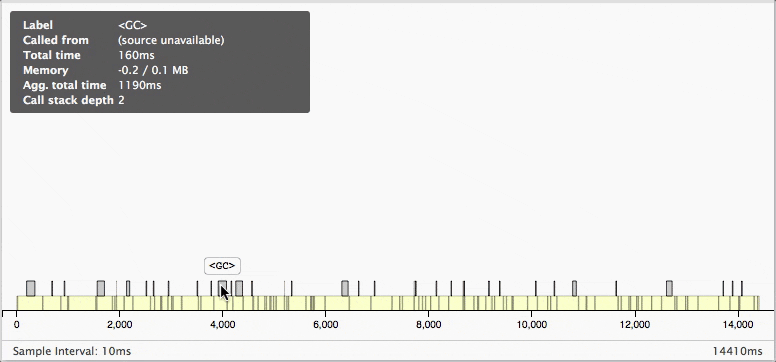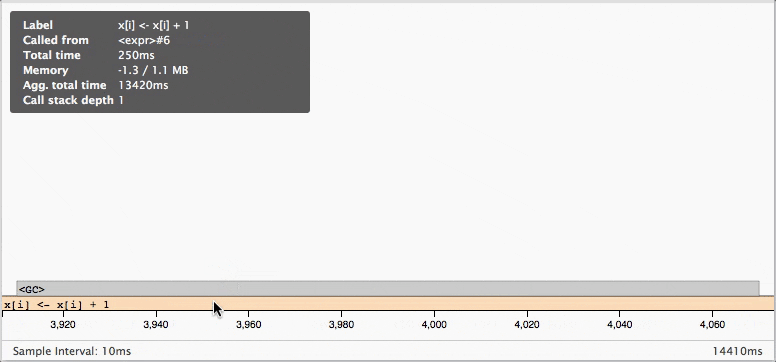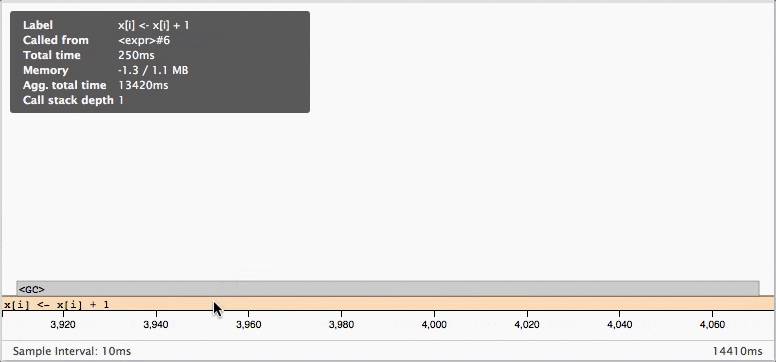
今回の例の実行結果を見ると、各配列の要素に加算をしている間で
ちょくちょくガーベジコレクションが働いていてパフォーマンスが低下しているのがわかりますね。
では、さきほどの処理と同じものをRらしくベクター処理として計算するとどうなるか、試してみましょう。
流すスクリプトは次の通りで、プロファイリングのやり方は一緒です。
# パターン2
# Rでは本来こうやるべきな書き方
x <- 1:(10^n)
x <- x + 1
パターン1と比べると処理時間が14410msから70msへ約200倍早くなってるのがわかりますね。
また、メモリ最大消費部分も倍近く違いがあるのがわかりますね。
また、処理の途中はガーベジコレクションが一切働いていないので、なぜforを使わない方が効率の良いのかがよくわかりますね。
さて、実際の処理では同じ関数をいろんなところで複数回呼び出すというケースが考えられますね。
そういう場合はDataタブを使う方が良いはずです。
Dataタブでは、どのコールで処理に時間が食っているか、関数の呼び出し関係から見る事が出来ます。
といっても、ピンと来ないと思うので次のようなテストスクリプトを流して結果を見てみましょう。
これは、パターン1とパターン2をわざと関数化して処理しているだけです。
add_me <- function(x1, x2){
return(x1+x2)
}
x <- 1:(10^n)
for(i in 1:length(x)){
x[i] <- add_me(x[i], 1)
}
x <- 1:(10^n)
x <- add_me(x, 1)
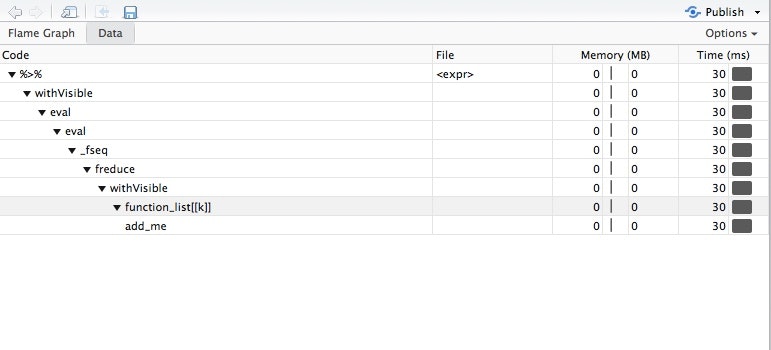
この実行結果をDataタブで見てみると。。。
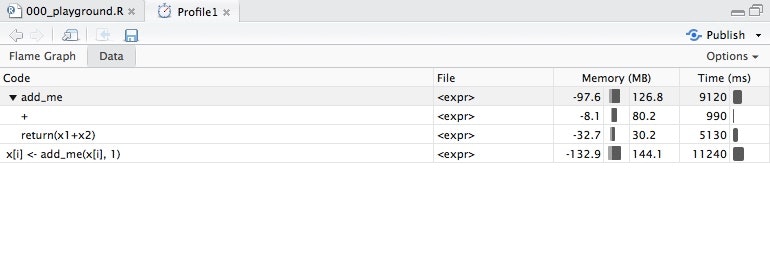
add_me関数の呼び出しと、その中でさらに評価されている関数たちのメモリ・CPU消費具合の合計が見えます。
ここでは、処理結果を変数に入れている部分が結局のところ高価な処理になってるみたいですね。
ここで、実験的にパイプを使ったコードを評価するとどうなるか?を試してみましょう。
テストスクリプトは先程のケースで使った関数と、magrittrを使って次の通りのを試してみましょう。
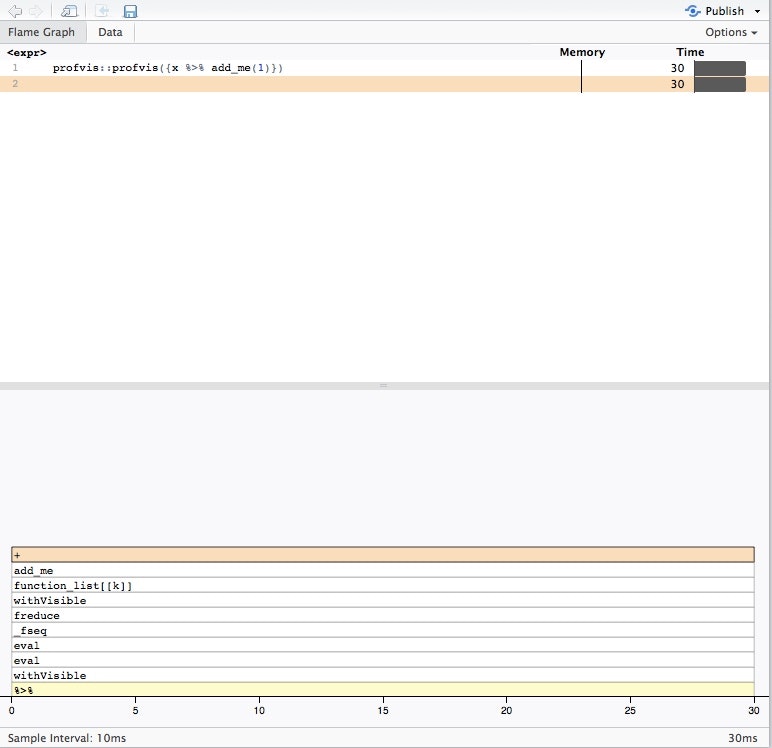
x %>% add_me(1)
おやおや、随分と複雑になってしまいましたね。。。
パイプ演算子の中身をズルズルっと評価して表示しているため、仕方ない状況なわけですね。
とは言え、関数の呼び出しが複雑なものの、メモリ消費や処理速度は今までの中で最も優れているのはわかりますね。
こんな感じで何だか処理が重いなーという時は、Profileを立ち上げて
コードの臭い部分をささっとあぶり出して、より効率の良い分析作業に繋げていきましょう!!
ENJOY!