できるようになること
brm関数で推定したデータをもとに、tidybayes関数を使うことで、以下のような事後分布の図が作成できるようになります。
やり方
パッケージの読み込み
library(brms)
library(tidyverse)
library(tidybayes)
brmsfit型のデータを作成
とりあえず何かしらのデータが欲しいので、Rにあらかじめ入っているアヤメのデータセットを使用します。でたらめな仮定で以下のモデルを作成しました。
従属変数にがくの広さ、独立変数にがくの長さを指定しました。また、切片の値やがくの長さの係数の値はアヤメの種類ごとに異なると仮定し、変量効果を設定しています。
dat <- iris
results <- brm(Petal.Width ~ Sepal.Length + (1 + Sepal.Length|Species),
family = gaussian(link = "identity"),
data = dat,
seed = 123)
データを抽出
tidybayesというとても便利なパッケージを使用します。今回は、パッケージの解説が目的ではないので、詳しい解説は省略します。詳しい解説が欲しい方は以下の二つが特におすすめです。
変数の名前の確認
以下の手順では、任意のデータの事後確率を抽出します。その際に、変数の名前を指定するので、確認しましょう。
get_variables(results)
事後確率を抽出
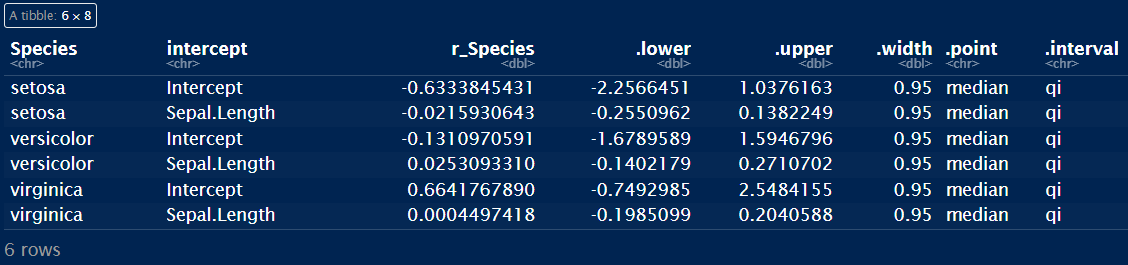
事後確率を抽出してみると、Speciesの列にアヤメの種類、interceptの列に切片とがくの長さの係数が入っています。今回はがくの長さの係数の事後確率をアヤメの種類ごとに描画したいと思います。従って、Sepal.Lengthのデータのみをfilter関数で取り出します。ちなみに、 median_qi関数で算出されたr_Speciesの列には中央値が表示されていますが、平均値などを表示してくれる関数もあります。
results %>%
spread_draws(r_Species[Species, "intercept"]) %>%
median_qi()
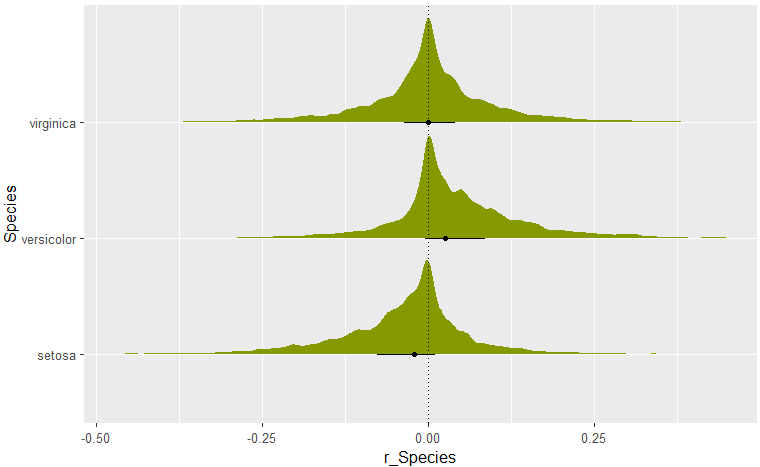
事後分布を描画
results %>%
# アヤメの種類ごとのモデルの推定値の事後確率を抽出
spread_draws(r_Species[Species, "intercept"]) %>%
# がくの長さの事後確率のみを指定
filter(intercept == "Sepal.Length") %>%
# Plotの指定
ggplot(aes(x = r_Species, y = Species)) +
stat_halfeye(.width = .5, size = 2/3, fill = "#859900") +
# 0をまたいでいるか見やすいように、垂直な線を描画
geom_vline(xintercept = 0, color = "black", linetype = 3)
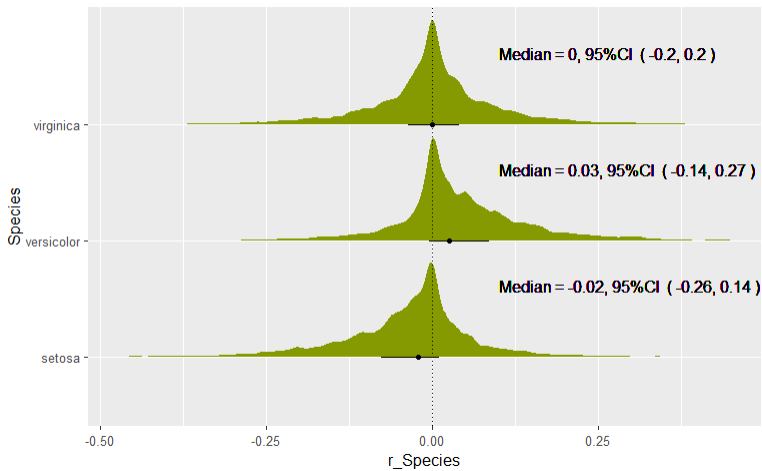
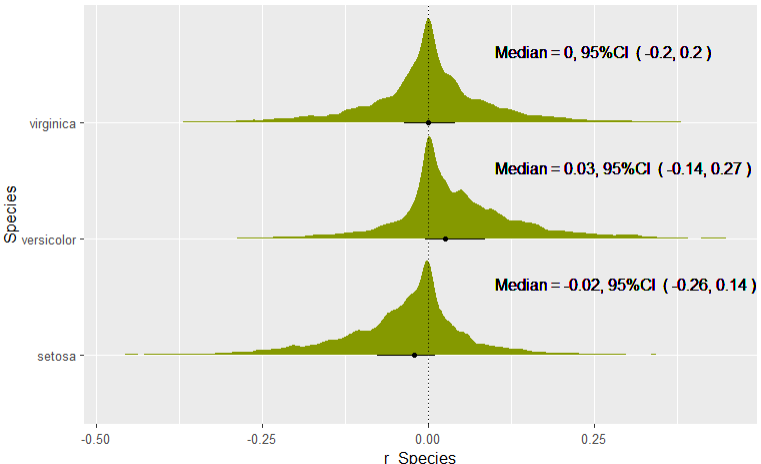
95%信用区間を図の中に書き入れる
ちなみに、メタ分析を行う場合のパッケージでは、信用(信頼)区間を描画するものもあるみたいです(参考記事:Doing Meta-Analysis in R: A Hands-on Guide, 6 Forest Plots )。
95%信用区間を算出し、データフレームで保存
まず、median_qi関数で、中央値、95%信用区間の上限、下限の値を計算し、それをデータフレームとして保存します。次に、データフレーム内の列名を変更しておきます。この後の手順で、このデータを上で描画した際に使用したデータと結合させるためです。
text_plot <- results %>%
spread_draws(r_Species[Species, "intercept"]) %>%
median_qi() %>%
filter(intercept == "Sepal.Length") %>%
select(Species, r_Species, .lower, .upper)
colnames(text_plot) <- c("Species", "CI_r_Species",
"CI_lower", "CI_upper")
データフレームの結合 + 描画
left_join関数で二つのデータを結合させます。ggplot2で作画したオブジェクトに文字を追加する場合、geom_text関数を使用します(参考記事:How to add OR and 95% CI as text into a forest plot?)。この際、手入力で信用区間を入力するのであれば、一つ上のデータフレームの作成はいりません。しかし、それではミスにつながるので、お勧めしません。そのため、label = 引数の中で、上で作成したデータフレーム(text_plot)内に格納した信用区間を変数として指定します。
results %>%
spread_draws(r_Species[Species, "intercept"]) %>%
filter(intercept == "Sepal.Length") %>%
# ここで、あらかじめ計算していた信頼区間のデータと結合させる
left_join(text_plot, by = "Species") %>%
# 作図
ggplot(aes(x = r_Species, y = Species)) +
stat_halfeye(.width = .5, size = 2/3, fill = "#859900") +
# 0をまたいでいるか見やすいように垂直な線を引く
geom_vline(xintercept = 0, color = "black", linetype = 3) +
# 信用区間を図に書き入れる
geom_text(aes(
# y軸(縦)の指定です。アヤメの種類を指定することで、各事後分布の横に表示されます。
y = Species,
# x軸(横)の指定です。
x = median(r_Species) + 0.1,
# ここで好きな文字を指定
label = paste0("Median = ",round(CI_r_Species, 2), ", 95%CI ", " ( ", round(CI_lower, 2),
", ", round(CI_upper, 2), " )")),
hjust = 0,
vjust = -5, # 重ならないように縦にずらす
position = position_dodge(width = 0.5), color = "black"
# 太字にもできます↓
#, fontface = "bold"
)
最後に
画質が影響しているのか、表示されている文字が少し粗い気がします。フォントを変えればいいのでしょうか...